|
ISSN 1666-7948 www.quimicaviva.qb.fcen.uba.ar |
Una
nueva visión del ARN: los ARN de interferencia. ¿Un nuevo genoma?
por
Juan Carlos Calvo
Dr. en Ciencias Químicas, Facultad de Ciencias Exactas y Naturales, Universidad de Buenos Aires. Profesor Adjunto del Departamento de Química Biológica, FCEyN, UBA. Investigador Independiente, CONICET. Director del Departamento de Química Biológica, FCEyN, UBA
Recibido 28 de octubre de 2003/ Aceptado 20 de noviembre de 2003
Introducción.
Cuando la noticia acerca de la decodificación del genoma humano fue tapa de periódicos y revistas, tanto científicas como de divulgación, parecía que todo estaba terminado en lo que a la información genética se refería. Sin embargo, todavía quedaba mucho más por dilucidar, y esto también salió a la superficie gracias al Proyecto Genoma Humano.
Con apenas 30.000 – 40.000 genes no se podía explicar la inmensa variedad, que supera ese número, de proteínas celulares. Tampoco lo podía explicar, tal vez porque aún no se lo conoce en su totalidad, el proceso de “corte y empalme” alternativo que sufre el ácido ribonucleico mensajero (ARNm) en el núcleo de las células eucarióticas, antes de poder ser utilizado por los ribosomas para la síntesis proteica.
Resultaba evidente que la información genética debía ser más compleja y, por lo tanto, lo que faltaba en la explicación debería ser buscado en otra parte.
Y dónde si no en el “ADN basura”, esa porción mayoritaria de ácido desoxirribonucleico para la que todavía no se había encontrado una función específica, más que la de asegurar la identidad genética de cada persona, tan de moda en estos días a través de los juicios criminales que son tapa de tantas revistas no científicas.
Y, como el que busca encuentra, se encontró ese “genoma oculto”, esa otra capa de información que promete ser, tal vez, incluso más importante que la conocida, porque podría ser la que controle la expresión de los genes “clásicos”.
La actividad científica se ha caracterizado siempre por su constante evolución, donde dogmas que parecían inalterables, como debiera ser todo dogma, debieron abrir paso a nuevos descubrimientos que los hacían menos rígidos. Tal el caso muy famoso de la oveja Dolly, quien rompió con el dogma de la imposibilidad de “desprogramar” una célula totalmente diferenciada.
Cada vez surge con más fuerza, esa frase de Ortega y Gasset que nos describe moldeados por nuestro “yo y las circunstancias”, sobre todo en el campo de la genómica, donde a los aspectos puramente genéticos se les suman los “epigenéticos” que, desde otro entorno, regulan y controlan su expresión. El ambiente influye, ciertamente, en la expresión de nuestros genes y, por lo tanto, es necesario mantener ese ambiente lo más puro y limpio que se pueda. Pero esto puede ser tema para otro artículo.
Hablemos un poco de esta otra capa de información, que llevará tal vez más tiempo para decodificar de lo que llevó el Proyecto Genoma Humano, porque se cuenta con menos elementos a partir de los cuales comenzar la investigación.
El dogma del flujo de información, desde su comienzo en los años 50, es relativamente simple: el ADN fabrica ARN, el ARN fabrica las proteínas y éstas son las que trabajan dentro y fuera de la célula. La información para este flujo se encuentra encerrada en la enroscada doble hélice de ADN. Un gen es una secuencia particular de cuatro bases químicas (A, T, C y G) encontrada en una de las dos cadenas, que contiene la información necesaria para sintetizar una proteína. Estas cuatro bases químicamente definidas se aparean en forma específica y universal, para mantener unidas las dos cadenas del ADN.
Con el mayor conocimiento de la actividad genética, se amplió la definición de gen a la codificación de otras moléculas no proteicas, como ARNs que cumplen funciones tan variadas como estructurales o enzimáticas (ribozimas).
En los organismos superiores, tales como los humanos, estos genes aparecen fragmentados en trozos de secuencias codificantes, separadas por otras secuencias, a veces más extensas, que no codifican para proteínas. Esto se tomó como un indicio de complejidad, dado que las bacterias no presentan esta característica. Es un hecho que la porción de ADN con información para proteínas representa menos del 2% del total en los cromosomas humanos. Es decir que los 3.000.000.000 o más de pares de bases que todos llevamos en nuestras células, deben estar allí por alguna otra razón.
Por este motivo ya no se habla de genes, sino que se prefiere hablar de “unidades transcripcionales”, dado que no todos los genes serán traducidos a proteínas, sino que su producto quedará como moléculas de ARN de diversas características.
Cuando se comparan porciones de ADN entre distintas especies, se llega a la conclusión que más de 1.000 segmentos aparecen con cambios mínimos entre las mismas, indicando que estas secuencias podrían contribuir a la adecuación evolutiva de las mismas. Solamente unas 200 se encuentran dentro de las porciones de ADN codificantes para proteínas, dos tercios se encuentran en intrones (secuencias que separan a los exones, que son las porciones que integrarán al ARNm) y el resto se encuentra disperso en ese “ADN basura” que aparece entre los genes.
El
ARN como actor principal en la nueva comedia genética.
Los investigadores han comenzado a prestar más atención a las moléculas de ARN y les encuentran cada vez más funciones.
La interacción vuelve a ser el centro de la escena: proteína-proteína, ARN-ARN, ARN-ADN, ARN-proteína, ARN-compuestos químicos pequeños. Los científicos han trasladado términos utilizados en otro contexto, al estudio de estas moléculas y, así, consideran a las proteínas como moléculas “analógicas”, mientras que el ARN sería una molécula “digital”, con secuencias específicas. Una molécula de ARN podría “flotar” hasta encontrarse con una molécula complementaria de ADN o ARN y, entonces, interactuar formando una cadena doble. Una proteína interactúa con otra molécula, por complementariedad de estructura, más que de secuencia, en forma bastante parecida a como una llave encaja en la cerradura correspondiente.
Genes
no convencionales.
Los genes, según la sabiduría “popular” son aquellas secciones de ADN que codifican para proteínas funcionales. Como ya dijimos, estas secuencias constituyen cerca del 2% del genoma humano. Sin embargo, el resto del genoma está lleno de ADN que es “no codificante” pero no por ello, “inservible”. Cada vez aparecen más “genes no codificantes” que, sin embargo, dan origen a ARNs activos, incluyendo variantes que pueden silenciar o regular a los genes “convencionales”.
Por ejemplo: si se transcribiese la hebra “codificante” de ADN, además de la que sirve como “molde” o “templado” en un gen, se sintetizaría una cadena de ARN que resultaría complementaria o “antisentido” del ARNm correspondiente. Estos ARNs “antisentido” podrían interceptar al ARNm transcripto a partir del gen correspondiente y, de este modo, impedir que aquel se traduzca en una proteína.
Los genes codificantes para proteínas, como dijimos, poseen secciones no codificantes llamadas intrones. Estas secuencias son eliminadas del transcripto inicial de ARN. Luego, las porciones codificantes se unen para generar el ARNm maduro. Aunque algunos intrones se degradan, otros poseen elementos activos que se convierten en “ARN de inteferencia” (iARN), microARN que pueden controlar otros genes. Un intrón no degradado puede ser tomado por la maquinaria celular generadora de estos iARN y ser utilizado para bloquear ciertos ARNm en forma específica.
Otra forma de ARN también descubierta, son los conocidos como “ribo-interruptores” (“riboswitches”) que actúan como interruptores genéticos de precisión. En muchos casos son producidos por porciones de ADN no codificantes que se encuentran entre genes. Estos ribo-interruptores adoptan una conformación compleja, de manera tal que una parte de la molécula puede unirse a un blanco proteico o químico específico y la otra parte poseer el código en ARN para un producto proteico. De esta manera, el ribo-interruptor se “enciende” y produce la proteína en cuestión, solamente si se encuentra presente su blanco. Estas moléculas de ARN, en su parte no codificante, son receptores muy sensibles para un blanco químico en particular. Una colisión con esta molécula activa al interruptor, causando un cambio conformacional en la otra punta de la molécula, que contiene un mapa clásico para el diseño de una proteína, tal como lo hace un gen clásico, pero que solamente se pone en funcionamiento cuando está presente la primera molécula en cuestión. Estos interruptores han sido encontrados en especies de todos los reinos vivientes. Tal vez representen el ancestro común más antiguo.
Un ejemplo de la importancia de ciertos tipos particulares de ARN, lo tenemos en la enfermedad conocida como “hipoplasia del cartílago piloso” (“CHH, cartilage hair hypoplasia”), una enfermedad recesiva identificada por primera vez entre la población Amish. Uno de cada 19 lleva una copia defectuosa del gen, que causa un tipo inusual de enanismo, en el que las personas no solamente son de estatura corta, sino que presentan alta propensión a cáncer y desórdenes inmunológicos. En el 2001 se identificó al culpable de esta enfermedad, un gen codificante solamente para ARN, llamado RMRP. El ARN transcripto por este gen es capaz de interactuar con proteínas para formar una enzima que altera la función mitocondrial. Un cambio en solamente una base de este ARN, puede significar la diferencia entre una vida sana, con estatura normal, o una vida breve (si es que se recibe la misma mutación de ambos padres) y con estatura corta.
Más
acerca de los iARN.
El área del silenciamiento específico de genes ha sido motivo de múltiples estudios, no solamente por su biología interesante sino también por su potencial como herramienta experimental.
El fenómeno del ARN doble cadena como inductor del silenciamiento genómico se conoce desde hace varios años en plantas, como una forma de co-supresión (referido al silenciamiento de lugares, o loci, endógenos, siguiendo la introducción de transgenes. También se refiere al silenciamiento de lugares transgénicos de una manera que depende del número de copias) y también como una forma de silenciamiento genómico inducido por virus. Cuando, años más tarde, se descubrió un mecanismo similar en animales, se pensó en la utilización de ARN doble cadena como una herramienta muy poderosa en el campo del silenciamiento de genes.
Primero se descubrió que el ARN doble cadena era un silenciador más potente que cualquiera de las cadenas simples (ARN “sentido” o “antisentido”). A este fenómeno se lo llamó “interferencia por ARN” (iARN) para distinguirlo mecanísticamente de la supresión clásica mediada por ARN antisentido.
Uno de los primeros misterios en ser resuelto fue cómo el ARN doble cadena (dsARN), disparador del silenciamiento, proveía de especificidad a la maquinaria silenciadora. Un paso clave se dio al descubrir la formación de pequeños ARNs que resultaron complementarios al blanco a silenciar. El descubrimiento de estos ARNs pequeños llevó a los estudios bioquímicos de la actividad que los generaba.
Una familia de ribonucleasas, RNasa III, reconoce en forma específica ARN doble cadena, y fue la primera candidata para la actividad que generaba estos pequeños ARNs de interferencia (siARNs). En Drosophila se encontró una clase de enzimas RNasa III que era capaz de generar siARNs, a partir de dsARNs largos, en un proceso dependiente de ATP. Estas enzimas están conservadas evolutivamente en organismos competentes para iARN, y, en muchos casos, se ha demostrado una actividad bioquímica característica.
Para ejemplificar esto de manera más simple, podemos ver la figura que sigue:
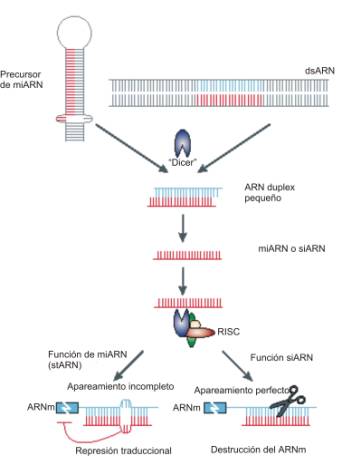
El sistema de iARN (interferencia por ARN) es un mecanismo de defensa, aparentemente muy antiguo, contra ARN doble cadena extraños. ARNs de solamente 22 nucleótidos en longitud, llamados ARN de interferencia pequeños (siARNs), son cortados a partir de dsARN más largos por una enzima llamada “dicer”. La hebra antisentido del siARN es utilizada por un complejo silenciador inducido por ARN (RISC) para guiar el clivaje de ARNm, promoviendo de esta forma la degradación del ARNm. Las abreviaturas utilizadas son: miARN, microARN, stARN, ARN pequeño temporario.
El proceso central que involucra al iARN es el corte de dsARN en pequeños trozos de una longitud definida por la enzima “dicer”. Esta enzima corta al dsARN en dos clases de ARNs pequeños: microARNs (miARNs) y ARNs pequeños de interferencia (iARNs), con una longitud aproximada de 21-23 nucleótidos. Aunque los microARNs también detienen la producción de proteínas, se piensa que los siARNs son los principales protagonistas en la interferencia por ARN. La enzima “dicer” entrega estos siARNs a un grupo de proteínas llamado el complejo silenciador inducido por ARN (RISC), que utiliza la cadena antisentido del siARN para unirse al ARNm y degradarlo, resultando en el silenciamiento del gen. El sistema de interferencia por ARN es extremadamente eficiente, porque RISC es una enzima y cataliza múltiples vueltas de interferencia, tal vez cientos o miles in vivo.
Dado que la interferencia por ARN confía en la interacción específica entre secuencias del siARN y del ARNm, los siARNs pueden ser diseñados para silenciar casi a cualquier gen. La relativa facilidad con que los genes pueden ser silenciados mediante esta técnica, ha causado una revolución en la Biología Molecular. Utilizando los datos provenientes de la secuenciación de diversos genomas, el silenciamiento de genes por ARN puede ser utilizado en una escala alarmante.
Las posibilidades terapéuticas de esta técnica son enormes. Tomar la secuencia de ADN de un gen y diseñar dsARN que puedan silenciar en forma específica y efectiva un gen relacionado con una enfermedad, es análogo a la producción de anticuerpos monoclonales. El problema que se presenta es la posibilidad de estimular la respuesta de interferón, ante la presencia de dsARNs de 30 nucleótidos o más de longitud, en células de mamíferos. Pero esto ya ha sido solucionado por transfección directa del siARN.
Todavía quedan muchos misterios del sistema de interferencia por ARN que deben ser resueltos, pero una cosa es segura, y es que estos pequeños iARN han revolucionado la forma en que los científicos piensan acerca del ARN y cómo el ADN, el ARN y las proteínas son controlados y funcionan. El futuro dirá hacia dónde y qué utilidad se les dará dentro de las compañías farmacéuticas o la investigación científica.
Bibliografía
consultada y sugerida:
-The unseen genome: gems among the junk. W. Wayt Gibas en Scientific American, 28-33. November 2003.
-Non-coding
RNA genes and the modern RNA world. Sean R. Eddy en Nature Reviews Genetics,
Vol. 2, 919-929, December 2001.
-An
expanding universe of noncoding RNAs. Gisela
Storz en Science, Vol. 296, 1260-1263, May 17, 2002.
-Challenging
the dogma : the hidden layer of non-protein-coding RNAs in complex
organisms. John S. Mattick en BioEssays, Vol. 25, 930-939, October 2003
-RNAi:
an ever-growing puzzle. Ahmet M. Denli and Gregory J. Hannon en TIBS, Vol. 28,
196-201, April 2003.
-RNA
interference: the next genetics revolution? Safia Wasi
en Understanding the
RNAissance: http://www.nature.com/horizon/rna/background.html