De átomos y sus orbitales a moléculas y estructuras biológicas, en realidad aumentada adaptada a teléfonos, tablets y computadoras sin instalar programas ni hardware especial
Fabio Cortés Rodríguez1, Celeste Aguirre-Pranzoni2, Analía Príncipe3 y Luciano A. Abriata1,4
1Laboratory for Biomolecular Modeling, École Polytechnique Fédérale de Lausanne and Swiss Institute of Bioinformatics, Suiza. 2Área de Química Orgánica, Departamento de Química, Facultad de Química Bioquímica y Farmacia, Universidad Nacional de San Luis, Argentina. 3Departamento de Ciencias Naturales, Universidad Nacional de Río Cuarto, Argentina. 4Protein Production and Structure Core Facility, École Polytechnique Fédérale de Lausanne, Suiza
Recibido: 15/09/2021 - Aceptado: 23/11/2021
Resumen
Introducimos a la comunidad de habla hispana una actualización del sitio MoleculARweb (https://molecularweb.epfl.ch) y las nuevas herramientas webXR de Google, que juntos permiten abordar estructuras y mecanismos químicos y biológicos en realidad aumentada accesible sin instalar programas ni hardware especiales, directo en navegadores web de teléfonos, tablets y computadoras, en forma gratuita y abierta. Con MoleculARweb, estudiantes y educadores pueden manipular representaciones virtuales de átomos, moléculas, orbitales, biomacromoléculas y estructuras biológicas, para enseñar y aprender sobre la estructura atómica, los orbitales atómicos y moleculares y la teoría TRPEV; la estereoquímica y las posibles distintas conformaciones de las moléculas; los ácidos, las bases, los equilibrios de protonación y los puentes de hidrógeno; la estructura de las proteínas, los ácidos nucleicos y sus interacciones; e incluso visualizar estructuras experimentales de ensamblados biológicos tan grandes como los virus. Por sobre esto, los modelos de realidad aumentada de organelas, células y tejidos disponibles en Google extienden la cobertura temática de MoleculARweb hacia la biología celular y de sistemas. Aquí explicamos brevemente cómo usar estas herramientas, ahora accesibles directamente desde la página principal de la revista Química Viva de la Universidad Nacional de Buenos Aires (http://www.quimicaviva.qb.fcen.uba.ar). Además, ejemplificamos cómo integrarlos en una experiencia completa que comienza en la relación entre estados de protonación y cargas, siguiendo con interacciones electrostáticas que estabilizan complejos ADN-proteína para luego visualizar como éstos se ensamblan en nucleosomas, los cromosomas dentro del núcleo celular dentro de una célula, luego el sistema circulatorio como ejemplo de nivel de tejidos, y finalmente volviendo a hacer zoom en una célula, una mitocondria, el grupo hemo de una proteína, y finalmente los orbitales 3d de un átomo.
Palabras clave:
Summary
We introduce the Spanish-speaking community to an update of MoleculARweb (https://molecularweb.epfl.ch, available in 6 languages including English and Spanish) and to the new WebXR resources from Google, that together enable the inspection of chemical and biological structures and mechanisms at various levels of resolution using augmented reality that runs on standard devices without special hardware and right inside web browsers without software installations, for free. Using MoleculARweb on smartphones, tablets, laptops or desktop computers, students and educators can manipulate virtual representations of atoms, molecules, their orbitals, and even complex biomolecules and biological structures, to teach and learn about molecular structure, atomic and molecular orbitals, and VSEPR theory; stereochemistry and conformations; acids, bases and hydrogen-bonding; the structures of proteins, nucleic acids and their complexes; and the structures of large biological structures like nucleosomes and viral particles. In turn, the commodity augmented reality models of subcellular organelles, cells and tissues available at Googleextend MoleculARweb’s focus on chemistry and molecular structure towards a more complete coverage of biology. Thus, together the two resources provide new pedagogic material of highly immersive nature, and easily available worldwide, spanning all the way from chemistry to cell biology. Here we explain how to use these resources, now linked to the home page of the Química Viva journal at the National University of Buenos Aires (http://www.quimicaviva.qb.fcen.uba.ar), and exemplify their integration into a full experience that spans from molecular protonation states and the resulting charges to protein-DNA interaction, their packing into nucleosomes, the chromosomes inside cells, and the human circulatory system, then zooming back out to cells, the mitochondrion, a heme group, and the 3d orbitals.
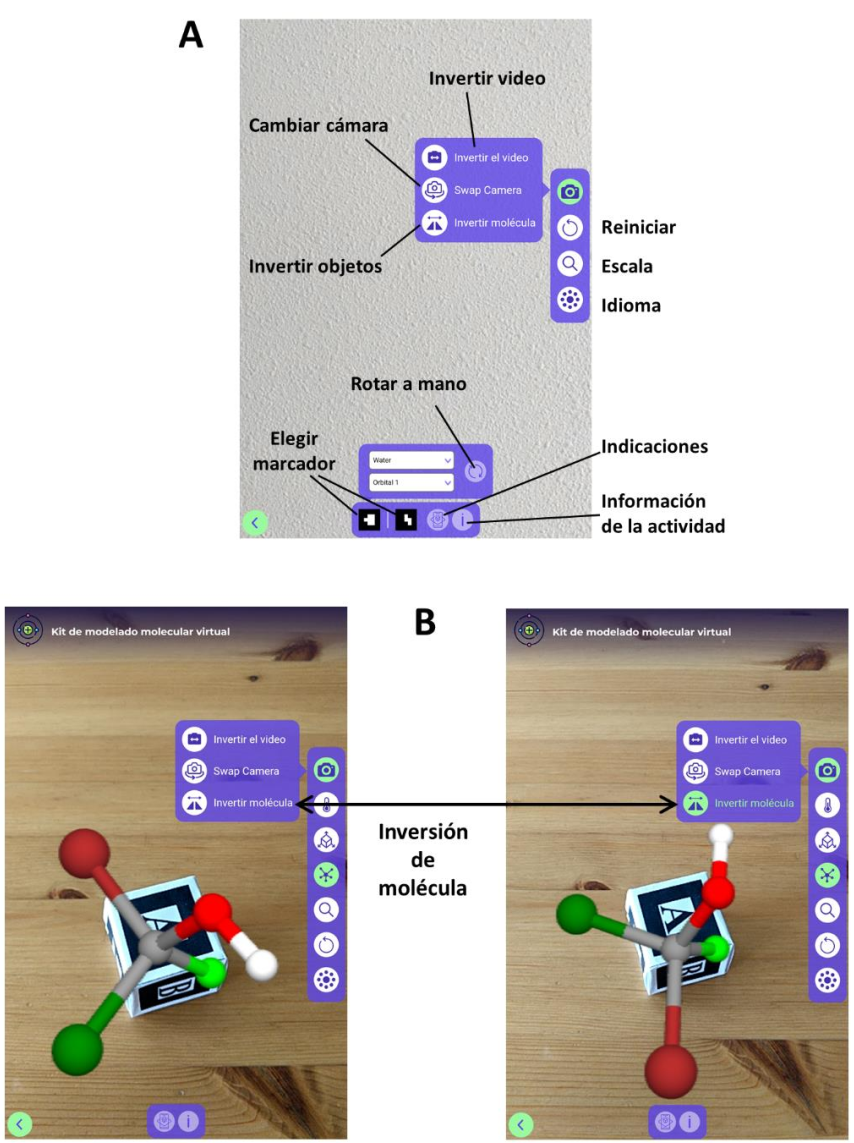
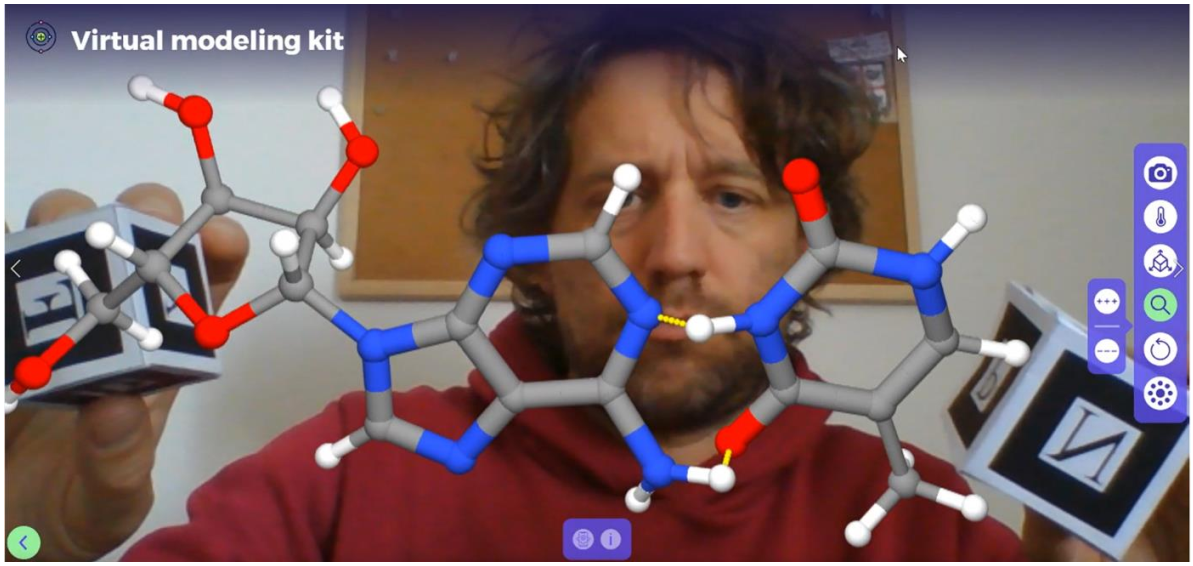
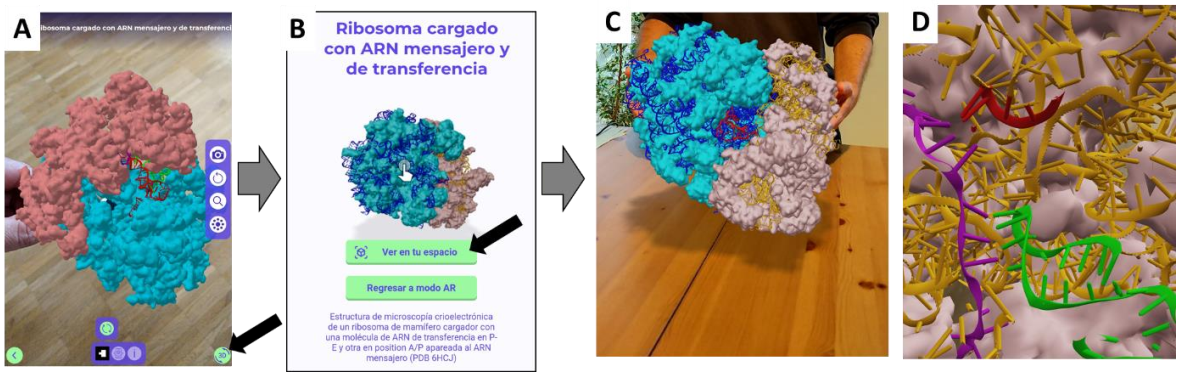
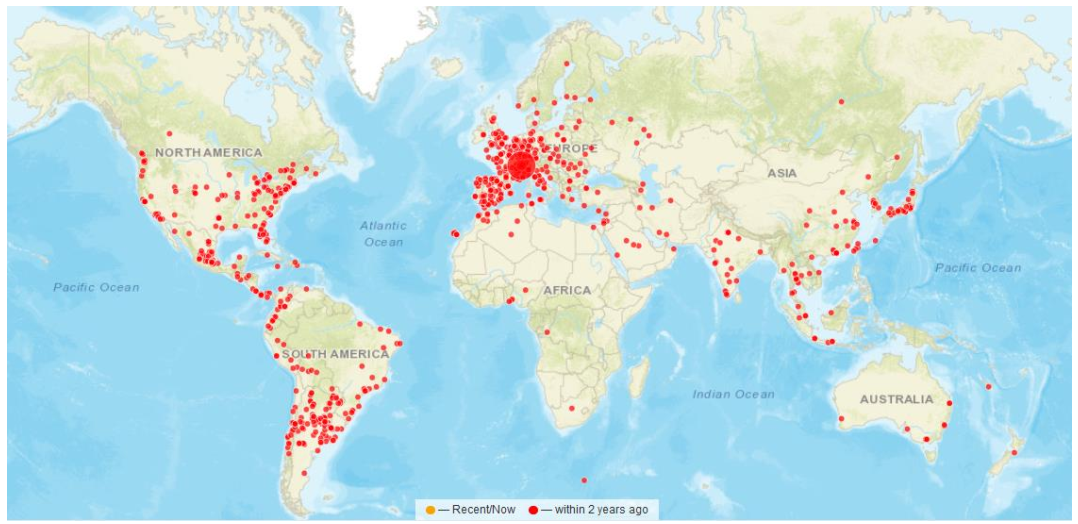
1. Introducción Los modelos de entidades de naturaleza abstracta, o imperceptibles por sus pequeños tamaños, o de difícil disponibilidad/adquisición, son extremadamente útiles para la enseñanza y el autoaprendizaje, al hacer más inteligibles los conceptos y entidades en cuestión. Esto es especialmente importante en el abordaje de la química, donde profesores y alumnos se enfrentan a conceptos complejos y sobre todo difíciles de visualizar. Ejemplos concretos abarcan desde las formas y energías de los orbitales atómicos puros y sus combinaciones en orbitales híbridos y moleculares, hasta la organización de átomos en el espacio especialmente en sistemas quirales; incluyendo también el hecho de que las moléculas son entidades flexibles que exploran variadas conformaciones, pasando por el carácter dinámico de los equilibrios, los cambios de estados de protonación, la distribución de propiedades moleculares como la carga o la aromaticidad. Sumado además a la interacción de todos estos y otros elementos en moléculas complejas como aquellas que forman los bloques de la biología. En estos y tantos otros ejemplos, la visualización de las entidades y los mecanismos implicados contribuyen a la enseñanza didáctica, facilitando así la comprensión y la construcción de sus propios conocimientos por parte de los estudiantes. Para eso, los materiales didácticos y las presentaciones en clases incorporan variados modelos, ilustraciones y gráficos. Pero estos mismos presentan dos inconvenientes: por un lado, muchos son representaciones bidimensionales de entidades intrínsecamente tridimensionales; por otro lado, este material suele ser estático o en los mejores casos, animado, pero rara vez interactivo, es decir que ni profesores ni alumnos pueden actuar sobre las entidades químicas o biológicas en cuestión. Muchos cursos hoy en día suplen parcialmente esa deficiencia recurriendo a programas de gráficos moleculares en computadora, los cuales permiten rápidamente visualizar prácticamente cualquier molécula en pseudo 3D (“pseudo” haciendo referencia a que la representación es normalmente en una pantalla plana). Incluso existen varias librerías de programación web que permiten insertar gráficos moleculares directo en páginas web, con lo cual se han creado variados recursos educativos en distintas ramas de la química y la biología molecular. El principal inconveniente de estos programas gráficos es que se limitan al manejo de objetos intrínsecamente tridimensionales mediante el empleo de un mouse sobre una superficie plana, lo cual dificulta operaciones de rotación y traslación simultáneas sobre dos moléculas; por lo que es prácticamente imposible comparar pares de moléculas o hacerlas interaccionar. Por el contrario, los modelos tangibles diseñados en plástico u otros materiales permiten una gran accesibilidad y capacidad de manipulación, donde el educador o el estudiante puede físicamente mover moléculas para entender su geometría, estereoquímica y quiralidad, e incluso forzar rotaciones alrededor de enlaces simples, todos estos más difíciles de lograr con programas de gráficos en computadora. Existen incluso modelos físicos diseñados en materiales como el plástico que representan ácidos nucleicos y proteínas, y hasta modelos físicos de células. Incluso, las impresoras 3D, abrieron la posibilidad de imprimir modelos en plástico para generar modelos físicos de casicualquier objeto con volumen, lo cual se ha explotado por ejemplo para producir modelos tangibles de orbitales atómicos.[1] Asimismo, estos modelos físicos poseen también sus desventajas en comparación a los gráficos: su –relativamente- elevado costo, la falta de resolución en los detalles, la dificultad de modificar la visualización y de anotar información, entre otras. En particular, los modelos moleculares son limitados en el número y tipo de átomos disponibles, además de que el usuario debe armar los modelos lo cual supone posibilidades de error para moléculas grandes, y obviamente disponer de un kit molecular o un medio alternativo de construcción. En los últimos años, las tecnologías llamadas de realidad virtual y realidad aumentada se han vuelto progresivamente más accesibles, con lo cual numerosos grupos han comenzado a elaborar distintos tipos de programas y aplicaciones que permitan integrar lo mejor del mundo virtual con lo mejor del mundo físico en lo que respecta a la manipulación y visualización de moléculas. Hoy en día hay una oferta variada de tales aplicaciones, en las cuales se identifican algunas limitaciones en relación a su uso: por un lado, los programas más completos suelen requerir equipamiento especial que al conectarse a una computadora proveen realidad virtual o aumentada muy inmersiva pero con un costo de cientos a miles de dólares, con lo cual es impensable lograr un buen alcance a estudiantes y educadores, incluso en países desarrollados. Por otro lado, la posibilidad de lograr realidad aumentada en teléfonos modernos, tablets y computadoras usando simplemente la webcam de estos dispositivos, permite a cualquier usuario lograr esa experiencia, que es menos inmersiva pero en principio más que los gráficos de computadora estándar; y más importante aún, permiten un mejor control espacial de la visualización y de los objetos de estudio. Pero aún aquí la mayoría de los programas disponibles tienen problemas: por un lado, casi todos presentan solo visualizaciones estáticas, con poca o ninguna interactividad con el usuario; además, casi todos estos programas y aplicaciones deben ser instaladas, lo cual puede no ser posible en sistemas compartidos donde los usuarios no tienen privilegios de administrador -o simplemente el usuario puede no querer o confiar en la instalación de aplicaciones. Teniendo en cuenta estos antecedentes, y tratando de unir lo mejor del mundo virtual y real en una forma accesible y fácil de usar, los autores FCR y LAA recientemente introdujimos un sitio web llamado MoleculARweb (https://molecularweb.epfl.ch) que propone una serie de actividades de realidad aumentada útiles para enseñar y estudiar varios conceptos de química y biología molecular.[2,3] Las actividades cubren desde la química general e inorgánica hasta la biología estructural, pasando por la química orgánica y la fisicoquímica. El sitio no consiste en actividades puntuales sino en herramientas con las cuales los educadores pueden hacer demostraciones y armar ejercicios para sus alumnos. Cada actividad en MoleculARweb utiliza un set de marcadores (Figura 1A) que se imprimen con impresoras comunes, y se arman de acuerdo a los videos de instrucciones disponibles en la página de inicio del sitio. Al entrar a la página web de cada actividad el usuario debe permitir al navegador web el uso de la webcam, para mostrar a esta los marcadores impresos. De esta manera, el sistema mostrará en la pantalla los objetos virtuales relevantes para la actividad de acuerdo al marcador utilizado, que luego el usuario podrá mover para manipularlos en el espacio (Figura 1B). Como mostramos en la sección 2, las actividades en MoleculARweb abarcan desde la química más fundamental (átomos, moléculas, sus orbitales, mecánica e interacciones) hasta la estructura de ensamblado moleculares biológicos complejos (ribosomas, nucleosomas, partículas virales). Y tal como ejemplificamos en la sección 3, el portal Google pone al alcance de cualquier usuario con un teléfono o tablet modernos, modelos que comienzan justo un nivel arriba de los ensamblados macromoleculares en la escala de complejidad biológica: desde organelas (mitocondrias, aparato de Golgi) hasta células, tejidos y sistemas (linfático, nervioso, etc.) e incluso varios seres vivos completos con gran resolución de detalles. Aunque estos modelos son mucho menos interactivos que los deMoleculARweb, permiten extender su contenido para poner juntas la química y la biología como actividades sin fronteras claras, donde la segunda está muy apoyada en la primera, tal como ejemplificamos en la sección 4 con un recorrido que conecta desde conceptos a nivel atómico hasta modelos de sistemas biológicos complejos. Esto último inspirado en una actividad real propuesta a los visitantes de un evento de ciencia abierta, llevada a cabo en EPFL en el año 2019. Aparte, durante 2021 utilizamos estas herramientas en talleres y actividades especiales con alumnos y con docentes, en modalidad mixta presencial y en línea. Más específicamente, en talleres de la materia Química Orgánica en la Universidad Nacional de San Luis, Argentina, y en actividades de conexión entre la Universidad Nacional de Río Cuarto, Argentina, y estudiantes y profesores de nivel secundario. Esto se detalla en la sección 5 de este artículo. Figura 1: Cómo usar MoleculARweb con marcadores. (A) Marcadores cúbicos para los kits de modelado virtual o planos para las otras actividades de MoleculARweb. El sitio está disponible gratuitamente, sin registración en 7 idiomas incluyendo el español en https://molecularweb.epfl.ch. Los marcadores se pueden imprimir desde https://molecularweb.epfl.ch/assets/markers/allmarkers.pdf. (B) Ejemplo de actividad corriendo en un teléfono, aquí mostrando cómo dos moléculas de agua interaccionan e intercambian protones mediante puentes de hidrógeno. Esta es la actividad 1 del módulo “Puentes hidrógeno, ácidos y bases”: https://molecularweb.epfl.ch/modules/acids-and-bases/waterwater/water.html 2. Actividades disponibles en MoleculARweb, y cómo utilizarlas El contenido de MoleculARweb se reparte en 4 módulos temáticos que presentamos aquí con algunas actualizaciones respecto a la versión liberada en 2020, además del llamado “Kit de Modelado Molecular Virtual” del cual presentamos aquí el prototipo de una nueva versión. Los módulos temáticos son “Formas de orbitales y moléculas”, “Puentes hidrógeno, ácidos y bases”, “Estructura atómica de moléculas biológicas”, y “Complejos macromoleculares biológicos”. Cada módulo incluye varias actividades que hemos descripto en un artículo reciente en inglés 2 y algunas de las cuales presentamos en videos en español en el canal de YouTube Moléculas en Realidad Aumentada [4]. Las actividades de estos módulos (sección 2.1) funcionan con los marcadores de doble lado que se pueden imprimir en el link dado en la Figura 1A. Algunas de estas actividades también tienen un modo sin marcadores, menos interactivo pero más inmersivo (sección 2.3). Por otro lado el kit de modelado molecular virtual (sección 2.2) utiliza marcadores tipo cubo que también pueden descargarse como se indica en la Figura 1A. Estos marcadores cúbicos permiten cargar cualquier par de moléculas desde sus coordenadas atómicas para visualizarlas en realidad aumentada, incluso simular sus movimientos, y ver cómo chocan e interaccionan. 2.1 Actividades con marcadores planos, en teléfonos, tablets y computadoras con webcam Todas las actividades soportan un modo de uso basado en marcadores que los usuarios pueden imprimir con una impresora normal, y mostrar a la webcam. Esta detectará los patrones de los marcadores y superpondrá los objetos virtuales correspondientes sobre el video del entorno en tiempo real. La primera vez que el usuario accede a una actividad el navegador pregunta si se permite el uso de la webcam; el usuario debe aceptar Figura 2: Ejemplificando una actividad de cada módulo y distintos modos de uso en computadoras y teléfonos. (A) Orbital híbrido sp3d capturado en la pantalla del teléfono móvil. Esta actividad está en https://molecularweb.epfl.ch/modules/orbitals-and-molecular-shapes/atomic-orbitals/atomic-orbitals.html (B) Dos moléculas de agua interaccionando e intercambiando protones mediante puentes hidrógeno, foto externa. Actividad en https://molecularweb.epfl.ch/modules/acids-and-bases/water-water/water.html (C) Educador acercando una proteína a una molécula de ADN doble hebra, capturado de la pantalla de una computadora. Actividad en https://molecularweb.epfl.ch/modules/atomic-structures/protein-dna/protein-dna.html (D) Un marcador dentro de una guía de lectura permite incorporar un modelo 3D al material didáctico. Actividad en https://molecularweb.epfl.ch/modules/biological-assemblies/nucleosome/nucleosome.html Ejemplos de actividades. La Figura 2 muestra un ejemplo de una actividad de cada módulo, ilustrando además distintos modos de uso. La Figura 2A muestra un orbital híbrido sp3d (actividad “Orbitales atómicos y teoría TRPEV” del módulo “Formas de orbitales y moléculas”) manipulado en un teléfono, tomando la foto directamente como captura de pantalla. La Figura 2B muestra cómo se manipulan dos moléculas de agua viéndolas a través del teléfono (actividad “Equilibrios de protonación del agua” del módulo “Puentes hidrógeno, ácidos y bases”); en este caso con una foto externa pero que bien podría tomarse directamente desde el propio teléfono también. La Figura 2C muestra un usuario acomplejando una molécula de ADN a una molécula de proteína (actividad “Complejo ADN-proteína” del módulo “Estructura atómica de moléculas biológicas”) en una computadora que solo tiene una cámara frontal por lo cual sólo funciona como espejo. La Figura 2D muestra la incorporación de un marcador a una guía de estudio acerca de los nucleosomas para que el alumno pueda visualizarlo directamente allí con su teléfono directamente sobre la guía (actividad “Nucleosoma” del módulo “Complejos macromoleculares biológicos”). Figura 3: Controles más generales. (A) Controles generales comunes a todas las actividades. (B) Ejemplo de aplicación del botón de invertir molécula en el kit de modelado molecular virtual, aquí para intercambiar entre los enantiómeros R y S deuna molécula de CFClBrOH (elegida sólo con fines demostrativos para tener átomos de distintos colores alrededor del carbono). Controles. La Figura 3A muestra los controles más comunes a todas las actividades (se ejemplifica en teléfono móvil donde hay mayor variedad de controles). Algunos de estos controles se destacan aquí con sus posibles utilidades. Primero, en caso de haber más de una webcam, por ejemplo en teléfonos con cámara delantera y trasera, el usuario puede cambiarlas en tiempo real mientras trabaja. Otro control permite invertir sólo la imagen, lo cual puede ser útil por ejemplo al estudiar enantiomería (Figura 3B, ejemplo tomado del kit de modelado molecular). Por su parte, los controles de rotación permiten lograr un ajuste fino de la orientación del objeto sin mover el marcador, lo cual puede ser útil por ejemplo cuando el marcador está integrado en un documento preparado por el educador como en la Figura 2D. Cada actividad tiene algunos controles específicos, generalmente en la parte inferior y con el fin de permitir al usuario elegir entre opciones. Por ejemplo, la Figura 3A muestra una vista en la pantalla del teléfono donde se aprecia en la parte inferior opciones para elegir qué orbital de cuál molécula se desea visualizar, junto con un control que activa la rotación a mano e indicaciones sobre la actividad. Aparte, las actividades con visualizaciones estáticas poseen un botón con el texto “3D” abajo a la derecha, que activa el modo sin marcadores descripto en la sección 2.3. Por último, el kit de modelado virtual tiene varios controles especiales, descriptos en la sección 2.2. 2.2 El kit de modelado virtual con marcadores tipo cubo, también en teléfonos, tablets y computadoras con webcam El kit de modelado virtual permite cargar cualquier molécula a partir de sus coordenadas atómicas, para mostrarla en 3D sobre un cubo de papel o cartón que el usuario puede mover y rotar libremente. Gracias a sus 6 caras, se logra un mejor manejo y estabilidad visual que con los marcadores planos. Aparte de mostrar las moléculas, el kit de modelado incluye un campo de fuerzas muy simple que permite visualizar los cambios conformacionales originados por el acople de los movimientos de los átomos. La versión original del kit de modelado virtual usa el cubo formado por las letras A, B, C, D, F y un símbolo como se observa en la Figura 1A. La versión de prueba 2.0 funciona con uno o dos de los marcadores tipo cubo de la Figura 1A (el segundo marcador cúbico se compone de las letras J, K, M, N, L y P), permitiendo controlar dos moléculas independientemente. En este último caso, el usuario puede cargar una molécula cualquiera en cada cubo, desde sus coordenadas atómicas, tomada de la lista preestablecida o construida externamente (en la versión 1) o incluso adentro mismo de la actividad (esto posible desde la versión 2). Algunos controles fueron reordenados en la versión 2; por ejemplo, la inversión de coordenadas puede ejecutarse específicamente en cada marcador, lo cual permite cargar en ambos la misma molécula quiral pero invertir sólo una para facilitar comparaciones. Además, la versión 2 del kit de modelado incorpora un campo de fuerzas muy sencillo que calcula y muestra interacciones entre moléculas, tanto choques como atracciones. La Figura 4 muetra un prototipo de la versión 2, donde cada cubo muestra una molécula distinta ambas interaccionando mediante dos puentes hidrógeno detectados en tiempo real. Los lectores ya pueden acceder a esta versión directamente desde la página principal de MoleculARweb. Figura 4: Prototipo de versión 2 del kit de modelado molecular virtual. Cada cubo puede cargar una molécula, y las moléculas de ambos cubos pueden interaccionar entre ellas. En este caso se ven dos bases nitrogenadas sobre las que el usuario investiga posibles patrones de interacciones por puente de hidrógeno. Las líneas punteadas amarillas representan los posibles puentes detectados en tiempo real. Esta actividad (aún en desarrollo pero ya utilizable) está disponible en https://molecularweb.epfl.ch/modules/virtual-modeling-kit-2 mientras que la versión previa (que funciona con un solo cubo y posee menos opciones y una mecánica más simple) está en https://molecularweb.epfl.ch/modules/virtual-modeling-kit. 2.3 Viendo modelos en el entorno, sin marcadores, usando teléfonos que soportan WebXR En la Figura 5 se muestra cómo activar el modo de realidad aumentada sin marcadores para el modelo de un ribosoma construido a partir de una estructura experimental donde se lo capturó con un segmento de ARN mensajero y dos de ARN de transferencia. Desde cierta distancia el usuario puede apreciar la forma general del ribosoma, las subunidades principales y sus componentes proteicos y nucleicos. Además el usuario también puede adentrarse para analizar detalles, como aquí por ejemplo un codón del ARNm interaccionando con un codón de uno de los ARNt (en cada molécula de ARN cada base está representada con un palo). Este tipo de actividades que no tienen contenido interactivo pueden también ejecutarse en un modo sin marcadores en MoleculARweb. Para ello se requiere el uso de teléfonos modernos que soporten WebXR (la mayoría de los teléfonos disponibles desde 2018). En los casos donde este modo está disponible, el usuario verá un ícono “3D” en la parte inferior derecha de la pantalla. Al cliquear el ícono “3D” se muestra una pantalla intermedia con el objeto en cuestión donde se visualiza además un botón que activa el modo de realidad aumentada sin marcadores (en teléfonos y tablets configurados en español este botón dice “Ver en mi entorno”). Al entrar en ese modo el usuario debe escanear el piso, una mesa, o cualquier otra superficie plana horizontal; entonces el programa colocará una representación virtual del objeto y lo dejará estático en un lugar, incluso si el usuario se mueve. Esto permite al usuario explorar el objeto desde cualquier ángulo e incluso “entrar” en los objetos para ver detalles. Todo esto acompañado de la posibilidad de tomar capturas de pantalla y grabar videos desde un programa externo instalado en el teléfono como el AZ Screen Recorder. Figura 5: Activación de realidad aumentada web sin marcadores en MoleculARweb. (A) Si la actividad presenta el ícono 3D en la parte inferior derecha (marcado con flecha negra), al cliquearla se abre una ventana intermedia (B) donde el usuario puede mover el objeto tocando la pantalla o pasar al modo de realidad aumentada sin marcadores cliqueando en “Ver en tu espacio”. (C) Una vez en el modo de realidad aumentada web, la visualización se ancla a la superficie detectada y el usuario puede moverse inspeccionarla libremente por su alrededor e incluso por dentro (D). El ejemplo es una estructura resuelta mediante microscopía crioelectrónica de un ribosoma de mamífero cargado con una molécula de ARN de transferencia en P/E y otra en position A/P apareada al ARN mensajero (PDB 6HCJ)[5]. En tonos azules/celestes la subunidad mayor y en tonos rosados/naranja la subunidad menor, las proteínas se muestran como superficies y las moléculas de ARN como tiras. El ARNm se ve extendido en color magenta y los ARNt en verde y rojo. La actividad mostrada corresponde a https://molecularweb.epfl.ch/modules/biological-assemblies/ribosome/ribosome.html. 3. Ejemplos de objetos 3D disponibles en Google para explorarlos en realidad aumentada La misma tecnología de realidad aumentada sin marcadores descripta arriba es explotada por Google para ofrecer una gran variedad de contenido educativo en realidad aumentada, provista por terceras partes en forma abierta. Buscando en un teléfono que soporte WebXR, Google devolverá links directos a objetos 3D predefinidos (notará que las búsquedas funcionan sólo en inglés; lo mismo ocurre con las anotaciones en los modelos). La Figura 6 muestra ejemplos donde se han buscado las palabras clave “muscular system” (Figura 6A) o conceptos relacionados a la estructura de las plantas vasculares (Figura 6B). Al igual que en los modelos de realidad aumentada sin marcadores de MoleculARweb, en los modelos de Google el usuario puede moverse con total libertad alrededor del objeto una vez que éste está anclado a la superficie detectada. Esto permite explorar en detalle ciertas estructuras como se ejemplifica en ambos paneles de la Figura 6 (detalle de la inserción de los tendones en 6A, y detalles del cloroplasto en 6B). Figura 6: Ejemplos de modelos de realidad aumentada en sitios web propuestos por Google, útiles en el área de la biología. (A) Tejido muscular humano, con algunas anotaciones predefinidas. Notar la facilidad con la cual esta tecnología permite explorar las distintas partes. Google provee gratuitamente modelos similares para varios otros sistemas del cuerpo humano. (B) Esquemas 3D del interior de una hoja de una planta, de una célula vegetal y de la ultraestructura de un cloroplasto, respectivamente. 4. Ejemplo de uso en comunicación de la ciencia: recorrido de átomos a tejidos y de regreso a átomos Finalmente, en la Figura 7 ejemplificamos cómo integrar algunas de las actividades de realidad aumentada presentadas arriba en una experiencia completa que permite entender la estructura de l materia viva a distintos niveles de complejidad. Este ejemplo en concreto comienza explorando la relación entre estados de protonación y cargas, siguiendo con la visualización de interacciones electrostáticas que estabilizan complejos ADN-proteína para luego visualizar como éstos se ensamblan en nucleosomas; para posteriormente ver los cromosomas dentro del núcleo celular, y alejarse lentamente hasta ver una célula entera, luego el sistema circulatorio como ejemplo del nivel de organización de tejidos, y finalmente volver a hacer zoom en una célula, una mitocondria, el grupo hemo de una proteína, y finalmente los orbitales 3d de un átomo. Un recorrido similar pero más abreviado fue utilizado durante un evento de comunicación de ciencias en la institución de los autores FCR y LAA. En ese caso los participantes comenzaban con una actividad similar a la de los paneles A y B de la Figura 7, divergiendo luego hacia una actividad de realidad aumentada para explorar la estructura de un virus obtenida mediante tomografía electrónica, y finalmente una actividad que emulaba la resolución de una estructura molecular mediante datos de difracción de rayos X. Todas estos ejemplos son actividades disponibles en MoleculARweb. Por último, los participantes realizaban ensayos de cristalización de lisozima, verificaban la obtención de estos cristales y luego se les explicaba mediante un póster cómo se obtienen los datos utilizados para resolver estructuras mediante difracción de rayos X. Figura 7: Ejemplo de un recorrido desde átomos hasta tejidos y nuevamente a átomos. (A) Un residuo de lisina y uno de glutamato, estableciendo un puente de hidrógeno e intercambiando un protón lo cual resulta en cambios de cargas (video mostrando el proceso dinámico en https://www.youtube.com/watch?v=J_idZmyDTUE ). (B) Los residuos cargados positivamente confieren a las proteínas de unión al ADN una interacción fuerte y de baja especificidad con los grupos fosfato del ADN (actividad preparada desde el PDB 1FJL). (C) Las proteínas tipo histona y el ADN se empaquetan en estructuras más grandes llamadas nucleosomas. Aquí se explora la estructura de un nucleosoma cristalizado con dos vueltas de ADN de doble hebra (PDB 1AOI). (D-E) Modelo de un núcleo celular rodeado del sistema de Golgi, en una vista cercana a los cromosomas(D) y otra alejándose para lograr ver toda la red de Golgi (E). (F) Modelo de una célula eucariota animal, mostrando las principales organelas y subestructuras con sus nombres (solo disponibles en inglés). (G-H) Modelo del sistema circulatorio humano en una vista global (G) y cerca del corazón (H). (I) Modelo de una mitocondria con nombres en algunas de sus subestructuras. (J-K) Subunidades I y II de la enzima citocromo oxidasa (J) y zoom en el sitio de reducción de oxígeno (K) que en este caso posee una molécula de óxido nítrico unida (PDB 3AG3, las esferas azul y roja son respectivamente los átomos de N y O de la molécula de NO unida directo al ión hierro). (L) Ejemplos de orbitales involucrados en la unión del oxígeno molecular a Fe+2 en proteínas: π* en la molécula de oxígeno (derecha) y dz2 en el ión Fe+2 de la proteína (izquierda). Los paneles A-C y J-L son capturas de pantalla de MoleculARweb, mientras que D-I son capturas de pantalla de modelos 3D de Google. Todas las capturas fueron obtenidas en el mismo teléfono pero fueron ajustadas y recortadas al componer la figura. 5. Experiencias áulicas en la enseñanza de química a nivel secundario y universitario Aparte del uso que ya documentamos al presentar la primera versión de moleculARweb [3], durante 2021 utilizamos estas herramientas en talleres y actividades especiales con alumnos y con docentes, en modalidad mixta presencial y en línea. Más específicamente, en talleres de la materia Química Orgánica en la Universidad Nacional de San Luis, Argentina, y en actividades de conexión entre la Universidad Nacional de Río Cuarto, Argentina, y estudiantes y profesores de nivel secundario. 5.1 Experiencia áulica en la enseñanza de química orgánica complementada con realidad aumentada en la Universidad Nacional de San Luis, Argentina El aprendizaje de estructuras químicas y orgánicas siempre implicó dificultades de comprensión y visualización de las diferentes geometrías, disposición espacial y cambios conformacionales en 3D. Dado que el desarrollo del pensamiento abstracto, necesario para visualizar la química no es una habilidad aprendida homogéneamente en el aula, los docentes nos vemos obligados a complementar la enseñanza con herramientas didácticas y accesibles al alumnado. Con el advenimiento de la pandemia COVID-19, la enseñanza áulica debió ser repensada y adaptada a la enseñanza virtual, dejando de este modo obsoleto el uso de modelos moleculares plásticos que el profesor normalmente llevaba a las aulas. En este contexto, en la asignatura de química orgánica de las carreras de Lic. En Biotecnología, Lic. En Bioquímica, Lic. En Biología Molecular, Lic. En Cs. Biológicas y Prof. Univ. de Biología de la Universidad Nacional de San Luis (UNSL) aplicamos el kit de modelado molecular virtual descripto arriba (evento documentado en la Figura 8). Dado que jamás se había trabajado con realidad aumentada, evaluamos su adaptación a través de test de uso y encuestas de satisfacción (resultados reservados para próxima publicación). El estudio abarcó alumnos de primer y segundo año de la Facultad de Química, Bioquímica y Farmacia. Todos los alumnos incluidos en el test de evaluación lograron satisfactoriamente adaptar el uso del sitio web a sus herramientas de trabajo para comprender de una manera más didáctica y divertida la estereoisomería y análisis conformacional de las moléculas orgánicas estudiadas en las guías de trabajo práctico. Dado el éxito logrado, esperamos confeccionar nuevas guías de trabajos prácticos que complementen el uso del kit de modelado virtual incorporando también su nueva versión. La adaptación de nuevas herramientas virtuales que faciliten el aprendizaje y sobre todo que genere entusiasmo en el alumnado son vitales para el aprendizaje. En futuros estudios intentaremos medir el efecto pedagógico real más allá del estímulo que pueda provocar la tecnología en sí misma. Figura 8: Test de evaluación presencial en las aulas de la Universidad Nacional de San Luis, utilizando MoleculARweb con alumnos del ciclo lectivo 2021 junto con el equipo docente a cargo de la Prof. Celeste Aguirre Pranzoni. 5.2 Exactas en línea con la Escuela: Empleo de la realidad aumentada en la escuela secundaria Otro ejemplo del uso de MoleculARweb fue a través de la propuesta: ¡Las Moléculas y estructuras biológicas se vuelven gigantes! ¿Las vemos en realidad aumentada? En esta actividad docentes y estudiantes de los últimos años de escuelas secundarias se familiarizaron con el sitio web, vivenciando, en formato virtual, el uso y aplicación práctica de la realidad aumentada al estudio de moléculas y estructuras biológicas en una nueva dimensión. Esta actividad fue desarrollada a través del proyecto Exactas en línea con la escuela, que impulsa la Facultad de Ciencias Exactas, de la Universidad Nacional del Río Cuarto con la intención de sostener y potenciar el vínculo de la Universidad con las escuelas secundarias (Resolución Nº 038/2021). Las actividades consistieron en un recorrido virtual con ejemplos sobre los diferentes módulos temáticos del sitio. Posteriormente se llevaron a cabo diferentes actividades prácticas diseñadas de acuerdo a los contenidos de los programas curriculares del nivel medio, incorporando el uso de la realidad aumentada a través de MoleculARweb. Entre las moléculas elegidas para la secuencia utilizada, se trabajó con los modelos en 3D del ATP, glucosa, dióxido de carbono; se analizaron interacciones moleculares entre moléculas de agua, entre el ADN y proteínas; la estabilidad de las moléculas y los cambios de temperatura; como así también se observaron estructuras biológicas complejas: poros nucleares, bacteriófagos, ribosomas, entre otras; las observaciones realizadas fueron integradas a conceptos teóricos de ambas disciplinas (química y biología). De esta manera fue posible presentar nuevas formas de aprendizajes, que resultan más atractivas e innovadoras. El uso y aplicación del sitio web al estudio de conceptos de Biología y Química fue valorada de manera positiva por los estudiantes y docentes que participaron de la propuesta, manifestando lo siguiente: “…Tuvo un impacto positivo ya que me parece muy interesante poder ver los átomos de cada molécula con realidad aumentada…” “…Facilita la enseñanza y comprensión de lo invisible a los ojos...” “…Pude comprender de una mejor manera aquellos conceptos que en general me resultaban abstractos…” “…Tuvo un impacto muy positivo. Un excelente aporte para explicar contenidos relacionados a ambas asignaturas que son muy difíciles de comprender porque requieren mucha imaginación. Además, considero que este tipo de programas motivan a los alumnos por llegar de otro modo a aprendizajes complejos…” De esta manera, a través de la propuesta presentada, consideramos que la implementación de MoleculARweb a los diseños curriculares de Biología y Química es una de las opciones de mayor proyección educativa tanto en la enseñanza secundaria como en los primeros años de las carreras de grado afines, además de ser una valiosa herramienta para motivar hacia el estudio y un aprendizaje más activo. Comentarios finales Con más de 28.000 accesos desde su estreno, MoleculARweb permea escuelas y hogares de todo el mundo (Figura 9) gracias a su disponibilidad en varios idiomas, su funcionamiento en dispositivos móviles y computadoras normales, y su fácil acceso directo a través simplemente de internet como cualquier otro sitio web gracias a que se basa exclusivamente en programación web [6-8]. Los tests publicados [9], estadísticas automatizadas online, nuestras propias evaluaciones en persona, y los comentarios recibidos de profesores de colegios secundarios y de cursos de nivel universitario demuestran la facilidad en el uso de la plataforma de MoleculARweb y el alcance del mismo desde el punto de vista pedagógico. En este sentido, muchos docentes manifestaron la importancia de contar con esta herramienta dado que se logra una mayor percepción, interacción y sobre todo motivación por parte de los alumnos, lo cual podría facilitar la comprensión y aprendizaje de conceptos que resultan difíciles de visualizar, tanto en química como en biología. Estamos actualmente completando el kit de modelado molecular virtual para liberar formalmente su versión 2.0 y creando un sistema con el cual los educadores podrán crear su propio contenido de realidad aumentada web para objetos más complejos. Por su parte Google y empresas como las mencionadas continuamente agregan nuevos modelos, incluyendo ahora también modelos animados de procesos biológicos y seres vivos completos. Estas tecnologías permiten más que nunca un aprendizaje didáctico e inmersivo, al alcance de cualquier persona que disponga de un teléfono y acceso a internet. Figura 9: Accesos a MoleculARweb tal como registrado en https://clustrmaps.com/site/1ar2f al 30 de agosto de 2021 Referencias: [1] de Cataldo R, Griffith KM, Fogarty KH (2018) Hands-On Hybridization: 3D-Printed Models of Hybrid Orbitals. J. Chem. Educ. 95 (9), 1601–1606. https://doi.org/10.1021/acs.jchemed.8b00078. [2] Cortes Rodriguez F, Frattini G, Krapp L, Martinez-Huang H, Moreno D, Salomon J, Stemkoski L, Traeger S, Dal Peraro M, Abriata L (2020) MoleculARweb: A Website for Chemistry and Structural Biology Education Through Interactive Augmented Reality out of the Box in Commodity Devices. ChemRxiv 2020. [3] Cortés Rodríguez F, Frattini G, Krapp LF, Martinez-Hung H, Moreno DM, Roldán M, Salomón J, Stemkoski L, Traeger S, Dal Peraro M (2021) MoleculARweb: A Web Site for Chemistry and Structural Biology Education through Interactive Augmented Reality out of the Box inCommodity Devices. J Chem Educ 2021. [4] Abriata LA. Moléculas en realidad aumentada https://www.youtube.com/channel/UCdhOVimtNZmri967KdTEAKg . [5] Juszkiewicz S, Chandrasekaran V, Lin Z, Kraatz S, Ramakrishnan V, Hegde RS (2018) ZNF598 Is a Quality Control Sensor of Collided Ribosomes. Mol Cell 2018 72(3), 469-481. e7. [6] Abriata LA (2020) Building Blocks for Commodity Augmented Reality-Based Molecular Visualization and Modeling in Web Browsers. PeerJ Comput Sci 6, e260. [7] Abriata LA (2017) Web Apps Come of Age for Molecular Sciences. Informatics 4(3), 28. [8] AbriataLA, Rodrigues JPGLM, Salathé M, Patiny L (2018). Augmenting Research, Education, and Outreach with Client-Side Web Programming. Trends Biotechnol 36 (5), 473–476. https://doi.org/10.1016/j.tibtech.2017.11.009. [9] Martínez-Hung H, García-López A, Escalona-Arranz JC (2017) Modelos de Realidad Aumentada Aplicados a La Enseñanza de La Química En El Nivel Universitario. Rev Cuba Quím 29(1), 13–25.





![]()
ISSN 1666-7948
www.quimicaviva.qb.fcen.uba.ar
Revista QuímicaViva
Número 3, año 20, Diciembre 2021
quimicaviva@qb.fcen.uba.ar