La biorremediación en la era post-genómica
Irma S. Morelli1,2, Bibiana M. Coppotelli1, Laura Madueño1 y María T. Del Panno1
1Centro de Investigación y Desarrollo en Fermentaciones Industriales, CINDEFI (UNLP; CCT-La Plata, CONICET), La Plata, Buenos Aires,
1Comisión de Investigaciones Científicas de la Provincia de Buenos Aires (CIC-PBA), La Plata, Buenos Aires, Argentina
Resumen
La biorremediación tiene hoy en día gran aceptación como una estrategia efectiva para la recuperación de suelos contaminados, sin embargo la falta de información sobre los factores que rigen el funcionamiento metabólico de las comunidades microbianas en los ambientes contaminados hace que, aún en la actualidad, los procesos de biorremediación tengan resultados impredecibles. Las técnicas moleculares basadas en el estudio del DNA, han permitido la identificación de numerosos genes catabólicos abriendo nuevas oportunidades en el desarrollo de los procesos de biorremediación. En esta nueva era post-genómica, las emergentes metatranscriptómica, metaproteómica y metabolómica resultan promesas notables como herramientas para estudiar los mecanismos implicados en la regulación de las vías de degradación, lo que en un futuro nos permitirá desarrollar modelos predictivos sobre la actividad degradadora de la comunidad microbiana del suelo en función de los distintos parámetros bióticos y abióticos, y establecer criterios específicos y claros sobre la elección y evaluación de las estrategias de biorremediación.
Palabras claves: Biodegradación, Comunidades Microbianas, Ómicas.
Abstract
Bioremediation is nowadays widely accepted as an effective strategy for the recovery of contaminated soils; however the lack of information about the factors that govern the metabolic functioning of microbial communities in contaminated environments means that, still at present, the bioremediation processes have unpredictable results. The molecular techniques based on DNA studies have allowed the identification of numerous catabolic genes opening new opportunities in the development of bioremediation processes. In this new post-genomic era, emerging metatranscriptomics, metaproteomics and metabolomics are remarkable promises as tools to study the mechanisms involved in the regulation of degradation pathways, which in the future will allow us to develop predictive models of degrading activity of the soil microbial community according to the various biotic and abiotic parameters, establishing specific and clear criteria for the selection and evaluation of bioremediation strategies.
Keywords: Biodegradation, Microbial Communities, Omics.
Introducción
La biorremediación, la explotación de las capacidades metabólicas de los microorganismos para degradar contaminantes, tiene hoy en día gran aceptación como una estrategia efectiva, costo-competitiva y ambientalmente amigable para la recuperación de aguas, suelos y sedimentos contaminados [1,2] (Fig. 1). Los microorganismos, y en especial las bacterias, son reconocidos por su amplia capacidad catabólica y su actividad en los procesos de biorremediación, sin embargo la falta de información sobre los factores que controlan el crecimiento y la actividad de los microorganismos en los ambientes contaminados hace que, aún en la actualidad, los procesos de biorremediación tengan resultados impredecibles y que la comunidad microbiana responsable del proceso sea considerada como una “caja negra” [3]. Idealmente, las estrategias de biorremediación deberían ser diseñadas en base al conocimiento de los microorganismos presentes en los ambientes contaminados, sus capacidades metabólicas, y la respuesta a los cambios en las condiciones ambientales [4].

Figura 1: Imagen conceptual de la Biorremediación
Esto ha conducido a los microbiólogos ambientales a reconocer al estudio de las comunidades microbianas de ambientes contaminados como un parámetro ecológico imperativo para dilucidar los mecanismos que determinan el éxito de la biorremediación y definir entonces condiciones operativas óptimas [2]. Sin embargo, debido a su gran diversidad, variabilidad metabólica y las limitaciones de los métodos de cultivo; la caracterización, identificación y análisis cuantitativo de las comunidades microbianas de muestras ambientales continúa siendo un gran desafío [5].
Biorremediación de suelos contaminados: abriendo la caja negra
Durante los procesos de biorremediación la comunidad microbiana evoluciona a través de sucesiones progresivas, desde la comunidad nativa hacia una comunidad degradadora adaptada a las condiciones ambientales. La evolución y adaptación de la comunidad microbiana a través del mantenimiento de una dinámica sucesional determinan la eficiencia de la biorremediación [6]. Entonces, ¿Cuáles son los factores que afectan la evolución de la comunidad microbiana? ¿Cómo podrían éstos limitar la degradación de un contaminante aún en presencia de una comunidad microbiana con capacidades genéticas adecuadas?
En este contexto la historia de la biorremediación podría ser dividida en tres eras: pre-genómica, genómica y post-genómica[4]. Convencionalmente los estudios de biodegradación se reducían al aislamiento y caracterización de cepas o consorcios bacterianos con potencial degradativo bajo condiciones controladas de laboratorio [7,8,9,10]. Estos estudios fueron seguidos por la caracterización de las rutas metabólicas de degradación y sus mecanismos de regulación, presentes en los microorganismos aislados [11,12,13]. Posteriormente el desarrollo de los métodos moleculares cultivo-independiente, principalmente los basados en la determinación de los perfiles de genes rRNA o de genes catabólicos específicos, permitieron el estudio de la diversidad y las capacidades catabólicas de las comunidades microbianas durante los procesos de biodegradación [14,15,16,17]. Estas técnicas hicieron posible la identificación de los grupos microbianos más sensibles, resilientes, predominantes o activamente involucrados en los procesos de biorremediación [2]. Actualmente, los estudios metagenómicos (análisis del genoma microbiano colectivo contenido en una muestra ambiental), son considerados como una estrategia amplia que permitirá el descubrimiento de nuevos genes involucrados en la biodegradación de contaminantes. La construcción de bibliotecas metagenómicas, por clonación de fragmentos de DNA extraídos de muestras ambientales, en un adecuado vector (plásmido, cósmico, fósmido, etc.) y el posterior análisis del inserto (por secuenciación directa o previo rastreo funcional), ha sido utilizada por varios autores para identificar los genes involucrados en los procesos de biorremediación [18,19]. Por otro lado la nueva generación de tecnologías de secuenciación masiva tienen el potencial de revolucionar nuestra capacidad de entender el mundo microbiano [20]. El primer estudio en que se utilizó secuenciación masiva “shotgun metagenomics”, para estudiar la dinámica de la comunidad microbiana durante un experimento de biorremediación, fue publicado en el año 2012 por Yergeau y col [21]. Utilizando la plataforma Roche 454 GS FLX pudieron demostrar que tanto la contaminación con hidrocarburos como las distintas estrategias de bioestimulación ensayadas (aireación y la fertilización) producen cambios drásticos en la comunidad microbiana del suelo, observándose una prevalencia de géneros de bacterias aeróbicas que han demostrado la capacidad potencial de degradar hidrocarburos (Pseudomonas, Rhodococcus, Sphingomonas, Caulobacter); y que la dominancia relativa de estos géneros bacterianos varía durante el tratamiento, probablemente como resultado de cambios en la calidad y disponibilidad de los diversos hidrocarburos y demás nutrientes durante la biorremediación.
Por otro lado, un método molecular clave para identificar los microorganismos directamente involucrados en la degradación de un contaminante es la utilización de sondas con isótopos estables (SIP, del inglés “stable isotope probing”). Se utilizan microcosmos de suelo que son contaminados con sustratos marcados (13C, 18O, 15N, etc.), luego de la incubación se extrae el DNA. El DNA que resulta marcado se separa del no marcado por ultracentrifugación en un gradiente de CsCl. Los microorganismos que asimilaron el sustrato son identificados a través del estudio de la secuencia del DNA marcado [22].
La era posgenómica: oportunidades y desafíos
Las técnicas independientes de cultivo, basadas en el estudio del DNA, han permitido la identificación de numerosos genes catabólicos abriendo nuevas oportunidades en el desarrollo de los procesos de biorremediación [4]. Sin embargo, estas técnicas no nos permiten obtener conclusiones sobre el estado de expresión de los genes, por lo que el rol funcional de los genes u organismos presentes en las muestras ambientales resulta todavía incierto [23].
En el esfuerzo por dilucidar la relación entre la estructura de la comunidad microbiana y la función del suelo los estudios del metatranscriptoma y del metaproteoma proveerían una información más específica acerca de la real funcionalidad de la comunidad [24], y en el caso de los procesos de biorremediación específicamente en relación a las vías de degradación de contaminantes y su cascada de regulación[25,26]. Sin embargo, la aplicación de estas técnicas se encuentra todavía en su infancia [4].
Hasta este punto, los estudios sobre la respuesta de la comunidad microbiana a los contaminantes había sido centrada en los cambios en la diversidad microbiana [27,28] o en las respuestas de un pequeño número de genes específicos identificados en base a la información obtenida a partir de cultivos puros [15,29]. Los avances en las técnicas de extracción directa de mRNA a partir de muestras de suelo han permitido la obtención del perfil de expresión de genes de la comunidad microbiana. Recientemente, de Menezes y col. [30] pudieron revelar por primera vez, a través de un análisis metatranscriptómico, la respuesta global de la comunidad microbiana de un suelo frente a la contaminación con un hidrocarburo, demostrando que la respuesta a nivel de comunidad es compleja, y tiene consecuencias generalizadas más allá de los organismos involucrados en la degradación.
Por otro lado, el desarrollo de le metaproteómica ha resultado técnicamente complejo, principalmente porque el suelo es una fuente pobre de proteínas, los métodos de extracción de proteínas a partir de muestras de suelo resultan dificultosos y han sido escasamente desarrollados, y porque no hay información suficiente en las bases de datos de secuencias para permitir la identificación de proteínas a través de espectrometría de masa [31]. Por tales motivos, los estudios metaproteómicos resultan actualmente un enfoque caro, en términos de costo y tiempo, para la comprensión de la funcionalidad de la comunidad microbiana. El desarrollo de métodos que aumenten el rendimiento en la extracción de proteínas a partir de suelos y permitan su normatización, junto con los avances en la secuenciación de genomas y su anotación, nos permitirán identificar proteínas indicadoras de los procesos del suelo, proporcionando algunas respuestas sobre cuáles son las proteínas responsable de la degradación de un contaminante, qué microorganismos las producen y en qué condiciones [32].
Por último, la metabolómica ofrece una definitiva y compuesto específica verificación del metabolismo in situ permitiendo evaluar y predecir el impacto del contaminante en la función del suelo mediante el seguimiento de un metabolito adecuado. Un biomarcador metabólico adecuado para el monitoreo de los procesos de biorremediación debe ser específico para el compuesto de origen y para el proceso biológico que se monitorea; debe estar ausente en el ambiente contaminado debiendo ser liberado por las células microbianas como un intermediario de la biodegradación y no como un producto del cometabolismo [33]. Actualmente la metabolómica se encuentra limitada por las capacidades analíticas y depende del conocimiento de las vías metabólicas de degradación.
Conclusiones
Un desarrollo eficaz y coherente de estrategias de biorremediación requiere del conocimiento profundo de los parámetros que rigen las estructuras y funcionamiento metabólico de las comunidades microbianas [2].
Aunque las nuevas técnicas de la biología molecular son herramientas emergentes, aún se mantienen como notables promesas en el estudio de los procesos de biorremediación. Uno de los mayores inconvenientes de las «ómicas» es su aplicación a ciegas, generando una gran cantidad de datos, los que deben ser luego, organizados e interpretados. Si bien el objetivo de la microbiología ambiental no es el desarrollo de las nuevas técnicas moleculares, el avance científico se consigue normalmente cuando las mismas son aplicadas a estudios con hipótesis claras [34].
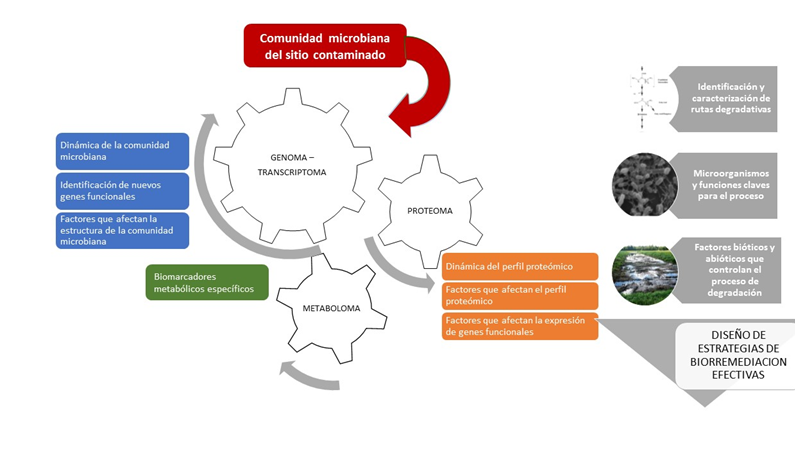
Figura 2: Utilización de las tecnologías ómicas, según un enfoque biológico sistematizado aplicado al estudio de las comunidades microbianas implicadas en el proceso de biorremediación.
Definitivamente la biorremediación ha entrado en la era de las ómicas (Fig. 2); desde la genómica a la metabolómica estas nuevas metodologías nos brindan la posibilidad de realizar una mirada aguda sobre las comunidades microbianas, lo que en un futuro nos permitirá desarrollar modelos predictivos sobre la actividad degradadora de la comunidad microbiana del suelo en función de los distintos parámetros bióticos y abióticos, y establecer criterios específicos y claros sobre la elección y evaluación de la estrategia de biorremediación; produciendo un enorme impacto en nuestros esfuerzos para hacer de la biorremediación una tecnología más fiable y segura.
Referencias:
1. Pandey J, Chauan A, Jain RK (2009) Integrative approaches for assessing the ecological sustainability of in situ bioremediation FEMS Microbiology Reviews 33:324-375.
2. Desai C, Pathak H, Madamwar D (2010)Advances in molecular and ‘‘-omics” technologies to gauge microbial communities and bioremediation at xenobiotic/anthropogen contaminated sites Bioresource Technology 101:1558-1569.
3. El Frantroussi S, Agathos SN (2005) Is bioaugmentation a feasible strategy for pollutant removal and site remediation? Current Opinion in Microbiology 8:268-275.
4. Chauhan A, Jain RK (2010) Biodegradation: gaining insight through proteomics. Biodegradation 21:861-879.
5. He Z, Deng Y, Zhou J (2012 )Development of functional gene microarrays for microbial community analysis Current Opinion in Biotechnology 23:49-55.
6. Paliwal V, Puranik S, Purohit HJ (2012) Integrated perspective for effective bioremediation Applied Biochemistry and Biotechnology 166:903-924.
7. Solari AA, Paiceira MT (1976) Study of a pseudomonas isolated from soil capable of utilizing naphthalene as source of carbon Acta Bioquímica Clínica Latinoamericana 10:321-326.
8. Heitkamp MA, Cerniglia CE (1988) Mineralization of polycyclic aromatic hydrocarbons by a bacterium isolated from sediment below an oil field Applied Environmental Microbiology 54: 1612-1614.
9. Stolz A, Knackmuss HJ (1993) Degradation of 2,4-dihydroxybenzoate by Pseudomonas sp. BN9 FEMS Microbiology Letters 108:219-224.
10. Samanta SK, Chakraborti AK, Jain RK (1999) Degradation of phenanthrene by different bacteria: evidence for novel transformation sequences involving the formation of 1-naphthol Applied Microbiology and Biotechnology 53:98-107.
11. Haggblom M (1990) Mechanisms of bacterial degradation and transformation of chlorinated monoaromatic compounds Journal of Basic Microbiology 30:115-141.
12. Arai H, Ohishi T, Chang MY, Kudo T (2000) Arrangement and regulation of the genes for meta-pathway enzymes required for degradation of phenol in Comamonas testosterone TA441 Microbiology 146:1707-1715.
13. Solyanikova IP, Golovleva LA (2004) Isolation, biochemical and molecular characterization of 2-chlorophenol-degrading Bacillus isolates Journal of Environmental Science Health B 39:333-351.
14. Del Panno MT, Morelli IS, Engelen B, Berthe-Corti L (2005) Effect of petrochemical sludge concentrations on microbial communities during a soil bioremediation process FEMS Microbiology Ecology 53:305-316.
15. Cébron A, Norini M-P, Beguiristain T, Leyval C (2008) Real-time PCR quantification of PAH-ring hydroxylating dioxygenase (PAH-RHDa) genes from Gram positive and Gram negative bacteria in soil and sediment samples Journal of Microbiological Methods 73:148-159.
16. Ibarrolaza A, Coppotelli BM, Del Panno MT, Donati E, Morelli IS (2009) Dynamics of microbial community during bioremediation of phenanthrene and chromium(VI)-contaminated soil microcosmos Biodegradation 20:95-107.
17. Vázquez S, Nogales B, Ruberto L, Hernández E, Christie-Oleza J, Lo Balbo A, Bosch R, Lalucat J, Mac Cormack W (2009) Bacterial community dynamics during bioremediation of diesel oil-contaminated antarctic soil Microbial Ecology 57:598-610.
18. Suenaga H, Tsutomu O, Kentaro M (2007) Functional screening of a metagenomic library for genes involved in microbial degradation of aromatic compounds Environmental Microbiology 9:2289-2297.
19. Vasconcellos SP, Figueiredo Angolini CF, Sierra García IN, Martins Dellagnezze B, Canedo da Silva C, Marsaioli AJ, Vaz dos Santos Neto E, Maia de Oliveira M (2010) Screening for hydrocarbon biodegraders in a metagenomic clone library derived from Brazilian petroleum reservoirs Organic Geochemistry 41:675-681.
20. Liu Z, DeSantis TZ, Anderson GL, Kinght R (2008) Accurate taxonomy assignments from 16S rRNA sequences produced by highly parallel pyrosequencers Nucleic Acids Researchs 36 e120.
21. Yergeau E, Sanschagrin S, Beaumier D, Greer CW (2012) Metagenomic analysis of the bioremediation of diesel-contaminated Canadian high Arctic soils PLoS One 7:e30058.
22. Woods A, Watwood M, Schwartz E (2011) Identification of a toluene-degrading bacterium from a soil sample through H218O DNA stable isotope probing Applied and Environmental Microbiology 77:5995-5999.
23. Urich T, Lanzen A, Qi J, Huson DH, Schleper C, Schuster FSC (2008) Simultaneous assessment of soil microbial community structure and function through analysis of the meta-transcriptome PLoS ONE 3:e2527
24. Nannipieri P, Ascher J, Ceccherini MT, Landi L, Pietramellara G, Renella G (2003) Microbial diversity and soil functions European Journal of Soil Science 54:655-670.
25. Wilmes P, Bond PL (2006) Metaproteomics: studying functional gene expression in microbial ecosystems Trends Microbiology 14:92–97.
26. Benndorf D, Balcke GU, Harms H, von Bergen M (2007) Functional metaproteome analysis of protein extracts from contaminated soil and groundwater ISME Journal 1:224–234.
27. Sun MY, Dafforn KA, Johnston EL, Brown MV (2013) Core sediment bacteria drive community response to anthropogenic contamination over multiple environmental gradients Environmental Microbiology 15:2517-2531.
28. Festa S, Coppotelli BM, Morelli IS (2015) Comparative bioaugmentation with a consortium and a single strain in a phenanthrene-contaminated soil: impact on the microbial community and biodegradation Applied Soil Ecology. Enviado
29. Wang B, Lai Q, Cui Z, Tan T, Shao Z (2008) A pyrene-degrading consortium from deep-sea sediment of the West Pacific and its key member Cycloclasticus sp. P1 Environmental Microbiology 10:1948-1963.
30. de Menezes A, Clipson N, Doyle E (2012) Comparative metatranscriptomics reveals widespread community responses during phenanthrene degradation in soil Environmental Microbiology 14:2577-2588.
31. Bastida F, Moreno JL, Nicolás C, Hernández T, García C (2009) Soil metaproteomics: a review of an emerging environmental science Significance, methodology and perspectives European Journal of Soil Science 60:845-59.
32. Bastida F, Hernández T, García C (2014) Metaproteomics of soils from semiarid environment: Functional and phylogenetic information obtained with different protein extraction methods Journal of Proteomics 101:31-42.
33. Callaghan AV (2013) Metabolomic investigations of anaerobic hydrocarbon-impacted environments Current Opinion in Biotechnology 24:506-515.
34. Kowalczyk A, Martin TJ, Price OR, Snape JR, van Egmond RA, Finnegan CJ, Schäfer H, Davenport RJ, Bending GD (2015) Refinement of biodegradation tests methodologies and the proposed utility of new microbial ecology techniques Ecotoxicology and Environmental Safety 111:9-22.
I.S. Morelli es profesora e investigadora de CIC–PBA, B. M. Coppotelli es investigadora de CONICET y jefa de trabajos prácticos, M.T. Del Panno es profesora y L. Madueño es becaria postdoctoral de CONICET y ayudante diplomado.
|
Revista QuímicaViva Número 1, año 14, Abril 2015 quimicaviva@qb.fcen.uba.ar |