Metabolitos de interés biotecnológico à la carte: Análisis de flujos metabólicos como herramienta para la evaluación de fenotipos industriales
Pablo I. Nikela,b
a Instituto de Investigaciones Biotecnológicas, Universidad Nacional de San Martín, Colectora Av. General Paz 5445, B1650KNA, INTI - Ed. 24, San Martín, Buenos Aires, Argentina
b Departamento de Química Biológica, Universidad de Buenos Aires, Av. Intendente Güiraldes 2160, C1428EGA, Ciudad de Buenos Aires, Argentina
E-mail: Pablo@qb.fcen.uba.ar
Resumen
La versatilidad del metabolismo microbiano ofrece la posibilidad de obtener una gran variedad de metabolitos de interés biotecnológico. Para el desarrollo racional de bioprocesos destinados a la obtención de dichos metabolitos es necesario contar con herramientas de análisis global que permitan el estudio holístico del metabolismo. El análisis de flujos metabólicos basado en marcación con sustratos que contienen 13C se ha convertido en una importante herramienta de análisis aplicada en Ingeniería Metabólica, y permite la cuantificación de los flujos intracelulares del metabolismo central de un microorganismo proveyendo una visión completa del mismo. El conjunto de metodologías utilizadas para este tipo de análisis ha evolucionado considerablemente en los últimos años y la introducción de nuevas técnicas ha permitido la implementación de análisis de flujos metabólicos en períodos de tiempo relativamente cortos. En esta actualización se resumen algunas de las aplicaciones recientemente descriptas en la bibliografía para el análisis de flujos metabólicos relacionado al estudio y optimización de bioprocesos industriales. El texto concluye con una perspectiva de desarrollos futuros.
Palabras clave: Análisis de flujos metabólicos; Ingeniería Metabólica; metabolitos de interés industrial; experimentos de marcación con carbono; isotopómero; cromatografía de gases acoplada a espectrometría de masas
Metabolites of biotechnological interest à la carte: Metabolic flux analysis as a tool for industrial phenotypes evaluation
Abstract
Microbial metabolism is highly versatile, and offers the possibility to obtain a wide range of biotechnological metabolites. In order to design bioprocesses rationally to obtain such metabolites, it is necessary to apply tools for global analysis, which give a holistic view of the metabolism. Metabolic flux analysis based on labeling with 13C-substrates has become an important tool in Metabolic Engineering, and allows the quantification of metabolic fluxes thus providing a whole picture of the central microbial metabolism. The methodologies involved in this kind of analysis have evolved considerably during the last few years, and the introduction of new techniques enables the implementation of accurate metabolic flux analysis with shortened experimental time. In this work, a number of recently-published applications of metabolic flux analysis on industrial bioprocesses study and optimization is shortly reviewed and discussed. An outlook to possible future developments concludes the text.
Keywords: Metabolic flux analysis; Metabolic Engineering; metabolites of industrial interest; carbon labeling experiments; isotopomer; gas chromatography couple to mass spectrometry
Introducción
La redundancia de enzimas individuales y de caminos metabólicos completos en numerosas especies bacterianas es una indicación inequívoca de la naturaleza altamente flexible del metabolismo microbiano (25). Dado que estas vías metabólicas son activas solamente bajo ciertas condiciones específicas, en cada ambiente o condición de crecimiento opera una determinada distribución de flujos metabólicos que permite a la célula acoplar la generación de energía y el catabolismo de la fuente de carbono con los requerimientos anabólicos para alcanzar un crecimiento óptimo. Por este motivo, conocer, identificar, y, consecuentemente, manipular las vías metabólicas activas en distintas condiciones de crecimiento es un requerimiento esencial para el diseño de bioprocesos destinados a la obtención biotecnológica de productos derivados del metabolismo microbiano.
La Ingeniería Metabólica comprende una serie de metodologías pertenecientes a diversas disciplinas y podría definirse, sensu lato, como la mejora racional y dirigida de la formación de un producto, o una propiedad celular, a través de la modificación de reacciones bioquímicas del microorganismo, o introduciendo nuevas rutas biosintéticas. El análisis de flujos metabólicos (AFM) se ha convertido en las últimas décadas en una de las herramientas más relevantes en Ingeniería Metabólica. El principal objetivo del AFM es la cuantificación detallada de todos los flujos metabólicos del metabolismo central en un organismo. El resultado final de un estudio de este tipo es, por lo tanto, un mapa de reacciones bioquímicas que muestra la distribución de flujos anabólicos y catabólicos en el contexto de una red de reacciones bioquímicas. Además de tratarse de una herramienta de gran importancia para el análisis fisiológico (41), surge en forma evidente la posibilidad de identificar (y modificar) reacciones para la obtención de uno o varios metabolitos a través de AFM.
En esta actualización se discuten brevemente las metodologías de análisis fenotípico global basadas en interpretación de flujos metabólicos (por cálculo estequiométrico directo y por marcación isotópica), y se exponen algunos ejemplos de la aplicación de estas metodologías en bioprocesos de interés industrial.
Análisis estequiométrico de flujos metabólicos
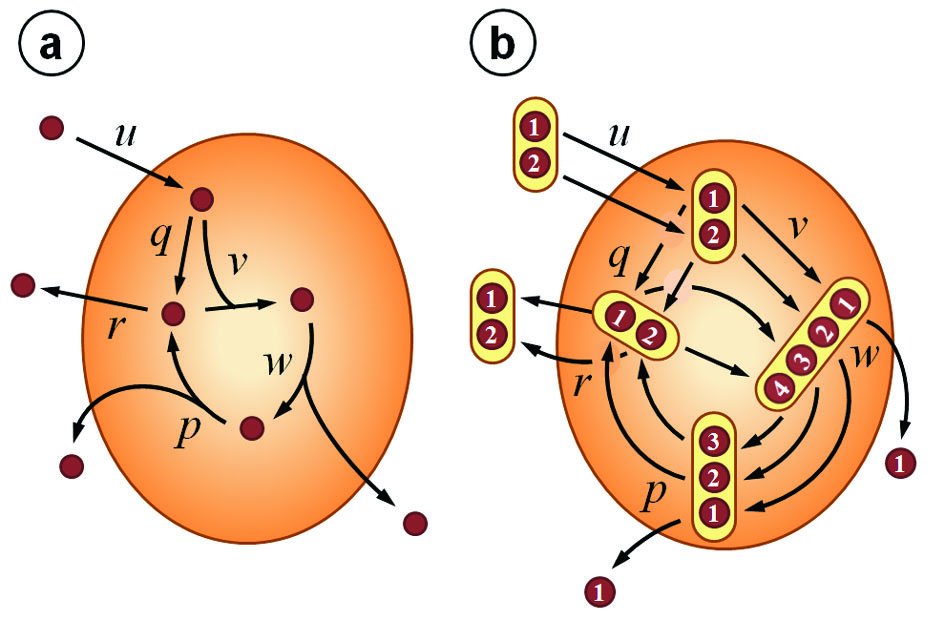
Esta metodología, establecida por Varma y Palsson en los años 90 para la interpretación del metabolismo central de Escherichia coli (38), hace uso del conocimiento de la estequiometría en cada una de las reacciones de una red metabólica determinada para la estimación de flujos. Una de las restricciones más importantes en este tipo de análisis es que el sistema debe encontrarse en un estado estacionario o cuasi-estacionario (esto es, la concentración intracelular de cada metabolito, y consecuentemente el flujo asociado al mismo, debe ser constante mientras dure el experimento). Estas condiciones pueden ser fácilmente establecidas en un cultivo continuo que ha alcanzado el estado de equilibrio estacionario y, con ciertas restricciones, durante la fase exponencial de un cultivo en lote o en un cultivo en lote alimentado (esto es, fed-batch) si la variación en el flujo de alimentación durante el experimento es pequeña. Para cualquiera de estas condiciones de equilibrio estacionario, la suma de los flujos ‘de entrada’ para cada metabolito iguala a la suma de los flujos ‘de salida’ (en otras palabras, no existe acumulación neta del metabolito). Esta situación puede describirse matemáticamente con una única ecuación lineal para cada metabolito considerado, procedimiento que colectivamente se denomina balance de masas. La Fig. 1a ilustra este concepto a partir de una red bioquímica sencilla, en donde puede observarse que existen flujos intracelulares y extracelulares.
Fig. 1. Ejemplo de un diagrama de flujos para una red bioquímica sencilla. a) Los flujos u, r, y p pueden ser determinados directamente mediante la estimación de la velocidad de consumo de sustrato y de producción de metabolitos extracelulares, respectivamente. A partir de la medición de al menos dos de estos flujos puede deducirse el valor de todos los flujos restantes. b) Para el AFM basado en marcación isotópica es necesario conocer el destino de cada uno de los átomos de carbono en los metabolitos de la red considerada con el objetivo de determinar el enriquecimiento posicional en el isótopo considerado (e.g., 13C).
En este caso, asumiendo que el sistema se encuentra en estado de equilibrio estacionario, se obtiene el siguiente conjunto de relaciones lineales:
u = q + v v = w w = p p + q = r + v
Es decir, existen cuatro relaciones lineales para seis variables de flujo. Esto significa que todos los patrones de flujo posibles en esta red bioquímica forman un sub-espacio bidimensional (lineal) dentro del espacio hexadimensional que forman las seis variables de flujo. Dado que en la mayoría de los casos prácticos la medición directa de los flujos intracelulares no es posible, normalmente se utilizan modelos matemáticos que permiten inferir dichos flujos intracelulares a partir de otros que pueden ser medidos con mayor facilidad. De esta manera, si se pudiesen medir directamente al menos dos de los seis flujos considerados en el ejemplo de la Fig. 1a, el sistema de ecuaciones se convertiría en un sistema determinado (en cuyo caso se puede deducir el valor de los seis flujos a partir de la medición de dos de ellos). Los flujos extracelulares (ejemplo: velocidad de consumo de sustrato, velocidad de síntesis de metabolitos extracelulares, evolución de CO2, etc.) usualmente se utilizan como datos para el cálculo de todos los flujos restantes. Para el ejemplo que se está analizando, supongamos que se pueden medir los flujos extracelulares u y r. Entonces,
v = u – r w = u – r p = u – r q = r
El resultado final es un mapa de flujos metabólicos que representa gráficamente cada una de las reacciones bioquímicas incluidas en el modelo junto a una estimación de la velocidad en condiciones de estado estacionario (viz., flujo) a la cual cada reacción tiene lugar. Este procedimiento sencillo condujo a la primera publicación, a principio de los años 90, describiendo el metabolismo central de Corynebacterium glutamicum (36), una bacteria tradicionalmente utilizada para la obtención industrial de aminoácidos.
Experimentos de marcación isotópica
A pesar de la relativa sencillez de su implementación, desafortunadamente existen numerosas limitaciones para la interpretación estrictamente estequiométrica de los flujos metabólicos en un microorganismo. Algunas de ellas son:
a) existencia de vías metabólicas paralelas, que conducen al mismo metabolito pero utilizando (iso)enzimas e intermediarios diferentes. Un caso típico es la presencia de dos vías enzimáticas independientes para la síntesis de lisina por C. glutamicum (15),
b) presencia de ciclos metabólicos en los cuales no se puede medir directamente al menos un flujo interno en el ciclo,
c) existencia de vías metabólicas bidireccionales que operan al mismo tiempo en ambos sentidos. Este es el caso para la enorme mayoría de las reacciones bioquímicas de una célula. De hecho, solamente pueden considerarse como unidireccionales aquellas reacciones cuya reversibilidad esté fuertemente impedida desde el punto de vista termodinámico. Un ejemplo particular de esta situación es el caso de las reacciones altamente reversibles catalizadas por las enzimas transaldolasa y transcetolasa en la vía de pentosas fosfato, y
d) dificultades en el balance de co-factores. En una red metabólica típica, que normalmente consta de la vía glicolítica de Embden-Meyerhof-Parnas, vía de pentosas fosfato, ciclo de ácidos tricarboxílicos, y reacciones anapleróticas, solamente se pueden determinar estequiométricamente los flujos asumiendo que la síntesis y el consumo de metabolitos asociados al balance energético y balance redox (e.g., ATP, ADP, NADH, NAD+, etc.) se encuentran perfectamente balanceados. Sin embargo, la evidencia experimental sugiere que existen numerosos ciclos fútiles, y que la conversión energética de equivalentes de reducción, como NADH y NADPH, a ATP no es constante incluso para el mismo microorganismo en condiciones de cultivo similares (26).
Estas limitaciones pueden ser evitadas, al menos parcialmente, utilizado un sustrato isotópicamente marcado para el experimento de AFM, tal como 13C- o 14C-glucosa. La utilización de 14C-glucosa se implementó en AFM en los años 80 (3), pero pronto fue desplazada por estrategias de marcación isotópica no radioactiva en lo que genéricamente se denomina experimentos de marcación con carbono. En este tipo de experimentos, el sistema biológico se alimenta con el sustrato marcado, y los átomos de éste se distribuyen entre los metabolitos de la red bioquímica hasta que en condiciones de equilibrio estacionario los átomos de 13C/14C se encuentran en estado de equilibrio isotópico (17). En este punto, el enriquecimiento isotópico de cada uno de los metabolitos en la red puede determinarse empleando técnicas de resonancia magnética nuclear (RMN) (31) o espectrometría de masas (EM) (5, 7, 44). El proceso descripto permite la identificación de cada uno de los metabolitos en la red y evita la mayoría de las complicaciones asociadas al análisis puramente estequiométrico dado que se conoce el destino de las moléculas marcadas y se puede seguir su ‘trayectoria’ intra y extracelular. En el contexto de este tipo de análisis surgen algunos términos específicamente asociados a los experimentos de marcación con carbono en los que se basa la técnica de 13C-AFM. Por ejemplo, isotopómero es un término derivado de isótopo e isómero, e identifica a cada uno de los posibles estados de marcación en los que puede encontrarse una molécula de un determinado metabolito. Dado que un metabolito que contiene n átomos de carbono podrá contener un isótopo o un átomo en su estado isotópico natural en cada posición, el número de isotopómeros para ese metabolito está dado por 2n.
Uno de los requerimientos más importantes para la implementación de 13C-AFM es que se debe conocer exactamente el destino de cada uno de los átomos de carbono dentro de los metabolitos de la red considerada (41). La Fig. 1b ilustra esta situación para la red bioquímica sencilla considerada anteriormente. En este esquema puede apreciarse la complejidad de la transición entre los átomos de carbono en cada metabolito. A partir de esta red de transición de átomos de carbono se puede deducir la red de isotopómeros esperada. Esta red indica cómo los isotopómeros reaccionan entre sí dando lugar a nuevos metabolitos marcados.
Si bien existen numerosas metodologías para asociar la presencia de isotopómeros y la distribución de metabolitos dentro de la red bioquímica en estudio, la medición de metabolitos marcados normalmente se realiza utilizando cromatografía de gases (CG) acoplada a EM (5, 10). Dentro del conjunto de las numerosas biomoléculas que proveen información acerca del estado isotópico de intermediarios metabólicos, usualmente se utilizan los aminoácidos proteinogénicos. Esto implica que después de someter a la biomasa a hidrólisis ácida completa, los aminoácidos componentes de la misma deben ser derivatizados para convertirlos en moléculas volátiles y permitir su separación por CG (7). Los compuestos que se eluyen de la columna del cromatógrafo son ionizados, lo cual genera fragmentos con diferentes relaciones m/z (masa/carga). Esta técnica es particularmente importante dado que no solamente puede determinarse la masa de los isotopómeros del ion molecular si no que además se analiza el espectro de masas de diferentes fragmentos del mismo isotopómero, lo cual aumenta y diversifica la información disponible acerca del metabolito analizado. Es preciso notar aquí que existen varias técnicas alternativas para la determinación de isotopómeros; como 1H-RMN y 13C-RMN, cromatografía líquida (CL) acoplada a EM, o MALDI-TOF* acoplado a EM (43).
* MALDI-TOF es una técnica de ionización suave utilizada en espectrometría de masas. Se denomina MALDI por sus siglas en inglés Matrix-Assisted Laser Desorption/Ionization (desorción/ionización láser asistida por matriz) y TOF por el detector de iones que se acopla al MALDI y cuyo nombre procede también de sus siglas en inglés Time-Of-Flight.
Evaluación in silico de experimentos de marcación isotópica y construcción del mapa de flujos
A pesar de que el análisis de la distribución de isotopómeros utilizando modelos matemáticos es una metodología sumamente compleja, es necesario conocer los lineamientos generales en los que se fundamenta para comprender la forma en la que se evalúan los experimentos de marcación con carbono. Básicamente, la interpretación de los resultados se basa en simulaciones matemáticas (32). En una simulación de este tipo, en cada iteración se asume que se conocen los valores para los flujos en la red bioquímica estudiada. A partir de un conjunto limitado de flujos ‘libres’ (a los cuales se les asigna un valor arbitrario) y de la composición isotópica del sustrato carbonado se puede reconstruir la distribución de isotopómeros de cada metabolito en el estado de equilibrio estacionario. Para ello, el modelo matemático utilizado para correlacionar la distribución de isotopómeros y los flujos intracelulares contiene una ecuación (normalmente, no lineal) para cada uno de los isotopómeros en el sistema. Por lo tanto, para una red bioquímica ‘mediana’ que incluya solamente las vías metabólicas centrales, se tiene un sistema de alrededor de 1000 ecuaciones no lineales. La dimensión del sistema se relaciona con el hecho de que ciertas moléculas pueden tener numerosos isotopómeros derivados. Por ejemplo, solamente la molécula de corismato* tiene 210 = 1024 isotopómeros asociados. Esto hace que los procesos de análisis isotopomérico y evaluación de flujos metabólicos requieran de herramientas informáticas adecuadas para el manejo de tal cantidad de datos y variables. Generalmente, los programas informáticos utilizados para la interpretación in silico de flujos metabólicos se basan en operaciones algebraicas con matrices (35).
*(Ácido corísmico) Es un intermedio clave en la síntesis del triptófano, la fenilalanina y la tirosina, es decir, en la síntesis de ácidos aromáticos, siendo el primer punto de ramificación, con una rama que conduce a triptófano, otra que conduce a la fenilalanina y otra a la tirosina.
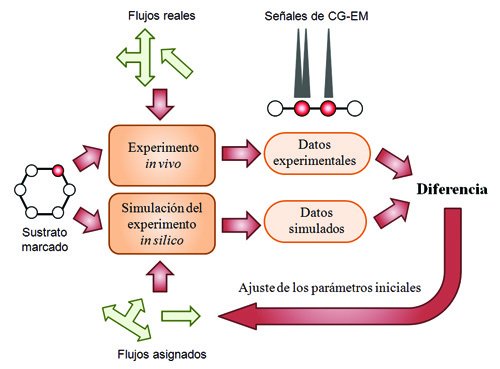
Una vez establecido un procedimiento para la simulación de la marcación isotópica, la estrategia para la determinación y evaluación de flujos (Fig. 2) normalmente incluye los siguientes pasos:
a) asignación arbitraria de algunos valores de flujos al sistema de modo tal que se cumplan las restricciones estequiométricas de las reacciones bioquímicas del modelo,
b) simulación de la distribución isotópica de los metabolitos en la red bioquímica, la cual debe estar de acuerdo con las restricciones estequiométricas mencionadas en a),
c) comparación del patrón de marcación isotópica y los valores de flujo obtenidos durante la simulación con los valores experimentales obtenidos por CG-EM,
d) repetición iterativa del procedimiento a través de un algoritmo de optimización apropiado (42), de modo tal que la diferencia entre la distribución in silico y la distribución experimental sea mínima, y
e) análisis estadístico de los resultados, en virtud de que la propagación de errores de los parámetros biológicos observables (e.g., distribución isotópica, flujos extracelulares, evolución de CO2, etc.) al mapa de flujos puede originar resultados precisos pero estadísticamente erróneos o inválidos (37).
Fig. 2. Esquema del procedimiento para la obtención del mapa de flujos metabólicos a partir de experimentos de marcación con carbono seguidos de integración in silico de los datos experimentales. En cada iteración, el patrón espectroscópico de fragmentos de isotopómeros generado a partir de un conjunto de flujos cuyos valores se asignan arbitrariamente se compara con los valores experimentales. La secuencia de iteraciones se repite hasta minimizar la diferencia entre ambos conjuntos de valores. CG-EM, cromatografía de gases acoplada a espectrometría de masa.
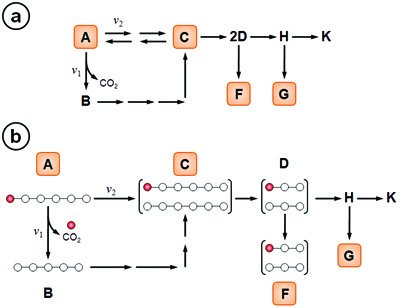
Para ilustrar la importancia de la determinación de flujos metabólicos por marcación con sustratos isotópicos, consideremos la secuencia metabólica sencilla que muestra la Fig. 3a. En esta red, el metabolito A es utilizado como sustrato por dos vías enzimáticas que operan con velocidades netas v1 y v2. Si pudiese medirse la velocidad de consumo de A y la velocidad de secreción de F y G (asumiendo que estos metabolitos fuesen excretados al medio extracelular), se podría analizar la distribución de flujos en los puntos de ramificación a nivel de los metabolitos D y H, y se podría derivar matemáticamente la velocidad de síntesis del metabolito K. Sin embargo, no sería posible estudiar la distribución de flujos en el ciclo metabólico A → B → C → A (esto es: no se pueden conocer los valores de v1 y v2). Por otra parte, si la reacción que convierte a C en D consta de más de un paso enzimático, la distribución de intermediarios de la vía no podría conocerse y necesariamente se debería simplificar la secuencia de reacciones a la forma C → 2D. Finalmente, y tal como lo ilustra la Fig. 3a, la conversión de A en C es reversible y no podría deducirse el grado de reversibilidad de la reacción a partir de AFM estequiométrico.
Fig. 3. Ejemplo hipotético de una red bioquímica sencilla incluyendo rutas enzimáticas lineales y ramificadas. a) La determinación de la velocidad de consumo del metabolito A y las velocidades de síntesis de los metabolitos F y G permite estimar los flujos para los metabolitos D, H, y K; pero no distingue los flujos en el ciclo metabólico caracterizado por v1 y v2. b) En un estudio de AFM basado en marcación isotópica el enriquecimiento posicional en el metabolito C permite distinguir si éste provino de A o B, y consecuentemente puede estimarse la contribución relativa de v1 y v2 a la velocidad de síntesis de C. Una situación análoga se verifica para los metabolitos D y F.
Consideremos ahora el estudio de la misma red bioquímica utilizando un sustrato isotópicamente marcado (Fig. 3b). En este caso, puede observarse que las reacciones bioquímicas en la red generan asimetrías en la distribución de átomos de carbono. Dichas asimetrías pueden deducirse del análisis de isotopómeros y, por lo tanto, los metabolitos intermediarios pueden distinguirse unos de otros incluso cuando conduzcan a la formación de un único metabolito final. En el ejemplo de la figura, el metabolito A es una molécula de seis átomos de carbono cuya primera posición está ocupada por un átomo de 13C. La primera reacción, representada por el flujo v1, consiste en una decarboxilación que origina una molécula del metabolito B sin marca isotópica. De esta manera, el patrón de marcación de la molécula C dependerá de si ésta se originó a partir de A (a través del flujo v1, en cuyo caso se obtiene una molécula marcada) o a partir de B (a través del flujo v2, en cuyo caso la molécula no contiene isótopos). De esta manera, el enriquecimiento isotópico del metabolito C (es decir, la cantidad de moléculas marcadas respecto de las que no contienen átomos de 13C) será directamente proporcional al cociente v2/v1. Esta relación también es cierta para los metabolitos D y F. El grado de enriquecimiento en 13C en la primera posición del metabolito F (el cual, como se dijo, puede medirse en el medio extracelular) provee información acerca de la velocidad relativa del flujo v2 y, en conjunción con otras mediciones de flujos y análisis estequiométrico, permite distinguir entre v1 y v2. Aunque arbitrario, este ejemplo esquemático guarda similitud con el catabolismo de una hexosa a través de la vía glicolítica de Embden-Meyerhof-Parnas (que, en el ejemplo, estaría asociada al flujo caracterizado por v2) y la rama oxidativa de la vía de pentosas fosfato (en el ejemplo, representada por el flujo v1).
Aplicación del análisis de flujos metabólicos en la selección y construcción de fenotipos de relevancia en Biotecnología
Los enfoques clásicos para la obtención de fenotipos industriales han evolucionado constantemente hacia la utilización de estrategias más sistemáticas. Tradicionalmente, el fenotipo de los microorganismos utilizados en bioprocesos industriales fue consecuencia de un programa de desarrollo que típicamente incluye varios ciclos de mutagénesis al azar y selección, lo cual lógicamente trae aparejados cambios a nivel molecular y celular que no pueden ser predichos con exactitud (29). De hecho, en la mayoría de los casos se desconoce la naturaleza de las mutaciones introducidas. Con el advenimiento de las metodologías y técnicas de Ingeniería Metabólica, a través del conocimiento de la arquitectura genómica, las vías metabólicas pudieron ser convenientemente amplificadas, suprimidas, o moduladas basándose en la información derivada de la red metabólica del microorganismo (16, 35). A pesar de que se trata de un proceso sistemático, este tipo de enfoque normalmente está limitado a modificaciones ‘locales’ más que a la comprensión global del metabolismo microbiano. Si bien el nivel de expresión de ciertos genes y la concentración intracelular de proteínas y metabolitos proveen información importante acerca del comportamiento de la red metabólica en estudio, existen limitaciones inherentes a esta metodología para la descripción del fenotipo celular debido a la falta de información que integre estos parámetros y los correlacione para una comprensión holística del metabolismo. Además, en la mayoría de los casos se ignora el bioproceso in toto, es decir, desde el diseño, construcción, y mejora de la cepa hasta el cultivo de la misma en un biorreactor.
Según se mencionó anteriormente, los flujos metabólicos pueden considerarse como una forma de representar el fenotipo de una célula como resultado de la interacción entre varios componentes celulares; los flujos observados reflejan las consecuencias conjuntas de los procesos de transcripción, traducción, y actividad enzimática, además de los (a menudo complejos) procesos regulatorios que los controlan. En otras palabras, se considera el sistema como un todo, y no sus partes constituyentes en forma individual. En forma aislada, cada flujo metabólico provee el grado de participación de la enzima o vía metabólica en estudio en funciones celulares y procesos metabólicos. De esta manera, en un ensayo de AFM se cuenta con información de los componentes individuales del metabolismo, pero dicha información puede ser integrada para la comprensión del metaboloma (ejemplo: el conjunto de flujos metabólicos del microorganismo en una condición determinada). Consecuentemente, la cuantificación de estos flujos in vivo es de especial importancia en el contexto de la fisiología celular para implementar estrategias efectivas de Ingeniería Metabólica (6), disciplina para la cual uno de los objetivos principales consiste en maximizar la conversión de un sustrato en un determinado producto (35). En la actualidad, el tiempo necesario para implementar un AFM completo disminuyó considerablemente gracias al desarrollo de metodologías de medición isotópica novedosas así como a la utilización de plataformas informáticas potentes.
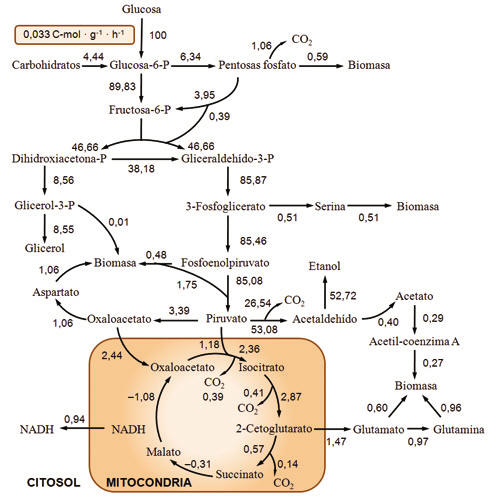
Como ejemplo de aplicación, en la Fig. 4 se muestra un mapa de flujos metabólicos obtenido para la levadura Saccharomyces cerevisiae en condiciones anaeróbicas de cultivo. La información provista por este análisis sugiere que el principal destino de la glucosa utilizada como sustrato carbonado es la decarboxilación reductiva que transforma el piruvato en acetaldehído y posteriormente en etanol. El diseño de cepas etanologénicas de S. cerevisiae hizo uso extensivo de la información provista por este AFM, ya que permitió el re-direccionamiento racional de algunas vías competitivas a la síntesis de etanol (viz., generación de precursores de biomasa) hacia la obtención del producto de interés, aumentando considerablemente el rendimiento de producto en sustrato (39).
Fig. 4. Representación gráfica de los resultados de un ensayo de 13C-AFM realizado en cultivos anaeróbicos de Saccharomyces cerevisiae. El sustrato carbonado utilizado fue una mezcla de 13CU-glucosa (uniformemente marcada), 13C1-glucosa (marcada en la posición C1), y glucosa no marcada. Para simplificar la visualización de los resultados, en muchos casos los flujos se representan como una fracción de la velocidad de consumo de sustrato (en este ejemplo, 0,033 C-mol de glucosa · g biomasa-1 · h-1). Para ello, se asigna arbitrariamente a este parámetro el valor de 100 y los flujos restantes toman valores porcentuales relativos respecto al consumo de sustrato. Obsérvese que el AFM permite explicitar la compartimentalización de las reacciones bioquímicas consideradas. Datos tomados de Nissen et al. (28).
En los últimos años se ha empleado el AFM como herramienta para el diseño y análisis de fenotipos industriales con frecuencia cada vez mayor y, de hecho, se ha sugerido que virtualmente cualquier metabolito en el metabolismo microbiano puede ser producido en forma eficiente si se manipulan los flujos metabólicos centrales en la forma adecuada. De acuerdo con este objetivo general, pueden distinguirse dos tipos de enfoque:
a) estudios en los cuales el AFM se utiliza para estudiar el metabolismo celular en condiciones compatibles con un bioproceso para el desarrollo de estrategias adecuadas para el crecimiento del microorganismo y producción de metabolitos de interés biotecnológico, y
b) utilización de AFM, llevados a cabo en condiciones estándar de cultivo, para identificar vías metabólicas cuya manipulación podría resultar ventajosa para la obtención de un fenotipo industrial.
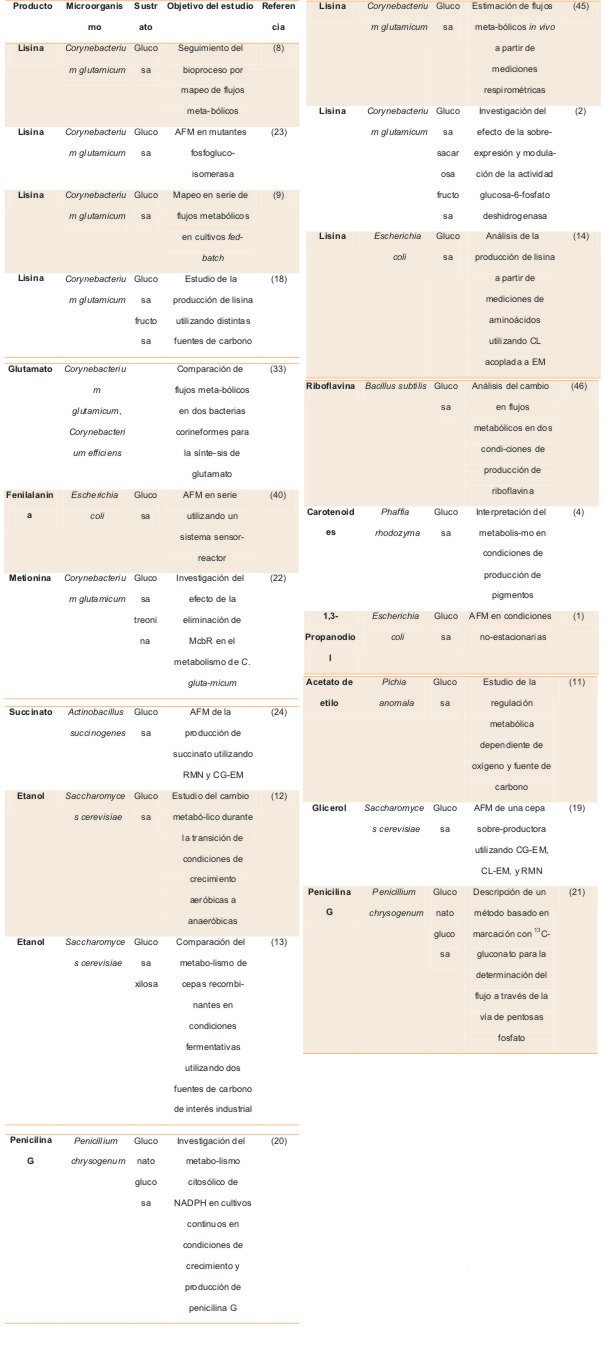
Algunos de los ejemplos más relevantes disponibles en la lbibliografía se muestran en la Tabla 1. Estos ejemplos corresponden al primero de los dos tipos de enfoque recién discutidos. Al mismo tiempo, existen numerosos ejemplos dentro de la segunda categoría de ensayos para AFM, en particular en relación a la manipulación de reguladores globales de la expresión génica en E. coli como herramienta relevante en Biotecnología (27, 30, 47).
Tabla 1. Algunos ejemplos de la aplicación de AFMa para la evaluación de fenotipos microbianos asociados a bioprocesos de interés industrial.
a Las abreviaturas utilizadas son: AFM, análisis de flujos metabólicos; CL, cromatografía líquida; EM, espectrometría de masas; RMN, resonancia magnética nuclear.
Conclusión y perspectivas
En un futuro, probablemente cercano, la integración de las tecnologías disponibles para el análisis del comportamiento metabólico, incluyendo 13C-AFM, permitirá la comprensión de la dinámica celular in vivo y conducirán al desarrollo eficaz y racional de bioprocesos industriales. En la actualidad estas metodologías están comenzando a ser utilizadas durante el desarrollo de procesos biotecnológicos con frecuencia cada vez mayor, pero aún así se recurre usualmente a las técnicas clásicas de mejora y selección para la mayoría de los protocolos de obtención de fenotipos industriales. Un desarrollo prometedor, ligado al advenimiento de metodologías de detección de metabolitos de alta sensibilidad y herramientas informáticas poderosas, es el AFM en tiempo real; esto es, durante el bioproceso. La elucidación de la distribución de flujos metabólicos in situ permitiría tomar acciones correctivas del bioproceso e indudablemente conduciría a una notable mejora del mismo. Por ejemplo, Soga (34) ha descripto la utilización de electroforesis capilar acoplada a EM para la detección simultánea y en tiempo real de más de 1000 metabolitos cargados. Otros aspectos en los cuales la introducción de mejoras en el análisis del metaboloma permitiría una mejor comprensión y/o el desarrollo efectivo de bioprocesos son:
a) AFM en células eucariotas (libres y en tejidos). Debido a la compartimentalización inherente a este tipo de células, el análisis de isotopómeros e integración matemática de los resultados se vuelven considerablemente más complejos. Por ejemplo, el oxaloacetato es un metabolito que se encuentra simultáneamente en la matriz mitocondrial y en el citosol en concentraciones distintas, y el patrón de marcación isotópica como única herramienta de estudio no permite discernir la localización sub-celular de este metabolito. Asimismo, en un cultivo de células de mamífero, las velocidades de crecimiento son tan bajas que la cuantificación de flujos metabólicos posee asociada importantes errores.
b) estudios poblacionales mediante AFM. Es un hecho conocido y ampliamente descripto que aún en un cultivo continuo que ha alcanzado condiciones de equilibrio dinámico existe más de una población metabólicamente identificable. En la mayoría de los casos, la existencia de sub-poblaciones no altera significativamente los resultados del análisis, pero si el equilibrio poblacional se relaciona con un fenotipo de interés industrial (e.g., pérdida de la capacidad de sintetizar un metabolito, o enriquecimiento del cultivo en células con mutaciones adaptativas, etc.) sería particularmente necesario distinguir entre las poblaciones presentes en el cultivo. Esta distinción requiere del desarrollo de técnicas que permitan el estudio de flujos metabólicos a nivel de células aisladas.
c) medición de metabolitos intracelulares y estimación de flujos independiente del análisis retrobiosintético, en el cual se basa la metodología de AFM. Como se mencionó anteriormente, los flujos metabólicos en un ensayo de 13C-AFM se deducen a partir de la presencia y abundancia de metabolitos marcados utilizando metodologías basadas en simulaciones in silico. Esto, por definición, implica la existencia de errores asociados al método de análisis, cuya magnitud podría ser reducida si la concentración intracelular de al menos algunos metabolitos en la red bioquímica pudiese ser directamente determinada in vivo sin alterar el metabolismo celular.
Por lo tanto, puede concluirse que la implementación de 13C-AFM como herramienta de análisis y control de bioprocesos se encuentra actualmente en un estadío temprano de desarrollo y es altamente plausible de ser mejorada y adaptada a los requerimientos de cada proceso en particular.
Agradecimientos
Agradezco muy especialmente a mis directores, Dr. Miguel A. Galvagno y Dra. Beatriz S. Méndez, por su constante apoyo y estimulación. Asimismo, agradezco al Dr. George N. Bennett, Dr. Jiangfeng Zhu, y Dr. Ka-Yiu San (Departamento de Bioquímica y Biología Celular, y Departamento de Bioingeniería; Rice University, Houston, Texas) por haberme ofrecido la posibilidad de trabajar en un laboratorio líder en el análisis de flujos metabólicos; y al Consejo de Investigaciones Científicas y Técnicas (Argentina) y la American Society for Microbiology (EE.UU.) por el apoyo económico brindado durante mi estadía en Rice University.
Referencias bibliográficas
1. Antoniewicz MR, Kraynie DF, Laffend LA, Gonzalez-Lergier J, Kelleher JK, Stephanopoulos G, 2007. Metabolic flux analysis in a non-stationary system: fed-batch fermentation of a high yielding strain of E. coli producing 1,3-propanediol. Metab. Eng. 9: 277-292.
2. Becker J, Klopprogge C, Herold A, Zelder O, Bolten CJ, Wittmann C, 2007. Metabolic flux engineering of L-lysine production in Corynebacterium glutamicum – Over expression and modification of G6P dehydrogenase. J. Biotechnol. 132: 99-109.
3. Blum JJ, Stein RB. On the analysis of metabolic networks. Biological regulation and development, Goldberger RF (ed.), Plenum Press, New York, 1982, 99-124.
4. Cannizzaro C, Christensen B, Nielsen J, von Stockar U, 2004. Metabolic network analysis on Phaffia rhodozyma yeast using 13C-labeled glucose and gas chromatography-mass spectrometry. Metab. Eng. 6: 340-351.
5. Christensen B, Nielsen J, 1999. Isotopomer analysis using GC-MS. Metab. Eng. 1: 282-290.
6. Christensen B, Nielsen J, 1999. Metabolic network analysis, a powerful tool in metabolic engineering. Adv. Biochem. Eng. Biotechnol. 66: 209-231.
7. Dauner M, Sauer U, 2001. GC-MS analysis of amino acids rapidly provides rich information for isotopomer balancing. Biotechnol. Prog. 76: 642-649.
8. Drysch A, El Massaoudi M, Mack C, Takors R, de Graaf AA, Sahm H, 2003. Production process monitoring by serial mapping of microbial carbon flux distributions using a novel sensor reactor approach: II. 13C-labeling-based metabolic flux analysis and L-lysine production. Metab. Eng. 5: 96-107.
9. Drysch A, El Massaoudi M, Wiechert W, de Graaf AA, Takors R, 2004. Serial flux mapping of Corynebacterium glutamicum during fed-batch L-lysine production using the sensor reactor approach. Biotechnol. Bioeng. 85: 497-505.
10. Fischer E, Sauer U, 2003. Metabolic flux profiling of Escherichia coli mutants in central carbon metabolism using GC-MS. Eur. J. Biochem. 270: 880-891.
11. Fredlund E, Blank LM, Schnurer J, Sauer U, Passoth V, 2004. Oxygen- and glucose-dependent regulation of central carbon metabolism in Pichia anomala. Appl. Environ. Microbiol. 70: 5905-5911.
12. Frick O, Wittmann C, 2005. Characterization of the metabolic shift between oxidative and fermentative growth in Saccharomyces cerevisiae by comparative 13C flux analysis. Microb. Cell Fact. 4: 30.
13. Grotkjaer T, Christakopoulos P, Nielsen J, Olsson L, 2005. Comparative metabolic network analysis of two xylose fermenting recombinant Saccharomyces cerevisiae strains. Metab. Eng. 7: 437-444.
14. Iwatani S, van Dien S, Shimbo K, Kubota K, Kageyama N, Iwahata D, Miyano H, Hirayama K, Usuda Y, Shimizu K, Matsui K, 2007. Determination of metabolic flux changes during fed-batch cultivation from measurements of intracellular amino acids by LC-MS/MS. J. Biotechnol. 128: 93-111.
15. Jetten MSM, Sinskey AJ, 1995. Recent advances in the physiology and genetics of amino acid-producing bacteria. Crit. Rev. Biotechnol. 15: 73.
16. Kacser H, Acerenza L, 1993. A universal method for achieving increases in metabolite production. Eur. J. Biochem. 216: 361-367.
17. Kelleher JK, 2001. Flux estimation using isotopic tracers: common ground for metabolic physiology and metabolic engineering. Metab. Eng. 3: 100-110.
18. Kiefer P, Heinzle E, Zelder O, Wittmann C, 2004. Comparative metabolic flux analysis of lysine-producing Corynebacterium glutamicum cultured on glucose or fructose. Appl. Environ. Microbiol. 70: 229-239.
19. Kleijn RJ, Geertman JM, Nfor BK, Ras C, Schipper D, Pronk JT, Heijnen JJ, van Maris AJ, van Winden WA, 2007. Metabolic flux analysis of a glycerol-overproducing Saccharomyces cerevisiae strain based on GC-MS, LC-MS and NMR-derived 13C-labelling data. FEMS Yeast Res. 7: 216-231.
20. Kleijn RJ, Liu F, van Winden WA, van Gulik WM, Ras C, Heijnen JJ, 2007. Cytosolic NADPH metabolism in penicillin-G producing and non-producing chemostat cultures of Penicillum chrysogenum. Metab. Eng. 9: 112-123.
21. Kleijn RJ, van Winden WA, Ras C, van Gulik WM, Schipper D, Heijnen JJ, 2006. 13C-labeled gluconate tracing as a direct and accurate method for determining the pentose phosphate pathway split ratio in Penicillium chrysogenum. Appl. Environ. Microbiol. 72: 4743-4754.
22. Kromer JO, Heinzle E, Schroder H, Wittmann C, 2006. Accumulation of homolanthionine and activation of a novel pathway for isoleucine biosynthesis in Corynebacterium glutamicum McbR deletion strains. J. Bacteriol. 188: 609-618.
23. Marx A, Hans S, Mockel B, Bathe B, de Graaf AA, McCormack AC, Stapleton C, Burke K, O'Donohue M, Dunican LK, 2003. Metabolic phenotype of phosphoglucose isomerase mutants of Corynebacterium glutamicum. J. Biotechnol. 104: 185-197.
24. McKinlay JB, Shachar-Hill Y, Zeikus JG, Vieille C, 2007. Determining Actinobacillus succinogenes metabolic pathways and fluxes by NMR and GC-MS analyses of 13C-labeled metabolic product isotopomers. Metab. Eng. 9: 177-192.
25. Neidhardt FC, Ingraham JL, Schaechter M, Physiology of the bacterial cell: a molecular approach. Sunderland, MA, Sinauer Associates, 1990.
26. Neijssel OM, Teixeira de Mattos MJ, 1994. The energetics of bacterial growth: a reassessment. Mol. Microbiol. 13: 179-182.
27. Nikel PI, Zhu J, San KY, Méndez BS, Bennett GN, 2009. Metabolic flux analysis of Escherichia coli creB and arcA mutants reveals shared control of carbon catabolism under microaerobic growth conditions. J. Bacteriol. 191: 5538-5548
28. Nissen TL, Schulze U, Nielsen J, Villadsen J, 1997. Flux distributions in anaerobic, glucose-limited continuous cultures of Saccharomyces cerevisiae. Microbiology 143: 203-218.
29. Park JH, Lee SY, Kim TY, Kim HU, 2008. Application of systems biology for bioprocess development. Trends Biotechnol. 26: 404-412.
30. Perrenoud A, Sauer U, 2005. Impact of global transcriptional regulation by ArcA, ArcB, Cra, Crp, Cya, Fnr, and Mlc on glucose catabolism in Escherichia coli. J. Bacteriol. 187: 3171-3179.
31. Sauer U, Hatzimanikatis V, Bailey JE, Hochuli M, Szyperski T, Wüthrich K, 1997. Metabolic fluxes in riboflavin-producing Bacillus subtilis. Nat. Biotechnol. 15: 448-452.
32. Schmidt K, Carlsen M, Nielsen J, Villadsen J, 1997. Modelling isotopomer distribution in biochemical networks using isotopomer mapping matrices. Biotechnol. Bioeng. 55: 831-840.
33. Shirai T, Nakato A, Izutani N, Nagahisa K, Shioya S, Kimura E, Kawarabayasi Y, Yamagishi A, Gojobori T, Shimizu H, 2005. Comparative study of flux redistribution of metabolic pathway in glutamate production by two coryneform bacteria. Metab. Eng. 7: 59-69.
34. Soga T, 2007. Capillary electrophoresis-mass spectrometry for metabolomics. Methods Mol. Biol. 358: 129-137.
35. Stephanopoulos G, Aristidou AA, Nielsen J, Metabolic engineering: principles and methodologies. San Diego, CA, Academic Press, 1998.
36. Vallino JJ, Stephanopoulos G, 1993. Metabolic flux distribution in Corynebacterium glutamicum during growth and lysine overproduction. Biotechnol. Bioeng. 41: 633-646.
37. van Winden W, Verheijen P, Heijnen S, 2001. Possible pitfalls of flux calculations based on 13C-labeling. Metab. Eng. 3: 151-162.
38. Varma A, Palsson BO, 1994. Metabolic flux balancing: basic concepts, scientific and practical use. Bio/Technology 12: 994-998.
39. Vertès AA, Inui M, Yukawa H, 2008. Technological options for biological fuel ethanol. J. Mol. Microbiol. Biotechnol. 15: 16-30.
40. Wahl A, El Massaoudi M, Schipper D, Wiechert W, Takors R, 2004. Serial 13C-based flux analysis of an L-phenylalanine-producing E. coli strain using the sensor reactor. Biotechnol. Prog. 20: 706-714.
41. Wiechert W, 2001. 13C Metabolic flux analysis. Metab. Eng. 3: 195-206.
42. Wiechert W, Möllney M, Isermann N, Wurzel M, de Graaf AA, 1999. Bidirectional reaction steps in metabolic networks. Part III: Explicit solution and analysis of isotopomer labeling systems. Biotechnol. Bioeng. 66: 69-85.
43. Wiechert W, Möllney M, Petersen S, de Graaf AA, 2001. A universal framework for 13C metabolic flux analysis. Metab. Eng. 3: 265-283.
44. Wittmann C, Heinzle E, 1999. Mass spectrometry for metabolic flux analysis. Biotechnol. Bioeng. 62: 739-750.
45. Yang TH, Wittmann C, Heinzle E, 2006. Respirometric 13C flux analysis. Part II: In vivo flux estimation of lysine-producing Corynebacterium glutamicum. Metab. Eng. 8: 432-446.
46. Zamboni N, Fischer E, Muffler A, Wyss M, Hohmann HP, Sauer U, 2005. Transient expression and flux changes during a shift from high to low riboflavin production in continuous cultures of Bacillus subtilis. Biotechnol. Bioeng. 89: 219-232.
47. Zhu J, Shalel-Levanon S, Bennett GN, San KY, 2006. Effect of the global redox sensing/regulation networks on Escherichia coli and metabolic flux distribution based on C-13 labeling experiments. Metab. Eng. 8: 619-627.
ISSN 1666-7948
www.quimicaviva.qb.fcen.uba.arRevista QuímicaViva
Número 2, año 8, Agosto 2009
quimicaviva@qb.fcen.uba.ar