Proliferación o quiescencia: una difícil decisión celular
Eduardo T. Cánepa *
Laboratorio de Biología Molecular, Departamento de Química Biológica.
Facultad de Ciencias Exactas y Naturales. Universidad de Buenos Aires.
Ciudad Universitaria Pabellón II piso 4. 1428 Ciudad de Buenos Aires. Argentina
ecanepa@qb.fcen.uba.ar
Recibido el 05/04/07. Aceptado el 19/04/07.
Resumen
El ciclo celular consiste en una serie de eventos precisamente coordinados que permiten el crecimiento y la proliferación celular. Para asegurar la correcta progresión a través del ciclo, las células han desarrollado una serie de puntos de control que previenen la entrada en una nueva fase del ciclo hasta que hayan completado exitosamente la anterior. Los principales componentes de la maquinaria del ciclo celular son las quinasas dependientes de ciclinas (CDK), las que, cuando son activadas, permiten a las células pasar de una fase del ciclo a la siguiente. Estas CDK son reguladas positivamente por las ciclinas y negativamente por las proteínas celulares denominadas inhibidores de las CDK (CKI). El cáncer representa una desregulación del ciclo celular y las células tumorales sufren una proliferación descontrolada. Dado que los tumores se originan, a menudo, en tejidos adultos en los que la mayoría de las células se encuentran en estado quiescente, la ventaja proliferativa que presentan las células tumorales se basa en la habilidad de sortear la quiescencia. El conocimiento de los mecanismos moleculares que controlan este balance entre los estados proliferativo y quiescente, permitirán aclarar el modo por el cual las células normales se convierten en tumorigénicas y el diseño de nuevas estrategias antitumorales.
Palabras clave: ciclo celular – cáncer – proliferación – quiescencia – CDK - ciclinasProliferation or quiescence: a critical cellular decision
Abstract
The cell cycle represents a series of tightly integrated events that allow the cell to grow and proliferate. To ensure proper progression through the cell cycle, cells have developed a series of checkpoints that prevent them from entering into a new phase until they have successfully completed the previous one. Critical parts of the cell cycle machinery are the cyclin-dependent kinases (CDK), which, when activated provide a means for the cell to move from one phase of the cell cycle to the next. The CDKs are regulated positively by cyclins and regulated negatively by naturally occurring CDK inhibitors (CKI). Cancer represents a dysregulation of the cell cycle and tumor cells undergo uncontrolled proliferation. Since tumors most often originate from adult tissues, in which most cells are quiescent, the proliferative advantage of tumor cells arise from their ability to bypass quiescence. Understanding the molecular mechanisms that control this balance between the proliferative and quiescent states, should provide insights into how normal cells become tumorigenic and to design new cancer strategies.
Key words: cell cycle – cancer – proliferation – quiescence – CDK - cyclinsIntroducción
La proliferación celular implica la división mitótica de una célula, luego de la duplicación de su material genético, para dar origen a dos células que, normalmente, son idénticas a la progenitora. El aumento en el número de células de un determinado tejido es inherente a la proliferación. Por el contrario, en el estado de quiescencia, la célula cumple con todas sus funciones específicas, al igual que en el caso anterior, pero sin duplicar su contenido genómico ni dividirse. A estos dos estados diferentes los une, inevitablemente, una de las más cruciales decisiones que toda célula de los metazoos debe adoptar, decisión que, erróneamente tomada, puede causar daños irreversibles y fatales para todo el organismo. Es así que una célula proliferante debe optar entre continuar en el ciclo celular dando otra ronda de división o, por el contrario, quedar en estado de quiescencia. De igual modo, las células quiescentes deben decidir si continuar en un estado no proliferativo o ingresar al ciclo celular y dividirse. Durante el desarrollo temprano, muy pocas células abandonan el ciclo celular, pero en el estado adulto, la mayoría de ellas se encuentran en estado quiescente. Sólo células especializadas, tales como las del sistema hematopoyético, el epitelio intestinal o hepáticas, mantienen una proliferación activa en el organismo adulto. Frente al hecho, entonces, que todas las células proliferantes tienen la capacidad de entrar en quiescencia y, por otro lado, todas las células quiescentes, salvo aquellas que han alcanzado una diferenciación terminal, pueden reingresar al ciclo de división mitótica, surge el interrogante acerca de cuales son los mecanismos que controlan esta verdadera toma de decisiones celular. Y más inquietante aún, ¿cuáles son las consecuencias asociadas a una desregulación de estos mecanismos? (Hahn and Weinberg, 2002; Hanahan and Weinberg, 2000; Malumbres and Barbacid, 2001). Las células cancerosas muestran, a menudo, alteraciones en los caminos de transducción de señales asociados a la proliferación en respuesta a estímulos externos. De hecho, muchos factores de crecimiento y sus receptores, así como sus intermediarios y efectores localizados en membrana, citoplasma o núcleo han sido identificados como oncogenes o como supresores tumorales. Además de estos, otros genes mutados en cánceres incluyen aquellos relacionados con la inactivación de la apoptosis, la inducción de la inestabilidad genómica o la promoción de la angiogénesis. La mutación en estas moléculas resulta en una alteración de los mecanismos regulatorios que controlan el ciclo celular (Ben-Porath and Weinberg, 2005; Blume-Jensen and Hunter, 2001; Sherr, 2004) .
Ciclo básico
El ciclo celular se divide en cuatro fases. Durante dos de ellas las células ejecutan los eventos fundamentales de la división celular: la generación de una copia fiel de su material genético (la fase de síntesis o S) y la partición de todos los componentes celulares entre las dos células hijas (mitosis o fase M). Las otras dos fases del ciclo, G1 y G2, representan períodos de espera (del inglés “gap”) durante los cuales las células se preparan para completar con éxito las fases S y M, respectivamente. Cuando las células cesan de proliferar, ya sea debido a señales antimitogénicas o por la ausencia de señales mitogénicas, egresan del ciclo y entran en el estado de no división quiescente, ya mencionado, y que se conoce como G0 (Lukas and Bartek, 2004). De modo de asegurar la progresión a través del ciclo, las células han desarrollado una serie de puntos de control que evitan el pasaje a una nueva fase del ciclo antes que la anterior haya sido completada exitosamente (Kastan and Bartek, 2004; Lukas et al., 2004; Martin, 2003). Es probable, también, que las células quiescentes deban pasar ciertos puntos de control antes que puedan entrar al ciclo. Por ejemplo, las células deben asegurarse que hayan llegado a su tamaño homeostático y adquirido la masa proteica necesaria ya que, de lo contrario, serán cada vez más pequeñas con cada ronda del ciclo celular. Los metazoos, además, deben controlar el número de células de cada órgano lo cual, junto con el tamaño celular, determinará el tamaño del organismo. Este punto, el crecimiento celular, es minimizado frecuentemente cuando se habla de proliferación. En realidad, ambos eventos deben estar perfectamente coordinados. Solamente en algunos sistemas biológicos, tales como los oocitos, las neuronas o las células musculares, donde puede haber crecimiento sin división, o en los huevos fertilizados, donde ocurre la división celular en ausencia de crecimiento, estos procesos funcionan en forma independiente. En la mayoría de las células, sin embargo, la división celular debe estar acompañada de un crecimiento acorde, de lo contrario, se obtendrán células cada vez más pequeñas lo que puede afectar su viabilidad (Radimerski et al., 2002; Wullschleger et al., 2006). Los elementos reguladores que controlan estos parámetros no son muy conocidos aunque, con toda seguridad, deben ser influenciados por el medio exterior, ya sea la cantidad de nutrientes disponibles o la información mitogénica recibida en un momento determinado. Evidentemente, el mantener un adecuado equilibrio entre estos dos parámetros es esencial para una correcta división celular. Si las células presentan requerimientos mitogénicos demasiado estrictos, pueden ser incapaces de proliferar cuando sea necesario, por ejemplo en el caso de heridas o durante una infección. Por el contrario, el relajamiento de los requerimientos mitogénicos puede llevar a una proliferación descontrolada con el consiguiente peligro de crecimiento neoplásico. Las células en cultivo tienen un período durante el cual dependen de la presencia de mitógenos para entrar y continuar en el ciclo, más allá del cual se independizan de ellos. Esta transición se conoce como punto de restricción o punto R y representa un punto de no retorno que compromete a las células a una nueva división celular (Stein et al., 2006).
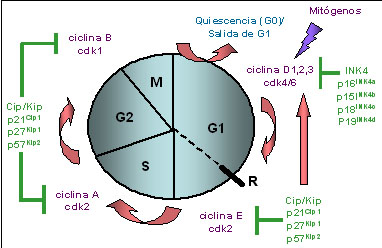
Figura 1. El ciclo celular. El esquema muestra las distintas etapas del ciclo y las proteínas y factores que regulan la transición entre cada una de ellas.
Moléculas Quinasas dependientes de ciclinas
Las moléculas centrales en el mecanismo de control del ciclo celular (Figura 1) son las quinasas dependientes de ciclinas (CDKs). Estas constituyen un grupo de serina/treonina quinasas que forman complejos heterodiméricos activos al unirse a sus subunidades regulatorias, las ciclinas. Varias CDKs, principalmente CDK4, CDK6, CDK2 y posiblemente CDK3, actúan en forma cooperativa para conducir las células hacia la fase S, a través de la fase G1. Las CDK4 y CDK6 están involucradas en el pasaje durante G1 temprano, mientras que CDK2 es requerida para completar G1 e iniciar la fase S. Las primeras forman complejos activos sólo con las ciclinas de tipo D (D1, D2 y D3) y están, desde un punto de vista estructural, estrechamente relacionados y son funcionalmente indiferenciables. Por su parte, la CDK2 es activada secuencialmente por las ciclinas de tipo E (E1 y E2), durante la transición entre las fases G1 y S, y por las ciclinas A (A1 y A2) durante la fase S. Todavía no se conoce el papel desempeñado por la CDK3 y debido a sus bajos niveles de expresión y a su estado inactivo en una gran variedad de tejidos, su participación en el ciclo celular sería prescindible (Murray, 2004; Sandal, 2002) . Ciclinas D Las ciclinas de tipo D desempeñan un papel importante en la integración de las señales mitogénicas y, se puede decir, que constituyen el punto de conexión entre el medio extracelular y el ciclo celular. Una vez que los factores de crecimiento interaccionan con sus receptores localizados en la membrana plasmática, se dispara una cascada de transducción de señales cuyo eje principal es la vía Ras/Raf/MAPK, uno de cuyos principales lo constituye la síntesis de ciclina D1. Esta ciclina D1, cuya interacción con CDK4/6 permite la entrada de la célula en la fase G1 del ciclo celular, es, a su vez, una molécula muy inestable. La ciclina D1 es blanco de la ubiquitinación por la ligasa SCF y, luego de su transporte desde el núcleo al citoplasma, degradada por el sistema de proteosoma. Esta exportación nuclear, luego de la ubiquitinación, es mediada por la enzima glucógeno sintetasa quinasa 3-β (GSK3), una quinasa que es inhibida por la vía de señalización Ras/PI3K/AKT. De este modo, la disponibilidad de ciclina D1 es controlada por un balance entre su síntesis, dependiente de Ras/Raf/MAPK y su estabilidad, dependiente de Ras/PI3K/AKT, por un lado y la actividad de GSK3 y SCF, por el otro. Sin embargo, recientemente se ha visto que las células que carecen de ciclina D1 pueden proliferar, indicando que esta molécula no sería estrictamente necesaria para la entrada en fase G1. Como las tres ciclinas D presentan una homología muy alta y son coexpresadas en la mayoría de los tejidos, es posible que sus funciones tengan redundancia (Lutzen et al., 2004; Murray, 2004; Ortega et al., 2002). Reguladores de CDK Se deduce que, regulando la actividad de las CDK, es posible controlar la entrada de las células al ciclo celular. Como ya se mencionó, las CDK pueden ser reguladas positivamente a través de su unión con las ciclinas, pero también negativamente por medio de su interacción con los inhibidores de CDK (CKI) (Sherr and Roberts, 1999). La actividad de las CDK es regulada también por fosforilación; éstas deben ser fosforiladas en un residuo treonina (T172 en CDK4 y T160 en CDK2) que causa una modificación en la conformación de la proteína y un aumento en la exposición del sitio catalítico. Esta modificación es llevada a cabo por el complejo CDK7/ciclina H, también conocida como CAK (quinasa activadora de CDK). También hay fosforilaciones inhibitorias, como las que se realizan en la CDK1 sobre los residuos T14 y Y15 y que son catalizadas por quinasas específicas como las Wee1 y MYT1. Esta inhibición es anulada cuando las fosfatasas CDC25A y CDC25B defosforilan dichos residuos (Donzelli and Draetta, 2003; Essers et al., 2005). Los CKI se dividen dos familias, cada una de ellas constituída por varios miembros, de acuerdo a las CDK que inhiben. La familia INK4 consta de cuatro proteínas, p16INK4a, p15INK4b, p18INK4c y p19INK4d, que ejercen su acción inhibitoria sobre las CDK4 y CDK6, uniéndose a ellas e impidiendo su asociación con las ciclinas de tipo D. Estas proteínas son estructuralmente muy relacionadas e igualmente potentes como inhibidores por lo que sus funciones aparentarían cierta redundancia. Recientemente, sin embargo, se han descripto funciones diferentes en algunas de ellas relacionadas con diversos procesos metabólicos como la reparación del DNA, senescencia y diferenciación (Cunningham and Roussel, 2001; Roussel, 1999). La familia restante, la de los inhibidores Cip/Kip, la constituyen tres miembros conocidos como p21Cip1/Waf1, p27Kip1 y p57Kip2. Estos forman complejos heterotriméricos con las CDK y ciclinas restantes inhibiendo, de este modo, su actividad quinasa. Hay evidencias que estos inhibidores no sólo no inhibirían los complejos CDK4/6 sino que, por el contrario, aumentarían su actividad favoreciendo la estabilidad de su unión con la ciclina D1. Los CKI son inducidos por una gran variedad de estímulos en respuesta a diferentes procesos metabólicos, regulación que, en muchos casos, aún no es completamente conocida (Pei and Xiong, 2005; Vidal and Koff, 2000). Sustratos de CDK Los principales sustratos de las CDK4/6 y CDK2 son las proteínas que se incluyen en la familia retinoblastoma (Rb), es decir la proteína retinoblastoma propiamente dicha, p107 y p130. Estas proteínas interaccionan con la familia de factores de transcripción E2F de modo de mantenerlos inactivos durante la fase M y en G0. Los complejos Rb-E2F participan en la represión activa de los promotores de ciertos genes, involucrados en la proliferación celular. Este mecanismo de represión involucra el reclutamiento, por medio del complejo Rb-E2F, de proteínas involucradas en el mantenimiento de una estructura cerrada de la cromatina, tales como histona deacetilasas e histona metiltransferasas. La actividad de las proteínas Rb es modulada por la fosforilación secuencial catalizada por los complejos CDK4/6-ciclina D y CDK2-ciclina E y, en menor medida, por la acetilación mediada por histona acetiltransferasas como p300/CBP. La hiperfosforilación de las proteínas Rb, libera las moléculas que permanecían unidas en las isoformas hipofosforiladas de Rb, principalmente los factores E2F, permitiendo la prosecución hacia otras etapas del ciclo celular (Trimarchi and Lees, 2002).
Regulación de la fase G1 en células normales
Durante la fase G1 temprana las células integran la información proveniente de los estímulos mitogénicos y de la disponibilidad de nutrientes y se preparan para comenzar la proliferación y el pasaje a través de las siguientes fases del ciclo celular. Como ya vimos, la activación de las vías de transducción apropiadas genera un aumento en los niveles nucleares de ciclinas de tipo D con la subsecuente formación de los complejos con las CDK4/6 y su posterior activación. La proteína Rb comienza a ser parcialmente fosforilada, lo que resulta en la activación de los factores de transcripción de la familia E2F quienes, interaccionando con los factores DP, inducen la expresión de un gran número de genes. Las moléculas proteicas generadas en respuesta a esta activación, entre ellas la ciclina E, son requeridas para completar la fase G1. El aumento de ciclina E, permite la activación de CDK2 que fosforila a su vez a la proteína Rb permitiendo una mayor disponibilidad de factores E2F-DP lo que asegura la síntesis de las proteínas necesaria para el ingreso en la siguiente fase del ciclo, la fase S. En esta situación la prosecución a lo largo del ciclo celular se haría independiente del influjo de mitógenos, ya que la actividad CDK2-ciclina E reemplazaría al complejo CDK4/6-ciclina D en la fosforilación de Rb y, por ende, en la activación de los factores E2F (Attwooll et al., 2004; Cam and Dynlacht, 2003). Las CDK4/6 contribuyen a la activación de CDK2 de dos modos. El primero, como ya se mencionó, fosforilando parcialmente a Rb e incrementando la síntesis de su subunidad regulatoria, la ciclina E. La segunda, por medio de la formación de complejos heterotriméricos CDK4/6-ciclina D-inhibidores Cip/Kip. Esta interacción impide que esta familia de CKI inhiba la naciente actividad de CDK2, permitiendo la transición G1/S. Al alcanzarse esta transición, la aparición de los inhibidores de la familia INK4, desplazan a la ciclina D de su interacción con CDK4/6, lo que lleva a la degradación de ciclina D, según ya se vio, y a la liberación de los inhibidores Cip/Kip, secuestrados por el complejo mencionado. El aumento de los niveles de estos inhibidores Cip/Kip, conducen a la inactivación de CDK2 y la degradación de ciclina E, ya que es sustrato de una ubiquitin ligasa específica. En estos momentos, han aumentado los niveles de ciclina A, que reemplaza a la anterior en su unión a CDK2, permitiendo la continuidad del ciclo a través de la fase S. Es evidente que, para una nueva ronda de proliferación, se necesitará de una renovada estimulación con factores mitogénicos (Massague, 2004).
Alteraciones en proteínas del ciclo celular encontradas en tumores humanos
El análisis genético y molecular de neoplasias desarrolladas en seres humanos demuestra que, en la gran mayoría de ellos, se encuentran mutaciones o alteraciones en la expresión de genes que codifican para proteínas reguladoras del ciclo celular. Este hecho subraya la importancia del estricto control que debe ser mantenido sobre el equilibrio quiescencia–proliferación para la prevención del cáncer en humanos. Es este el motivo por el cual, prácticamente en todos los tumores, hay algún tipo de mutación o alteración de la expresión en elementos del ciclo que gobiernan el pasaje por la fase G1 o la transición G1/S. El eje ciclina D–CDK4/6–INK4–Rb se encuentra casi universalmente alterado en los cánceres humanos. Si bien en algunos tumores se registra la pérdida por mutación de Rb, la mayoría de ellos conservan una proteína Rb salvaje, pero tienen fuertemente incrementada la actividad del complejo ciclina D-CDK4/6 debido a múltiples factores. El principal mecanismo para el aumento de esta actividad corresponde a la inhibición de la familia de inhibidores INK4, especialmente p16INK4a, ya sea por supresión parcial del gen, mutaciones puntuales o silenciamiento transcripcional por metilación de su promotor. Este bloqueo de p16INK4a permite a las células, durante la evolución maligna, sortear la senescencia y la deja con una alta actividad de CDK4/6 lo que se traduce en una ventaja proliferativa y de crecimiento. La proteína p16INK4a es, por lo tanto, un supresor tumoral muy importante, habiéndose demostrado que, ratones que tienen anulado el gen para dicha proteína, presentan una marcada predisposición a la tumorigénesis. Otros mecanismos alternativos para la alteración del eje mencionado consisten en la amplificación del gen de CDK4, con el consiguiente aumento en el número de moléculas proteicas, lo que incrementa significativamente la actividad proliferativa, y la mutación conocida como CDK4R24C. Esta mutación puntual en el gen de la CDK4, frecuente en melanomas muy agresivos, resulta en una CDK4 sin capacidad de unirse a las proteínas INK4, por lo que mantiene una actividad catalítica prácticamente constitutiva. Estos factores que predisponen a la formación de tumores: la pérdida de Rb, la inactivación de p16INK4a y la amplificación o mutación de CDK4, son eventos mutuamente excluyentes. Se ha visto que, en modelos animales, la expresión de la mutante CDK4R24C y la supresión de las proteínas INK4 no actúan en forma cooperativa en el desarrollo de tumores, siendo este hecho consistente con la hipótesis que indica que la función primaria de las proteínas de la familia INK4 es la inhibición de la actividad de CDK4 (Lowe et al., 2004; Shapiro, 2006). Otro evento común en el desarrollo de tumores es la sobrexpresión de ciclina D1, debida a rearreglos cromosomales, que se presenta en hiperplasias mamarias, linfomas y mielomas, o por amplificación génica. También se ha observado un “splicing” alternativo del mRNA de ciclina D1, obteniéndose un transcripto que codifica a la ciclina D1b a la que le falta el extremo carboxilo. En este extremo se ubica la treonina 286 que es fosforilada por la GSK3 que, como se mencionó más arriba, señaliza para la exportación nuclear y ulterior degradación de dicha ciclina. Esta variante, entonces, es constitutivamente nuclear exhibiendo una elevada capacidad transformadora sugiriendo que la disponibilidad de la ciclina D1 en el núcleo y su mayor estabilidad es un factor más importante para la tumorigenicidad que la sobrexpresión. Al contrario de lo que ocurre con CDK4, la sobrexpresión de ciclina D1 a menudo es acompañada por la pérdida de la función de las INK4, sugiriendo que ambos eventos cooperan en la promoción de la transformación oncogénica. Por otro lado, la pérdida de p16INK4a o la sobrexpresión de ciclina D1 incrementa la cantidad de CDK4 disponible para su ensamblado con ciclina D y con las proteínas de la familia Cip/Kip. Este secuestro de los inhibidores Cip/Kip por el complejo CDK4/6-ciclina D resulta en una mayor activación de CDK2-ciclina E y, por ende, en un incremento de la fosforilación e inactivación de la proteína retinoblastoma. Por lo tanto, el aumento de la actividad de CDK2 y de la capacidad proliferativa celular, es también consecuencia de alguna de las alteraciones descriptas en el eje ciclina D–CDK4/6–INK4–Rb. En algunos tumores se encuentra una forma hiperactiva de ciclina E, de bajo peso molecular, generada por proteólisis debida a la acción de la elastasa o la calpaína o la presencia de niveles disminuidos del inhibidor p27Kip1 como resultado de un incremento en su degradación mediada por proteasoma. Ambas situaciones dan como consecuencia una proliferación celular aumentada (Malumbres and Barbacid, 2001; Massague, 2004; Schwartz and Shah, 2005).
Conclusiones
Podemos considerar el cáncer como una patología del ciclo celular. En forma precisa, una enfermedad restringida al inicio del ciclo, es decir la fase G1 y la transición hacia la fase S. En estas etapas, residirían los eventos moleculares críticos, cuya desregulación, habilita, potencialmente, la formación de tumores. Aunque, como hemos visto, muchos mecanismos reguladores específicos del ciclo celular han sido estudiados en profundidad in vitro, quedan todavía muchas incógnitas acerca de cómo estos procesos celulares están desregulados en forma coordinada con la división celular en cánceres humanos. Los estudios actuales se han focalizado en aspectos cruciales de la regulación del ciclo celular in vivo, tales como los caminos de síntesis y degradación o proteólisis de proteínas esenciales para el ciclo, su regulación por modificaciones postraduccionales y las funciones redundantes o específicas entre los miembros de las distintas familias de proteínas intervinientes, en especial las de los CKI. La estrecha relación entre el ciclo celular y el cáncer se ve reflejada en el gran número de terapias antitumorales, en distintas fases de los estudios clínicos, que apuntan a algunos de los mecanismos que gobiernan las etapas mencionadas del ciclo celular.
Referencias
Attwooll, C., Denchi, E. L., and Helin, K. (2004). The E2F family: specific functions and overlapping interests. Embo J.
Ben-Porath, I., and Weinberg, R. A. (2005). The signals and pathways activating cellular senescence. Int J Biochem Cell Biol 37, 961-76.
Blume-Jensen, P., and Hunter, T. (2001). Oncogenic kinase signalling. Nature 411, 355-65.
Cam, H., and Dynlacht, B. D. (2003). Emerging roles for E2F: beyond the G1/S transition and DNA replication. Cancer Cell 3, 311-6.
Cunningham, J. J., and Roussel, M. F. (2001). Cyclin-dependent kinase inhibitors in the development of the central nervous system. Cell Growth Differ 12, 387-96.
Donzelli, M., and Draetta, G. F. (2003). Regulating mammalian checkpoints through Cdc25 inactivation. EMBO Rep 4, 671-7.
Essers, J., van Cappellen, W. A., Theil, A. F., van Drunen, E., Jaspers, N. G., Hoeijmakers, J. H., Wyman, C., Vermeulen, W., and Kanaar, R. (2005). Dynamics of relative chromosome position during the cell cycle. Mol Biol Cell 16, 769-75.
Hahn, W. C., and Weinberg, R. A. (2002). Modelling the molecular circuitry of cancer. Nat Rev Cancer 2, 331-41.
Hanahan, D., and Weinberg, R. A. (2000). The hallmarks of cancer. Cell 100, 57-70.
Kastan, M. B., and Bartek, J. (2004). Cell-cycle checkpoints and cancer. Nature 432, 316-323.
Lowe, S. W., Cepero, E., and Evan, G. (2004). Intrinsic tumour suppression. Nature 432, 307-15.
Lukas, J., and Bartek, J. (2004). Cell division: the heart of the cycle. Nature 432, 564-7.
Lukas, J., Lukas, C., and Bartek, J. (2004). Mammalian cell cycle checkpoints: signalling pathways and their organization in space and time. DNA Repair (Amst) 3, 997-1007.
Lutzen, A., Bisgaard, H. C., and Rasmussen, L. J. (2004). Cyclin D1 expression and cell cycle response in DNA mismatch repair-deficient cells upon methylation and UV-C damage. Exp Cell Res 292, 123-34.
Malumbres, M., and Barbacid, M. (2001). To cycle or not to cycle: a critical decision in cancer. Nat Rev Cancer 1, 222-31.
Martin, G. S. (2003). Cell signaling and cancer. Cancer Cell 4, 167-74.
Massague, J. (2004). G1 cell-cycle control and cancer. Nature 432, 298-306.
Murray, A. W. (2004). Recycling the cell cycle: cyclins revisited. Cell 116, 221-34.
Ortega, S., Malumbres, M., and Barbacid, M. (2002). Cyclin D-dependent kinases, INK4 inhibitors and cancer. Biochim Biophys Acta 1602, 73-87.
Pei, X. H., and Xiong, Y. (2005). Biochemical and cellular mechanisms of mammalian CDK inhibitors: a few unresolved issues. Oncogene 24, 2787-95.
Radimerski, T., Montagne, J., Rintelen, F., Stocker, H., van der Kaay, J., Downes, C. P., Hafen, E., and Thomas, G. (2002). dS6K-regulated cell growth is dPKB/dPI(3)K-independent, but requires dPDK1. Nat Cell Biol 4, 251-5.
Roussel, M. F. (1999). The INK4 family of cell cycle inhibitors in cancer. Oncogene 18, 5311-7.
Sandal, T. (2002). Molecular aspects of the mammalian cell cycle and cancer. Oncologist 7, 73-81.
Schwartz, G. K., and Shah, M. A. (2005). Targeting the cell cycle: a new approach to cancer therapy. J Clin Oncol 23, 9408-21.
Shapiro, G. I. (2006). Cyclin-dependent kinase pathways as targets for cancer treatment. J Clin Oncol 24, 1770-83.
Sherr, C. J. (2004). Principles of tumor suppression. Cell 116, 235-46.
Sherr, C. J., and Roberts, J. M. (1999). CDK inhibitors: positive and negative regulators of G1-phase progression. Genes Dev 13, 1501-12.
Stein, G. S., van Wijnen, A. J., Stein, J. L., Lian, J. B., Montecino, M., Zaidi, S. K., and Braastad, C. (2006). An architectural perspective of cell-cycle control at the G1/S phase cell-cycle transition. J Cell Physiol 209, 706-10.
Trimarchi, J. M., and Lees, J. A. (2002). Sibling rivalry in the E2F family. Nat Rev Mol Cell Biol 3, 11-20.
Vidal, A., and Koff, A. (2000). Cell-cycle inhibitors: three families united by a common cause. Gene 247, 1-15.
Wullschleger, S., Loewith, R., and Hall, M. N. (2006). TOR signaling in growth and metabolism. Cell 124, 471-84.
*Doctor en Ciencias Químicas Profesor Adjunto DE Investigador Independiente CONICET
ISSN 1666-7948
www.quimicaviva.qb.fcen.uba.arRevista QuímicaViva
Número 1, año 6, Mayo 2007
quimicaviva@qb.fcen.uba.ar