Dos premios Nobel para el RNA
Paula Cramer
Investigadora del CONICET, Jefe de trabajos prácticos del Depto. de Fisiología, Biología Molecular y Celular, FCEN, UBA
Laboratorio de Fisiología y Biología Molecular, Departamento. de Fisiología, Biología Molecular y Celular-IFIBYNE-CONICET, Facultad de Ciencias Exactas y Naturales, Universidad de Buenos Aires. Ciudad Universitaria. Pabellón 2. 2do Piso. C1428EGA. Ciudad Autónoma de Buenos Aires. Argentina.
Recibido el 21/11/06. Aceptado el 5/12/06.
Two Nobel awards for RNA
This year, the Royal Swedish Academy of Sciences awarded its prizes in Chemistry and Physiology or Medicine to researchers working in key aspects of the regulation of gene expression. The reasons for the choice of awardees, the historical context and impact of their research are discussed.
Este año, la Academia Real Sueca de Ciencias otorgó sus premios de Química y Medicina a investigadores que trabajan en aspectos claves de la regulación de la expresión génica.
La información necesaria para la supervivencia, mantenimiento y crecimiento de nuestras células está contenida en los genes, compuestos de ácido desoxirribonucleico (DNA) y localizados en el núcleo. Cuando se requiere la información guardada en un determinado gen, éste se "expresa" promoviendo la generación de una copia de sí mismo compuesta de ácido ribonucleico (RNA). A este proceso de copiado se lo denomina transcripción. Luego de sufrir una serie de modificaciones, este RNA "mensajero" (mRNA) viaja fuera del núcleo hacia el citoplasma, en donde es leído por una maquinaria que genera a partir de él una proteína. Para que se pueda producir el mRNA, los genes contienen regiones denominadas promotores, que son capaces de reclutar a una serie de proteínas que conforman la maquinaria de transcripción. Ésta básicamente reconoce el inicio del gen y es capaz de fabricar el mRNA en el momento y cantidad requeridos.
Las células de nuestro hígado son distintas de nuestras neuronas porque -a pesar de poseer la misma información genética- expresan distintos genes en distintas situaciones. Asimismo, las células normales pueden volverse tumorales cuando se altera la expresión de ciertos genes responsables de funciones esenciales tales como el control de la proliferación. Es decir, que en la regulación de la expresión génica está la clave para entender estos procesos.
Premio Nobel de Química 2006: Roger Kornberg
Una vida dedicada a la transcripción del RNA
El premio Nobel de Química 2006 fue otorgado a Roger Kornberg, de la Universidad de Stanford, EE.UU, quien dedicó toda su carrera científica a estudiar distintos aspectos del proceso de transcripción. Durante su postdoctorado en los años 70 descubrió los nucleosomas, mientras trabajaba con los premios Nobel Aaron Klug y Francis Crick, en el Laboratorio de Biología Molecular del Medical Research Council (MRC), en Cambridge, Reino Unido.
Los nucleosomas son estructuras formadas por unas proteínas llamadas histonas, alrededor de las cuales se empaqueta el DNA. Todo el DNA de la célula se halla empaquetado en series de nucleosomas, constituyendo la cromatina. Hoy sabemos que la regulación de la expresión de los genes está determinada en parte por la estructura de la cromatina.
Una vez en su laboratorio de Stanford, Kornberg se abocó a estudiar la maquinaria de transcripción de los eucariontes (organismos cuyo DNA se encuentra en el núcleo). Eligió como sistema de estudio a las levaduras, organismos unicelulares muy sencillos de cultivar en el laboratorio. Estas resultaron un muy buen modelo de estudio ya que la maquinaria transcripcional se encuentra en todos los organismos vivos y que hoy sabemos que con excepción de las bacterias (que carecen de núcleo) es muy similar en todos ellos.
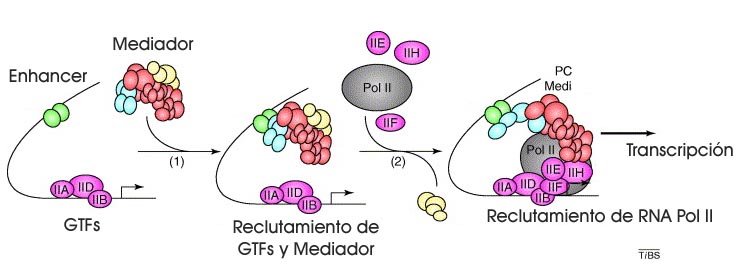
Al mismo tiempo, muchos otros laboratorios estudiaron la maquinaria transcripcional en levaduras o en otros sistemas biológicos. Así, se estableció que ésta se compone de una serie de proteínas denominadas factores generales de transcripción (GTFs), que son reclutados por el promotor y que a su vez reclutan a la RNA polimerasa II (pol II), la proteína encargada de fabricar el mRNA. También se describió la existencia de otras regiones de los genes, denominadas enhancers, que a su vez reclutan a los coactivadores, proteínas capaces de estimular el reconocimiento del promotor por parte de la pol II.
Kornberg se destacó al descubrir un complejo de unas veinte proteínas, que denominó mediador, que asiste al reconocimiento de los promotores por parte de la maquinaria transcripcional. El mediador puede reclutar coactivadores y también represores de la transcripción (Figura 1). La caracterización de este complejo permitió entender el hecho de que los enhancers ejercieran su efecto sobre los promotores aún a grandes distancias. En distintos tejidos, los enhancers reclutan distintos tipos de coactivadores y de complejos mediadores, lo cual explica en parte la expresión diferencial de tejidos de ciertos genes.
 |
|
Figura 1: La maquinaria transcripcional de levaduras
|
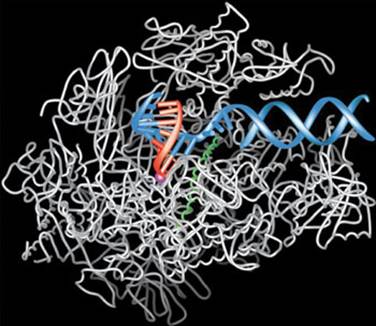
Recientemente, el laboratorio de Kornberg se dedicó a obtener una imagen del proceso de transcripción en acción. Para ello, obtuvo imágenes de los complejos DNA-pol II-RNA mediante cristalografía de rayos X. Esta técnica consiste en obtener grandes cantidades del objeto de estudio muy puro, en forma de cristales, que luego se irradian con rayos X. La dispersión de los rayos al atravesar los cristales permite deducir -mediante ecuaciones- la composición de átomos de la molécula. Obtener la estructura átomo a átomo de semejante complejo macromolecular fue un logro sorprendente, comunicado por la revista Science en el año 2001, ver Figura 2.
¿Para qué sirve obtener una imagen del complejo en acción? No se puede entender ni manipular lo que no se conoce, y conocer los detalles de la estructura macromolecular permite no sólo explicar los efectos de ciertas mutaciones o cambios hallados en la naturaleza, sino también saber qué regiones de la pol II son claves para su interacción con el DNA y el RNA, y eventualmente seleccionar fármacos que modifiquen estas interacciones. Tales fármacos son herramientas invalorables en el estudio de la regulación de la expresión génica y hasta podrían ser usados en terapias moleculares para tratar enfermedades humanas.
|
|
|
Figura 2: El complejo transcripcional en acción. Imagen del complejo transcripcional compuesto por DNA, RNA polimerasa II y transcripto de RNA, obtenida por cristalografía de rayos X. En gris se muestra la estructura de la RNA polimerasa II, en azul, el DNA que se usa como "molde" en la transcripción, y en rojo, el RNA transcripto. Adaptado de (2). |
Sin duda fue necesaria la contribución de muchísimos investigadores de diversas instituciones para alcanzar el nivel de conocimiento actual que se tiene de la maquinaria transcripcional. La Academia Sueca quizás haya querido premiar a Roger Kornberg por la dedicación de toda una vida a la transcripción, con numerosas contribuciones seminales y coronada con la imagen del complejo transcripcional en acción.
Para algunos, este premio habría sido más apropiado para la categoría Fisiología o Medicina. El comité de selección defendió su elección basándose en que la obtención de la imagen del complejo de transcripción a alta resolución
es pertinente para un premio Nobel de Química. Quizás lo que subyace a esta discusión es tan sólo el hecho de que estas categorías estancas -creadas hace más de un siglo- van perdiendo sentido a medida que la ciencia se vuelve más y más interdisciplinaria.
Es interesante destacar que Kornberg es un buen ejemplo de cómo las escuelas científicas pueden producir excelencia en forma sostenida. No sólo es hijo de otro premio Nobel (Arthur Kornberg, 1959, por el descubrimiento de la DNA polimerasa) sino que, como se mencionó antes, se formó en el MRC bajo la supervisión de científicos cuyos trabajos contribuyeron a la descripción de la estructura del DNA.
Premio Nobel de Fisiología o Medicina 2006: Andrew Fire y Craig Mello
Genes silenciados, de las petunias a los gusanos
A principios de los años 90, un grupo de investigación en EE.UU y otro en Holanda estaban estudiando la ruta de biosíntesis de pigmentos en las flores de petunia. Con el fin de lograr flores de color más intenso, los investigadores generaron plantas transgénicas que debían expresar cantidades altas de la enzima clave de este camino biosintético. Sorprendidos, observaron que algunas de las petunias transgénicas eran blancas. Es decir que no sólo no fabricaban altas cantidades de la proteína en cuestión sino que, por el contrario, habían silenciado su expresión tanto a partir del transgen como del gen endógeno o propio (Figura 3). Por otro lado, el grupo de investigación de David Baulcombe, en el Reino Unido, demostraba que, al introducir genes virales en plantas transgénicas, se las protegía de la infección por el virus. En tanto en Roma, otros investigadores hicieron observaciones similares en el hongo Neurospora crassa.
|
|
|
Figura 3: Silenciamiento de la pigmentación de las flores La figura muestra flores de petunias transgénicas para la chalcona sintetasa, principal responsable de la expresión de pigmentos en las flores. Las petunias originales eran de color azul oscuro. Las distintas plantas muestran diferentes grados de silenciamiento del gen. Tomado de (3).
|
En ese momento se desconocía el mecanismo por el cual ocurría esto, y se especulaba que el silenciamiento estaba de algún modo mediado por el RNA y que se debía a modificaciones en el DNA que alteran su interacción con las histonas.
Unos años después, estudiando la regulación de la expresión génica en el gusano nematodo Caenorhabditis elegans, Andrew Fire y Craig Mello encontraron que la inyección de RNA doble cadena correspondiente a una proteína de músculo, provocaba el mismo comportamiento que el de los gusanos que carecían totalmente del gen para la proteína en cuestión. Extendieron esta observación a muchos otros genes, concluyendo que es posible silenciar la expresión de un gen a través de la introducción del RNA doble cadena correspondiente. Aún más, este silenciamiento se dispersaba a lo largo del gusano y era heredable, según se reportó en la revista Nature, en 1998. Por fin se podía explicar el silenciamiento que se había observado en las plantas y los hongos. Este hallazgo proveyó de una herramienta fundamental a la genética funcional, que marcó un antes y un después, probablemente sólo comparable al de la invención de la técnica de la reacción en cadena de la polimerasa, PCR (Polymerase Chain Reaction).
¿Para qué sirve silenciar un gen? A partir del estudio de los efectos que produce el anular la expresión de un gen puede deducirse nada menos que su función.
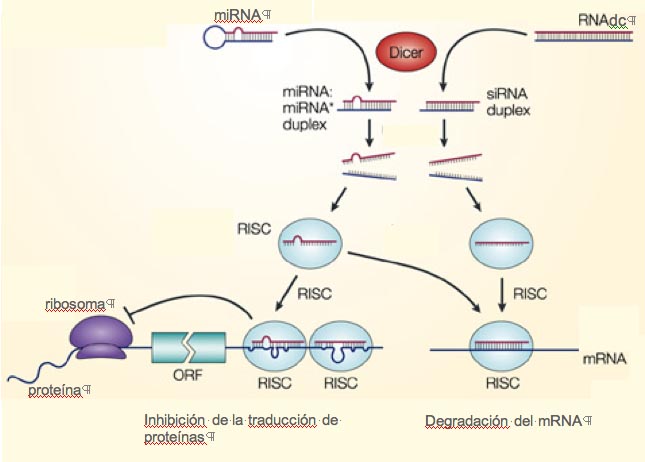
Dada la importancia del hallazgo no tardaron en describirse los detalles del mecanismo por el cual estos RNA bloquean la expresión génica. Mientras que el DNA de los genes está compuesto por dos hebras o cadenas de DNA complementarias, el RNA transcripto a partir de él es de simple cadena. Algunos virus generan RNA doble cadena (RNAdc) dentro de las células a las que infectan. Lo que los experimentos de Fire y Mello permitieron descubrir fue que las células infectadas detectan estos RNA de cadena doble y montan una respuesta de defensa, que termina con la degradación del RNA invasor. En esta respuesta participa la proteína Dicer, que corta el RNAdc en pequeños fragmentos de 21 a 23 nucleótidos, denominados siRNA (por small interfering RNA, en inglés), que luego son reclutados por un complejo proteico denominado RISC. Este complejo se encarga luego de utilizar la información del siRNA para silenciar específicamente la expresión del gen del cual se había originado (Figura 4).
|
|
|
Figura 4: Los RNA pequeños regulan la expresión génica La proteína Dicer genera siRNA pequeños a partir de microRNA propios de la célula (miRNA) o de RNA de doble cadena invasores (RNA dc). El complejo proteico RISC reconoce estos RNA pequeños e impide o la traducción de la proteína o la degradación del mRNA correspondiente a los RNA pequeños. Adaptado de (4)
|
En el escaso tiempo transcurrido desde los experimentos de Fire y Mello, floreció el campo de los microRNA (miRNA). Se descubrió que aparte de participar en la defensa contra infecciones virales, los RNA pequeños participan en múltiples procesos biológicos a nivel celular, inclusive en la regulación de la transcripción de los genes. Los miRNA estaban en todas partes, pero hasta hace muy poco, no sabíamos de su existencia.
Por algún tiempo no fue posible el uso de siRNA en células de mamíferos, porque disparaban la respuesta suicida de interferón de las células. Hasta que en el año 2001, el laboratorio de Thomas Tuschl, del Max-Planck-Institute de Goettingen, Alemania, encontró el modo de utilizar el RNA de interferencia en células de mamífero en cultivo. En cuestión de semanas, la técnica de siRNA se había dispersado por los laboratorios de todo el mundo. Esto permitió un progreso increíble en el estudio de la función de los genes y también promete un gran avance en terapias génicas en las que se busca, por ejemplo, silenciar algún gen involucrado en el desarrollo de tumores.
La espiral de la Historia
Si bien todos los laboratorios mencionados tienen una afiliación académica, las primeras observaciones de silenciamiento de genes se hicieron en el contexto del mejoramiento de cultivos de interés económico: la manipulación del color de las flores en Holanda, la generación de plantas resistentes a virosis. Mello y Fire estaban interesados en estudiar la regulación de la expresión génica en un nematodo cuando desentrañaron el mecanismo de silenciamiento por siRNA que resultó casi universal. Como se mencionó antes, el impacto de este hallazgo es tan profundo para quienes investigan la función de un gen como para el desarrollo de nuevas vacunas o terapias génicas.
La historia reciente de los siRNA ilustra cuán difícil es saber hacia dónde conduce un proyecto de investigación, o intentar encasillarlo en las categorías de ciencia básica o aplicada.
Referencias
1. Malik S. And Roeder R . Dynamic regulation of pol II transcription by the mammalian Mediator Complex. Trends in Biochem. Sci. 2005. 30 (5): 256-263
2. Gnatt A.L. ,Cramer P., Fu J.l., Bushnell D., Korberg R.D. Structural Baseof Transcription. RNA polymerase II Elongation Complex at 3.2 A resolution. Science. 2001. 292 (5523):1876-1882.
3. Napoli C., Lemieux C. and Jorgenesen R. Introduction of a Uumeric Chalone Synthase Gene in Petunia Results in Reversible Co-suppresssion of Homologous Genes in Trans . Plant Cell. 1990 2: 279-289 .
4. He, L. and Hannon, G. MicroRNAs small RNAs with a big role in gene regulation. Nature Reviews Genetics 2004. 5, 522–531.
|
|
Revista QuímicaViva Número 3, año 5, diciembre 2006 quimicaviva@qb.fcen.uba.ar |