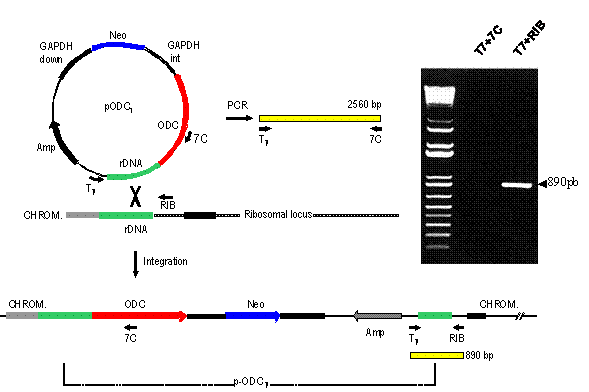
Figura 12A Esquemas posibles de organización génica. B Productos
de PCR.
El gen heterólogo de ADC introducido en el plásmido recombinante se
encuentra bajo el control del promotor ribosomal. Por esta razón la enzima
responsable de la transcripción del gen resultó ser la RNA polimerasa I (que
también sintetiza el RNA ribosomal) y no RNA polimerasa II que transcribe la
mayoría de los genes que codifican proteínas. Estas conclusiones se basan en
la sensibilidad a alfa-amanitina de la transcripción del gen de ADC (Figura
13).
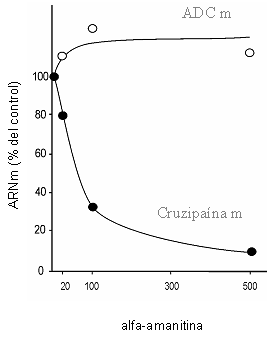
Figura
13: Sensibilidad a alfa-amanitina de la transcripción de ADC y cruzipaína
Conclusiones
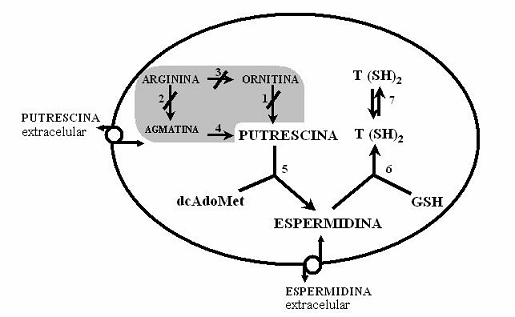
a) El
protozoario Trypanosoma cruzi no contiene las actividades enzimáticas
de ODC y ADC, por lo que es incapaz de sintetizar poliaminas “de novo”.
b) La
falta de las actividades enzimáticas se debe a la ausencia de los genes
correspondientes en su genoma.
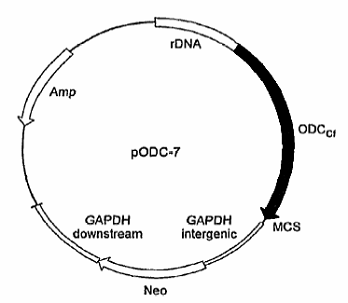
c) Los
parásitos transformados con plásmidos recombinantes adecuados pueden expresar
genes heterólogos de ODC y ADC, demostrando que el medio intracelular de T.
cruzi no inhibe la expresión de los genes mencionados.
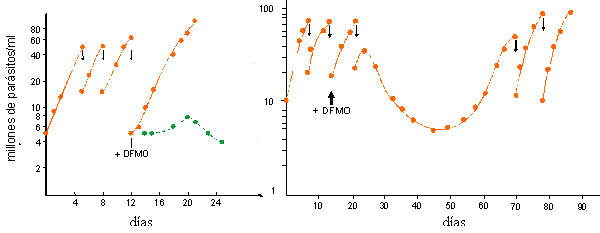
d) La
presencia de DFMO por períodos prolongados en los cultivos de parásitos
transformados con el gen de ODC induce la amplificación del gen exógeno y la
aparición de T. cruzi resistentes a la droga.
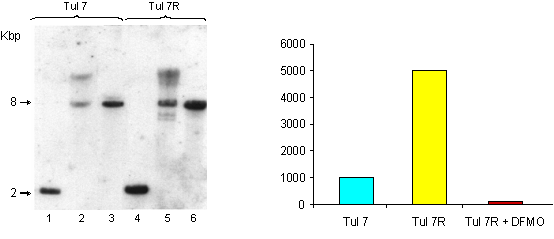
e)
Ambos genes heterólogos se integran al genoma del parásito aunque posiblemente
con diferentes mecanismos: 1) los T. cruzi transformados con ODC
muestran una integración temprana de una copia del plásmido; 2) en cambio, en
los parásitos transfectados con ADC, el plásmido recombinante permanece como
episoma por varias semanas. Durante este período la actividad de la enzima es
transitoria y posteriormente, a las 4 o 6 semanas, la expresión se vuelve
estable con la concomitante integración de dos o más copias en “tandem” del
plásmido transformante en el genoma de T. cruzi.
Agradecimientos
Los experimentos
descriptos realizados en nuestro laboratorio fueron financiados en parte por
el Consejo Nacional de Investigaciones Científicas y Técnicas, la Universidad
de Buenos Aires, la Agencia de Promoción Científica y Tecnológica y el
International Centre for Genetic Engineering and Biotechnology (ICGEB, Trieste,
Italia).
Bibliografía
1.
Cohen S, 1998. A guide to the polyamines.
Oxford University Press, Oxford.
2.
Pegg AE, 1988. Polyamine metabolism and its importance in neoplastic
growth and as a target for chemotherapy. Cancer Res. 48: 759-774.
3.
Wallace HM, 1998. Polyamines: specific metabolic regulators or multifunctional
polycations? Biocem. Soc. Trans. 26: 569-571.
4.
Wallace HM, Fraser AV, Hughes A, 2003. A perspective of polyamine metabolism.
Biochem. J. 376: 1-14.
5.
Igarashi K, Kashiwagi K, 2000. Polyamines: mysterious modulators of cellular
functions. Biochem. Biophys. Res. Commun. 271: 559-564.
6.
Tabor CW, Tabor H, 1984. Polyamines. Annu. Rev. Biochem. 53: 749-790.
7.
Pegg AE, 1986. Recent advances in the biochemistry of polyamines in eukeryotes.
Biochem. J. 234: 249-262.
8.
Bolkenius FN, Seiler N, 1981.
Acetyl derivatives as intermediates in polyamine catabolism. Int. J. Biochem.
13: 287-292
9.
Heby O, Persson L, 1990. Molecular genetics of polyamine synthesis in
eukaryotic cells. Trends Biochem. Sci. 15: 153-158.
10.
Hayashi S, Murakami Y, 1995.
Rapad and regulated degradation of ornithine decarboxylase. Biochem. J. 306:
1-10.
11.
Hayashi S, Murakami Y, Matsufuji S, 1996. Ornithine decarboxylase antizyme: a
novel type of regulatory protein. Trends. Biochem. Sci. 21: 27-30.
12.
Murakami Y, Matsufuji S, Hayashi S, Tanahashi N, Tabaka K, 2000. Degradation
of ornithine decarboxylase by the 26S proteosome. Biochem. Biophys. Res.
Commun. 267: 1-6.
13.
Metcalf BW, Bey P, Danzin C, Jung MJ, Casara P, Vevert JP, 1978. Catalytic
irreversible inhibition of mammalian ornithine decarboxylase by substrate and
product analogs. J. Am. Chem. Soc. 100: 2551-2553.
14.
Marton LJ, Pegg AE, 1995. Polyamines as targets for therapeutic entervention.
Annu. Rev. Pharmacol. Toxicol. 35: 55-91.
15.
Wang CC, 1995. Molecular mechanisms and therapeutic approaches to the
treatment of African trypanosomiasis. Annu. Rev. Pharmacol. Toxicol. 35:
93-127.
16.
Pegg AE, McCann PP, 1982. Polyamine metabolism and function. Am. J. Physiol.
243: C212-C221.
17.
Saydjari R, Alexander RW, Upp Jr JR, Barranco SC, Townsed Jr. CM, Thompson JC,
1991. Differential sensitivity of various human tumors to inhibition of
polyamine biosynthesis in vivo. Int. J. Cancer. 47: 44-48.
18.
Fairlamb AH, 1981. Alternate metabolic pathways in protozoan energy
metabolism. Parasitology. 82: 1-30.
19.
Pegg AE, McCann PP, 1988. Polyamine metabolism and function in mammalian cells
and protozoans. ISI Atlas of Science: Biochemistry 11-18.
20.
Sánchez CP.
González NS, Algranati ID, 1989. Stable ornithine decarboxylase in
promastigotes of Leishmania mexicana. Biochem. Biophys. Res. Común.
161: 754-761.
21.
Phillips MA, Coffino P, Wang CC, 1987.
Cloning and sequencing of the ornithine decarboxylase gene from T. brucei.
J. Biol. Chem. 262: 8721-8727.
22.
Goda L, Phillips MA, Bass KE, Wang CC, Coffino P, 1990. Trypanosome ornithine
decarboxylase is stable because it lacks sequences found in the carboxyl
terminus of the mouse enzyme which target the latter for intracellular
degradation. J. Biol. Chem. 265: 11823-11826.
23.
Goda L, Sidney D, Macrae M, Coffino P, 1992. Structural elements of ornithine
decarboxylase required for intracellular degradation and polyamine-dependent
regulation. Mol. Cell. Biol. 12: 2178-2185.
24.
Svanson F, Ceriani C, Lövkvist Wallström E, Kockum I, Algranati ID, Heby O,
Persson L, 1997. Cloning of a trypanosomatid gene cloning for an ornithine
decarboxylase that is metabolically unstable even though it lacks the
C-terminal degradation domain.
Proc.
Nat. Acad. Sci. USA. 94: 397-402.
25.
Carrillo C, Cejas S, Cortés M, Ceriani C, Huber A, González NS, Algranati ID,
2000.
Sensitivity of trypanosomatid protozoa to DFMO and metabolic turnover of
ornithine decarboxylase. Biochem. Biophys. Res. Commun. 279: 663-668.
26.
Bacchi CJ, Nathan HC, Hutner SH, McCann PP, Sjoerdsma A, 1980. Polyamine
metabolism: a potencial therapeutic target in trypanosomes, Science 210:
332-334.
27.
Bacchi CJ, McCann PP, 1987. Inhibition of polyamine metabolism. Biological
significance and bases for new therapies. Academic Press, Inc, pp 317-344.
28.
Van Nieuwenhove S, Schechter PJ, Declercq J, Boné G, Burke J, Sjoerdsma A.
1985. Treatment of gambiense sleeping sickness in the
Sudan with oral DFMO, an inhibitor of ornithine decarboxylase: first field
trial. Trans. R. Soc. Trop. Med. Hyg. 79: 692-698.
29.
Fairlamb AH, Cerami A, 1985. Identification of a novel thiol-containing
co-factor essential for glutathione reductase enzyme activity in
trypanosomatids. Mol.Biochem. Parasitol. 14: 187-198.
30.
Shames SL, Fairlamb AH, Cerami A,
Walsh CT, 1986. Purification and characterization of trypanothione reductase
from Crithidia fasciculata, a newly discovered member of the family
disulfide-containing flavoprotein reductases. Biochemistry 25: 3519-3526.
31.
Carrillo C, Cejas S, González NS, Algranati ID, 1999.
Trypanosoma cruzi
epimastigotes lack ornithine decarboxylase but can express a foreign gene
encoding this enzyme.
FEBS
Lett. 454: 192-196.
32.
Algranati ID, Sánchez CP, González NS, 1990.
Polyamines in Trypanosoma cruzi and Leishmania mexicana. The
biology and chemistry of polyamines.
IRL
Press, pp 137-146.
33.
Carrillo C, Serra MP, Pereira CA, Huber A, González NS, Algranati ID, 2004.
Heterologous expression of a plant arginine decarboxylase gene in
Trypanosoma cruzi.
Biochim.
Biophys. Acta. 1674: 223-230.
34.
Carrillo C, Cejas S, Huber A, González NS, Algranati ID, 2003.
Lack of arginine decarboxylase in Trypanosoma cruzi epimasigotes. J.
Eukaryot. Microbiol. 50: 312-316.
35.
González NS, Huber A, Algranati ID, 2001.
Spermidine is essential for normal proliferation of trypanosomatid protozoa.
FEBS Lett. 508: 323-326.
36.
Serra MP, Carrillo C, González NS, Algranati ID, 2006.
Modulation of oat arginine decarboxylase gene expression and genome
organization in transgenic Trypanosoma cruzi epimastigotes. FEBS
Journal 273: 628-637.