Especificidad de la señalizacion cAMP-PKA en Saccharomyces cerevisiae
Silvia Rossi
Universidad de Buenos Aires, Facultad de Ciencias Exactas y Naturales, Departamento Quimica Biologica, CONICET, Instituto de Quimica Biologica de la Facultad de Ciencias Exactas y Naturales (IQUIBICEN), Buenos Aires, Argentina
Resumen
Las células deben poder proteger y mantener su homeostasis frente a la variabilidad de las condiciones externas. La levadura Saccharomyces cerevisiae ha desarrollado sistemas de detección y redes de señalización complejas para responder de manera eficiente a variaciones repentinas y frecuentes del medio como pueden ser fluctuaciones de temperatura, osmolaridad, acidez del ambiente, presencia de toxinas o períodos escasez nutricional. La transmisión de señales celulares requiere de interacciones moleculares reguladas en espacial y temporalmente. El circuito de señalización cAMP-Proteína quinasa A (PKA) se encuentra entre las vías más estudiadas involucradas en la coordinación precisa de las respuestas celulares a diversos estímulos. En levaduras, la glucosa extracelular y una variedad de señales relacionadas con el crecimiento y estrés regulan los niveles intracelulares de AMPc que modulan la actividad de la PKA, que a su vez regula diferentes procesos celulares. La señalización cAMP-PKA requiere una especificidad que permite lograr la respuesta adecuada a cada estimulo. En esta revisión se discutirán mecanismos moleculares que están involucrados en el establecimiento de la especificidad en la vía de señalización cAMP-PKA en S. cerevisiae.
Palabras clave: cAMP-PKA - Especificidad - Transducción de señales - Proteínas de anclaje - BCY1- TPK - Transcripción - Saccharomyces cerevisiae
Summary
Cells must be able to protect and maintain their homeostasis against the variability of external conditions. The yeast Saccharomyces cerevisiae has developed has developed sensing systems and complex signaling networks to efficiently respond to sudden and frequent variations in the environment such as fluctuations in temperature, osmolarity, acidity of the environment, presence of toxins or periods of nutritional starvation. Cellular signal transmission requires spatially and temporally regulated molecular interactions. The cAMP-Protein Kinase A (PKA) signaling circuit is among the most studied pathways involved in the precise coordination of cellular responses to various stimuli. In yeast, extracellular glucose, and a variety of growth- and stress-related signals regulate intracellular levels of cAMP that modulate PKA activity, which in turn regulates different cellular processes. cAMP-PKA signaling requires a specificity that makes it possible to achieve the appropriate response to each stimulus. In this review we will discuss molecular mechanisms that are involved in the establishment of specificity in the cAMP-PKA signaling pathway in S. cerevisiae.
Keywords: cAMP-PKA - Specificity - Signal transduction - Anchoring Proteins -BCY1- TPK - Transcription - Saccharomyces cerevisiae
Introducción
Las células detectan estímulos extracelulares para ajustar los procesos intracelulares de manera apropiada a esos cambios y mantener la homeostasis celular. Por lo tanto, la clave es garantizar que las señales sean específicas y sujetas a un estricto control [1-3]. En la mayoría de los casos, las células responden a los cambios ambientales mediante la acción de cascadas de enzimas. El control espacial y temporal de la transducción de señales se logra con frecuencia mediante la compartimentación de los efectores intracelulares a través proteínas de anclaje [4].
En muchos casos, la señal reconocida por un receptor desencadena la síntesis de un segundo mensajero que a su vez controla la actividad de las quinasas. Estas quinasas fosforilan los sustratos blanco. Un ejemplo ampliamente conocido de un segundo mensajero es el AMPc, que activa la Proteína Quinasa dependiente de AMPc (PKA) [5,6].
La PKA en Saccharomyces cerevisiae es un tetrámero compuesto por dos subunidades regulatorias y dos subunidades catalíticas. La subunidad regulatoria de unión de cAMP está codificada por un solo gen BCY1, mientras que las subunidades catalíticas están codificadas por los genes TPK1, TPK2 y TPK3 [7]. En ausencia de cAMP, el dímero Bcy1 se une a dos subunidades catalíticas (Tpk) y la enzima está en estado inactivo. La señalización de cAMP-PKA en S. cereviciae [8,9] controla una variedad de procesos celulares esenciales asociados con el crecimiento fermentativo, la entrada en la fase estacionaria, las respuestas al estrés y las vías de desarrollo [10-12]. Dos estímulos principalmente inducen la síntesis de AMPc en la levadura: los azúcares fermentables extracelulares y acidificación intracelular [13]. El aumento que se produce en los niveles de AMPc intracelulares produce la consiguiente activación de PKA.
Considerando el papel pleiotrópico de la vía de señalización de cAMP-PKA en S. cerevisiae, una pregunta importante es cómo se logra la especificidad, es decir, cómo la célula asegura la fosforilación del sustrato correcto cuando la célula está sometida a diferentes estímulos que desencadenan la producción de cAMP como segundo mensajero único.
Aunque las tres isoformas de Tpk demostraron ser funcionalmente redundantes para la viabilidad celular, se han descripto funciones específicas para cada una [14-18]. La especificidad del sustrato para Tpk1, Tpk2 y Tpk3 no parece depender de los residuos presentes en la secuencia alrededor del sitio de fosforilación ni de una diferencia en la constante catalítica (Kcat) para cada isoforma, aunque el sustrato si tiene un papel importante en la activación de la holoenzima [19,20].
Deben existir, por lo tanto, otras estrategias celulares que intervienen para determinar la especificidad de la señalización de PKA en S. cerevisiae.
Esta revisión se centrará en discutir las estrategias que en nuestro laboratorio hemos estudiado durante los últimos años y que contribuyen en determinar la especificidad de la señalización de cAMP-PKA en S. cerevisiae.
Anclaje de PKA a través de proteínas que hacen interacción con la subunidad Regulatoria Bcy1
La subunidad regulatoria (R) es una proteína que posee en el extremo C-terminal dos dominios de unión a AMPc altamente conservados, y un dominio N-terminal más variable que está involucrado en la dimerización y anclaje (DD) [21]. En los mamíferos, existen dos formas de la holoenzima PKA, PKAI y PKAII, formadas por las isoformas RI o RII (subclasificadas en subtipos RIα, RIβ, RIIα y RIIβ) [6,22,23]. Las subunidades R dirigen la holoenzima a compartimentos subcelulares definidos a través de la interacción de su dominio DD con las AKAP (Proteínas de Anclaje de la Quinasa A) [24,25]. Las proteínas AKAP de mamíferos se unen a través de una α-hélice anfipática (14-18 residuos) con alta afinidad a una superficie hidrofóbica en los dominios DD diméricos [26,27].
Las proteínas AKAP se subdividen en tres clases: específicas de RI, específicas de RII o de especificidad dual (se unen tanto a RI como a RII) [28]. Las primeras descriptas fueron aquellas que se unen a la subunidad RII y aunque, las holoenzimas PKAI se encuentran normalmente en la fracción soluble de la célula pueden unirse a las AKAP especificas de RI o aquellas AKAP con especificidad dual. Hay trabajos de revisión que detallan los mecanismos involucrados para restringir espacial y temporalmente los eventos de fosforilación de PKA en mamíferos [24,25,29]
Nuestro laboratorio ha trabajado en los últimos años en encontrar proteínas de anclaje en S. cerevisiae y caracterizar como es el mecanismo molecular que determina la localización de la PKA en este organismo modelo.
La subunidad Bcy1 posee la estructura clásica de dominios de las subunidades R como se puede ver en la Figura 1 A, con los dominios de unión de AMPc y el DD
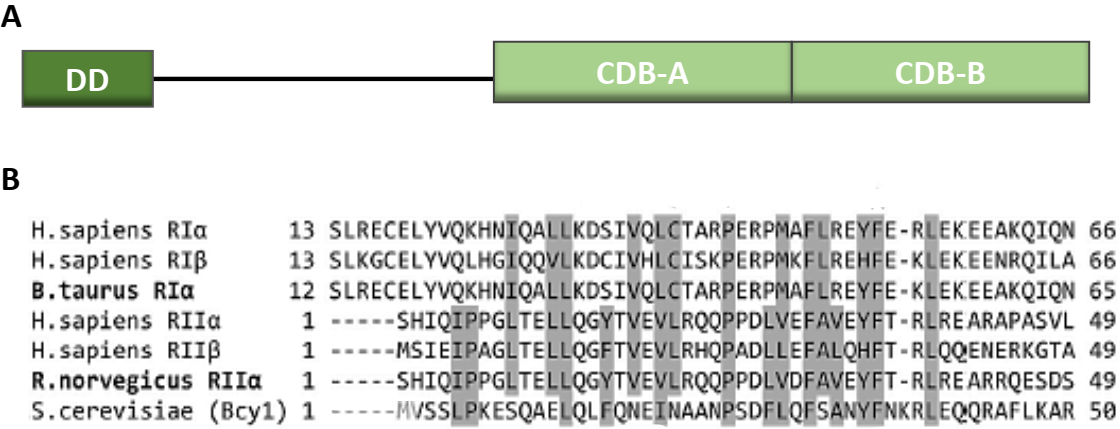
Figura 1: Estructura de dominios de subunidad Bcy1 y secuencia de aa de dominio DD. (A) representación a modo de esquema de los dominios presentes en la subunidad Bcy1 de PKA de S. cerevisiae. D/D: Dominio de Dimerizacion y Anclaje (del Inglés Docking Dimerization Domain); CBD: Dominio de unión de AMPc (del Ingles cAMP Binding Domain). (B) Análisis de secuencias múltiple del dominio DD. Residuos conservados hidrofóbicos marcados en gris. (Adaptado de Bardeci G. N 2016)
Hemos podido demostrar que este mecanismo es diferente al descripto en mamíferos. En contraste con a lo que ocurre con la PKA de eucariotas superiores, la localización de Bcy1 es dinámica y responde a las condiciones nutricionales ambientales [30,31]. Recientemente hemos demostrado que Bcy1 existe como una subunidad R homotetramérica, un estado oligomérico que nunca antes se había reportado en otros organismos [32]. Sin embargo, el extremo N-terminal de Bcy1 exhibe el motivo clásico de hélice-giro-hélice y se conservan los residuos clave para la dimerización presentes en un dominio DD canónico similar a RIIα [32,33] (Fig.1B).
Varias proteínas que hacen interacción con Bcy1 en forma dependiente de su extremo N terminal han sido descriptas. La primera reportada fue Zds1, identificada utilizando un análisis de doble híbrido de levadura, utilizando el dominio N-terminal de Bcy1 como carnada. Se demostró que la proteína Zds1 regula la localización citoplásmica de Bcy1 [34]. También se ha demostrado que la fosforilación de dos grupos de residuos de serina ubicados en la región N-terminal de Bcy1 es necesaria para determinar su localización citoplásmica cuando las células están en un medio de cultivo sin glucosa [34]. La fosforilación del grupo II de serinas aumenta la afinidad de la interacción entre Bcy1 y Zds1, lo que resulta en la retención de Bcy1 en el citoplasma [34].
En nuestro laboratorio, posteriormente, se identificaron a Eno2 (Enolasa II), Hsp60 (Chaperonina mitocondrial) e Ira2 (Proteína Activadora de RAS GTPasa) como proteínas que interactúan con Bcy1 utilizando un abordaje proteómico basado en espectrometría de masa junto con un análisis bioinformático [20]. Se demostró también la relevancia fisiológica de las interacciones de Ira 2 y Hsp60 con Bcy1. Ira2 media la unión de PKA al complejo Ras, conocido regulador de los niveles de AMPc en la levadura [35], mientras que la chaperona Hsp60 facilita la localización de la holoenzima PKA en las mitocondrias y proporciona estabilidad a las subunidades catalíticas [20]. Avanzando en nuestro estudio, se predijo el dominio de interacción en las proteínas de unión a Bcy1 mediante análisis bioinformático. Se demostró que péptidos diseñados a partir de estas predicciones pueden unir Bcy1. Sin embargo, los péptidos de unión muestran características moleculares diferentes al dominio AKAP canónico. En una interacción clásica DD-AKAP, los residuos hidrófobicos son críticos [26,27,36,37]. Sin embargo, en la interacción péptido Ira2-Bcy1 solo los aminoácidos cargados positivamente fueron esenciales [20]. Con posterioridad en el laboratorio se pudo lograr la cristalización del dominio DD, el análisis estructural de los cristales del dominio DD tetramérico de Bcy1 proporciono no solo información sobre los determinantes de la oligomerización de estas subunidades, sino también permitió inferir qué residuos cargados negativamente en la superficie de Bcy1 podrían estar participando en la interacción con el péptido Ira2 (González Bardeci N., comunicación personal).
Transcriptional regulation of the protein kinase A subunits in Saccharomyces cerevisiae
En mamíferos, hay muchos trabajos que describen la regulación transcripcional y post-transcripcional que regulan los niveles de expresión de las subunidades R y C. Se ha demostrado que las isoformas Cα, Cβ, RIα, RIβ, RIIα y RIIβ se expresan según las etapas de desarrollo, de diferenciación y también son específicas de tejido. [38-43]. Varios estudios demostraron que el AMPc tiene efecto a nivel transcripcional y postranscripcional sobre el ARNm, como así también sobre la estabilidad de la proteína de las subunidades después de la disociación de la holoenzima [44-47]. Todos estos mecanismos contribuyen a para que existan diferentes niveles de las distintas isoformas de C y/o R, y se conformen distintas holoenzimas.
En S. cerevisiae, la situación no es muy diferente dado que en distintas condiciones hay expresión diferencial de las distintas isoformas de cada subunidad. Se ha demostrado por estudios transcriptómicos que la expresión de los genes TPK1, TPK2, TPK3 y BCY1 está regulada positivamente en respuesta al estrés térmico y salino, [48-52]. La alta actividad de PKA en la levadura se asocia con varios fenotipos, uno de ellos es la baja resistencia al estrés debido a la represión de genes que están bajo el control del elemento de respuesta al estrés (STRE) y los factores de transcripción Msn2/4. La baja actividad de PKA por el contrario, conduce a fenotipos opuestos que desencadenan la regulación positiva de los genes controlados por STRE y, por lo tanto, resulta en una alta resistencia al estrés [53]. Aparentemente, existe una contradicción ya que la transcripción de las subunidades de PKA se estimula en respuesta al estrés, pero una mayor actividad de PKA conduce a una menor resistencia a esta situación adversa. Al analizar los niveles de proteína Tpk1 frente al estrés térmico hemos observado la falta de aumento en los niveles proteicos, con la resultante falta de correlación con el aumento en la actividad transcripcional del promotor [54, Cañonero Comunicación personal]. Estos resultados indicaron un mecanismo complejo de regulación de la expresión de subunidades de PKA.
Trabajos de nuestro laboratorio demuestran la expresión diferencial de las subunidades de PKA durante diferentes condiciones como el crecimiento en presencia de glucosa o glicerol como fuentes de carbono [55]. Los niveles de expresión de proteínas de cada Tpk son diferentes y cambian de niveles bajos bajo metabolismo fermentativo a niveles más altos durante el cambio a metabolismo no fermentativo asociado con la fase estacionaria [31].
Pautasso y colaboradores han demostrado que los promotores de todos los genes que codifican las subunidades de PKA, TPK1, TPK2, TPK3 y BCY1, están regulados negativamente por la misma actividad de PKA en un mecanismo autorregulatorio isoforma dependiente. Cada subunidad catalítica tiene un efecto negativo sobre la actividad los promotores de cada subunidad. Sin embargo, Tpk2 es la isoforma con mayor efecto inhibidor sobre los promotores TPK1 y TPK3, pero a su vez carece de inhibición hacia su propio promotor [54].
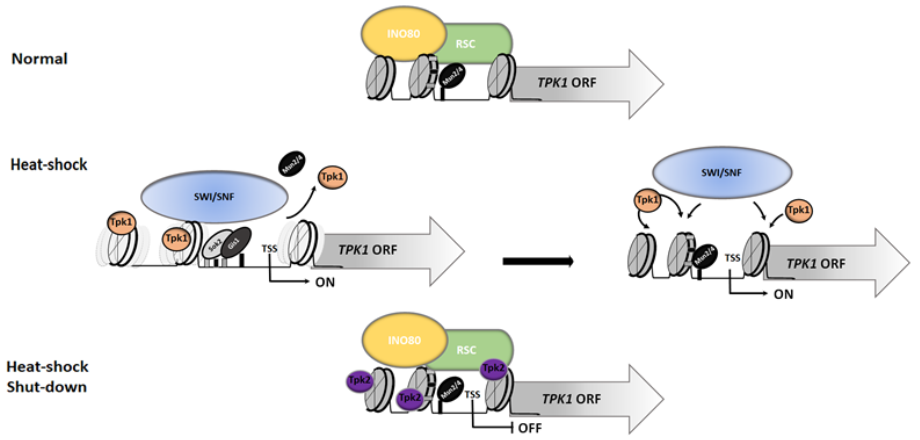
Figura 2: La estructura de la cromatina del promotor TPK1 cambia con el estrés termico. INO80 y RSC son esenciales para mantener una estructura de cromatina represiva del promotor TPK1, los factores de transcripción Msn2/4 están precargados en el elemento STRE más accesible del promotor TPK1 y probablemente en el segundo sitio. SWI/SNF se recluta dependiente de Msn2/4 y remodela la cromatina. Durante el estrés, Msn2/4 se libera del promotor TPK1 y Sok2 se recluta junto con Gis1 [54]. Las isoformas catalíticas Tpk1 y Tpk2 de PKA se unen al promotor TPK1 con patrones de asociación especulares durante el estrés térmico. Tpk1 es necesaria para el remodelado de la cromatina durante el estres y Tpk2 para mantener una conformación represiva. (Adaptado de Reca 2020)
Durante condiciones de estrés, térmico y osmótico, el promotor TPK1 es el único de los promotores de la subunidad PKA que está regulado positivamente. Este promotor presenta tres nucleosomas posicionados que se pierden cuando la célula es sometida a estrés. Este remodelado de la cromatina en el promotor TPK1 correlaciona con la inducción de actividad del promotor y del aumento de los niveles de ARNm de TPK1 [54, 56]. Hemos demostrado que la quinasa Rim15 y los factores de transcripción Msn2/4, Gis1 y Sok2 están involucrados en la regulación positiva de TPK1 durante el estrés térmico [54]. Los remodeladores RSC e INO80 son necesarios para el posicionamiento de los nucleosomas, contribuyendo a la represión de TPK1 en condiciones normales de crecimiento, mientras que SWI/SNF juega un papel en la perdida de nucleosomas y la activación del gen después del estrés térmico [56]. El reclutamiento del complejo SWI/SNF tras el estrés depende de Msn2/4. Algo importante y llamativo que también hemos demostrado es el reclutamiento de las subunidades Tpk1 y Tpk2 al promotor TPK1, las cuales se reclutaron con patrones temporales opuestos tras el estrés térmico [56]. Además, mientras que la actividad catalítica de Tpk1 es necesaria para el remodelado de la cromatina en el promotor TPK1, las actividades de Tpk2 (y Tpk3) mantuvieron una conformación de la cromatina represiva inhibiendo la actividad del promotor [56]. Los resultados de este estudio revelaron un intrincado mecanismo de regulación por retroalimentación de las diferentes subunidades de Tpk sobre el promotor TPK1, única subunidad que es regulada positivamente ante el estrés térmico a nivel transcripcional. Estamos avanzando en los mecanismos de regulación post-transcripcional que determinan la estabilidad del ARNm, y los niveles proteicos de Tpk1.
Síntesis
La respuesta única a un estimulo determinado es lograda por la especificidad de la respuesta del camino de señalización cAMP-PKA. Para lograr esa especificidad existe una interrelación dinámica, concertada y cooperativa de distintos niveles de control. En S. cerevisiae una estrategia celular es restringir la localización de cada subunidad de Tpk en compartimentos subcelulares definidos por su interacción con Bcy1, que interactúa con proteínas anclaje. Aunque este mecanismo se parece al descripto para las subunidades R de mamíferos, las características del dominio de unión en las proteínas que hacen interacción a la subunidad regulatoria de la PKA de levadura son diferentes a las de las AKAPs canónicas. Otro nivel de control en la especificidad cAMP-PKA es la regulación de la expresión de cada subunidad de PKA que demostró ser diferencial durante las fases de crecimiento, durante el crecimiento fermentativo versus respiratorio y frente al estrés térmico o salino. El efecto combinado de cada uno de estos niveles de control, operando simultáneamente, proporciona especificidad y un ajuste fino a la respuesta de la señalización cAMP-PKA.
Referencias:
1. Li G, Qian H (2003) Sensitivity and Specificity Amplification in Signal Transduction. Cell Biochem Biophys 39:45–60. DOI:10.1385/CBB:39:1:45
2. Hynes NE, Ingham PW, Lim WA, et al (2013) Signalling change: signal transduction through the decades. Nat Rev Mol Cell Biol 14:393–398. DOI:10.1038/nrm3581
3. Lee MJ, Yaffe MB (2016) Protein Regulation in Signal Transduction. Cold Spring Harb Perspect Biol 8:. DOI:10.1101/cshperspect.a005918
4. Pawson CT, Scott JD (2010) Signal integration through blending, bolstering and bifurcating of intracellular information. Nat Struct Mol Biol 17:653–658. DOI:10.1038/nsmb.1843
5. Taylor SS, Zhang P, Steichen JM, et al (2013) PKA: lessons learned after twenty years. Biochim Biophys Acta 1834:1271–8. DOI:10.1016/j.bbapap.2013.03.007
6. Zhang P, Kornev AP, Wu J, Taylor SS (2015) Discovery of Allostery in PKA Signaling. Biophys Rev 7:227–238. DOI:10.1007/s12551-015-0170-x
7. Toda T, Cameron S, Sass P, et al (1987) Cloning and characterization of BCY1, a locus encoding a regulatory subunit of the cyclic AMP-dependent protein kinase in Saccharomyces cerevisiae. Mol Cell Biol 7:1371–7
8. Thevelein JM, Bonini BM, Castermans D, et al (2008) Novel mechanisms in nutrient activation of the yeast protein kinase A pathway. Acta Microbiol Immunol Hung 55:75–89. DOI:10.1556/AMicr.55.2008.2.1
9. Conrad M, Schothorst J, Kankipati HN, et al (2014) Nutrient sensing and signaling in the yeast Saccharomyces cerevisiae. FEMS Microbiol Rev 38:254–299. DOI:10.1111/1574-6976.12065
10. Palecek SP, Parikh AS, Kron SJ (2002) Sensing, signalling and integrating physical processes during Saccharomyces cerevisiae invasive and filamentous growth. Microbiology 148:893–907
11. Santangelo GM (2006) Glucose signaling in Saccharomyces cerevisiae. Microbiol Mol Biol Rev 70:253–82. DOI:10.1128/MMBR.70.1.253-282.2006
12. Gancedo JM (2008) The early steps of glucose signalling in yeast. FEMS Microbiol Rev 32:673–704. DOI:10.1111/j.1574-6976.2008.00117.
13. Thevelein JM, Winde JH De (1999) MicroReview Novel sensing mechanisms and targets for the cAMP ± protein kinase A pathway in the yeast Saccharomyces cerevisiae. 33:904–918
14. Robertson LS, Fink GR (1998) The three yeast A kinases have specific signaling functions in pseudohyphal growth. Proc Natl Acad Sci USA 95:13783–7
15. Robertson LS, Causton HC, Young RA, Fink GR (2000) The yeast A kinases differentially regulate iron uptake and respiratory function. Proc Natl Acad Sci 97:5984–5988. DOI:10.1073/pnas.100113397
16. Pan X, Heitman J (2002) Protein kinase A operates a molecular switch that governs yeastpseudohyphal differentiation. Mol Cell Biol 22:3981–93
17. Chevtzoff C, Vallortigara J, Avéret N, et al (2005) The yeast cAMP protein kinase Tpk3p is involved in the regulation of mitochondrial enzymatic content during growth. Biochim Biophys Acta - Bioenerg 1706:117–125. DOI:10.1016/j.bbabio.2004.10.001
18. Palomino A, Herrero P, Moreno F (2006) Tpk3 and Snf1 protein kinases regulate Rgt1 association with Saccharomyces cerevisiae HXK2 promoter. Nucleic Acids Res 34:1427–38. DOI:10.1093/nar/gkl028
19. Mok J, Kim PM, Lam HYK, et al (2010) Deciphering Protein Kinase Specificity Through Large-Scale Analysis of Yeast Phosphorylation Site Motifs. Sci Signal 3:ra12–ra12. DOI:10.1126/scisignal.2000482
20. Galello F, Moreno S, Rossi S (2014) Interacting proteins of protein kinase A regulatory subunit in Saccharomyces cerevisiae. J Proteomics 109:261–75. DOI:10.1016/j.jprot.2014.07.008
21. Taylor SS, Ilouz R, Zhang P, Kornev AP (2012) Assembly of allosteric macromolecular switches: lessons from PKA. Nat Rev Mol Cell Biol 13:646–58. DOI:10.1038/nrm3432
22. Brandon EP, Idzerda RL, McKnight GS (1997) PKA isoforms, neural pathways, and behaviour: making the connection. Curr Opin Neurobiol 7:397–403. DOI:10.1016/s0959-4388(97)80069-4
23. Taylor SS, Yang J, Wu J, et al (2004) PKA: a portrait of protein kinase dynamics. Biochim Biophys Acta 1697:259–69. DOI:10.1016/j.bbapap.2003.11.029
24. Calejo AI, Taskén K (2015) Targeting protein-protein interactions in complexes organized by A kinase anchoring proteins. Front Pharmacol 6:192. DOI:10.3389/fphar.2015.00192
25. Torres-Quesada O, Mayrhofer JE, Stefan E (2017) The many faces of compartmentalized PKA signalosomes. Cell Signal 37:1–11. DOI:10.1016/j.cellsig.2017.05.012
26. Gold MG, Lygren B, Dokurno P, et al (2006) Molecular basis of AKAP specificity for PKA regulatory subunits. Mol Cell 24:383–95. DOI:10.1016/j.molcel.2006.09.006
27. Kinderman FS, Kim C, von Daake S, et al (2006) A dynamic mechanism for AKAP binding to RII isoforms of cAMP-dependent protein kinase. Mol Cell 24:397–408. DOI:10.1016/j.molcel.2006.09.015
28. Jarnaess E, Ruppelt A, Stokka AJ, et al (2008) Dual specificity A-kinase anchoring proteins (AKAPs) contain an additional binding region that enhances targeting of protein kinase A type I. J Biol Chem 283:33708–18. DOI:10.1074/jbc.M804807200
29. Gold MG (2019) Swimming regulations for protein kinase A catalytic subunit. Biochem Soc Trans 47:1355–1366. DOI:10.1042/BST20190230
30. Griffioen G, Anghileri P, Imre E, et al (2000) Nutritional control of nucleocytoplasmic localization of cAMP-dependent protein kinase catalytic and regulatory subunits in Saccharomyces cerevisiae. J Biol Chem 275:1449–56. DOI:10.1074/jbc.275.2.1449
31. Tudisca V, Recouvreux V, Moreno S, et al (2010) Differential localization to cytoplasm, nucleus or Pbodies of yeast PKA subunits under different growth conditions. Eur J Cell Biol 89:339–48. DOI:10.1016/j.ejcb.2009.08.005
32. González Bardeci N, Caramelo JJ, Blumenthal DK, et al (2016) The PKA regulatory subunit from yeast forms a homotetramer: Low-resolution structure of the N-terminal oligomerization domain. J Struct Biol 193:. DOI:10.1016/j.jsb.2015.12.001
33. Griffioen G, Thevelein JM (2002) Molecular mechanisms controlling the localisation of protein kinase A. Curr Genet 41:199–207. DOI:10.1007/s00294-002-0308-9
34. Griffioen G, Branduardi P, Ballarini A, et al (2001) Nucleocytoplasmic Distribution of Budding Yeast Protein Kinase A Regulatory Subunit Bcy1 Requires Zds1 and Is Regulated by Yak1-Dependent Phosphorylation of Its Targeting Domain. Mol Cell Biol 21:511–523. DOI:10.1128/MCB.21.2.511-523.2001
35. Thevelein JM (1994) Signal transduction in yeast. Yeast 10:1753–90
36. Newlon MG, Roy M, Morikis D, et al (2001) A novel mechanism of PKA anchoring revealed by solution structures of anchoring complexes. EMBO J 20:1651–62. DOI:10.1093/emboj/20.7.1651
37. Sarma GN, Kinderman FS, Kim C, et al (2010) Structure of D-AKAP2:PKA RI complex: insights into AKAP specificity and selectivity. Structure 18:155–66. DOI:10.1016/j.str.2009.12.012
38. Oyen O, Sandberg M, Eskild W, et al (1988) Differential regulation of messenger ribonucleic acids for specific subunits of cyclic adenosine 3’,5’-monophosphate (cAMP)-dependent protein kinase by cAMP in rat Sertoli cells. Endocrinology 122:2658–66. DOI:10.1210/endo-122-6-2658
39. Landmark BF, Oyen O, Skålhegg BS, et al (1993) Cellular location and age-dependent changes of the regulatory subunits of cAMP-dependent protein kinase in rat testis. J Reprod Fertil 99:323–34. DOI:10.1530/jrf.0.0990323
40. Skålhegg BS, Rasmussen AM, Taskén K, et al (1994) Cyclic AMP sensitive signalling by the CD28 marker requires concomitant stimulation by the T-cell antigen receptor (TCR/CD3) complex. Scand J Immunol 40:201–8. DOI:10.1111/j.1365-3083.1994.tb03451.x
41. Cadd G, Mcknight GS (1989) Distinct Patterns of CAMP-Dependent Gene Expression in Mouse Brain. 3:71–79
42. Cummings DE, Brandon EP, Planas J V, et al (1996) Genetically lean mice result from targeted disruption of the RII beta subunit of protein kinase A. Nature 382:622–6. DOI:10.1038/382622a0
43. Reinton N, Haugen TB, Ørstavik S, et al (1998) The Gene Encoding the C g Catalytic Subunit of cAMP-Dependent Protein Kinase Is a Transcribed Retroposon. 297:290–297
44. Houge G, Vintermyr OK, Døskeland SO (1990) The expression of cAMP-dependent protein kinase subunits in primary rat hepatocyte cultures. Cyclic AMP down-regulates its own effector system by decreasing the amount of catalytic subunit and increasing the mRNAs for the inhibitory (R) subunits of cAMP-d. Mol Endocrinol 4:481–8. DOI:10.1210/mend-4-3-481
45. Taskén KA, Knutsen HK, Attramadal H, et al (1991) Different mechanisms are involved in cAMPmediated induction of mRNAs for subunits of cAMP-dependent protein kinases. Mol Endocrinol 5:21–28
46. Tasken K, Skilheggs S, Tasken KA, et al (1993) Reciprocal Regulation of mRNA and Protein for Subunits of CAMP-dependent Protein Kinase ( R h and Ca ) by cAMP in a Neoplastic B Cell Line (Reh )* hormones
47. Dahle M, Knutsen H, Taskén K, et al (2001) Cyclic AMP regulates expression of the RI alpha subunit of cAMP-dependent protein kinase through an alternatively spliced 5’ UTR. Eur J Biochem 268:5920–9. DOI:10.1046/J.0014-2956.2001.02542.X
48. Rep M, Krantz M, Thevelein JM, Hohmann S (2000) The transcriptional response of Saccharomyces cerevisiae to osmotic shock. Hot1p and Msn2p/Msn4p are required for the induction of subsets of high osmolarity glycerol pathway-dependent genes. J Biol Chem 275:8290–300
49. Posas F, Chambers JR, Heyman JA, et al (2000) The transcriptional response of yeast to saline stress. J Biol Chem 275:17249–55. DOI:10.1074/jbc.M910016199
50. Gasch AP, Spellman PT, Kao CM, et al (2000) Genomic expression programs in the response of yeast cells to environmental changes. Mol Biol Cell 11:4241–57
51. Causton HC, Ren B, Koh SS, et al (2001) Remodeling of yeast genome expression in response to environmental changes. Mol Biol Cell 12:323–37
52. Castells-Roca L, García-Martínez J, Moreno J, et al (2011) Heat shock response in yeast involves changes in both transcription rates and mRNA stabilities. PLoS One 6:e17272. DOI:10.1371/journal.pone.0017272
53. Estruch F (2000) Stress-controlled transcription factors, stress-induced genes and stress tolerance in budding yeast. FEMS Microbiol Rev 24:469–486. DOI:10.1016/S0168-6445(00)00035-8
54. Pautasso C, Rossi S (2014) Transcriptional regulation of the protein kinase A subunits in Saccharomyces cerevisiae: Autoregulatory role of the kinase A activity. Biochim Biophys Acta - Gene Regul Mech 1839:275–87. DOI:10.1016/j.bbagrm.2014.02.005
55. Galello F, Pautasso C, Reca S, et al (2017) Transcriptional regulation of the protein kinase a subunits in Saccharomyces cerevisiae during fermentative growth. Yeast 34:495–508. DOI:10.1002/yea.3252
56. Reca S, Galello F, Ojeda L, et al (2020) Chromatin remodeling and transcription of the TPK1 subunitof PKA during stress in Saccharomyces cerevisiae. Biochim Biophys acta Gene Regul Mech 1863:194599. DOI:10.1016/j.bbagrm.2020.194599
|
Revista QuímicaViva Número 3, año 19, Diciembre 2020 quimicaviva@qb.fcen.uba.ar |