Hacia la reprogramación de crecimiento bacteriano mediante edición genómica: la bioinformática puesta a prueba
Alfonso Soler Bistue
Grupo Genómica de Vibrio, Laboratorio de Patogenia Microbiana, Instituto de Investigaciones Biotecnológicas “Dr. Rodolfo A. Ugalde” InstitutoTecnológico de Chascomús, CONICET, Universidad Nacional de San Martín, San Martín, BuenosAires, Argentina.
Introducción
Gracias las nuevas tecnologías de secuenciación, aumentó exponencialmente el número de genomas disponibles en bases de datos [1]. En paralelo, el costo de síntesis química de DNA se redujo de manera drástica [2]. Es decir, somos capaces de “leer” y “escribir” DNA de manera cada vez más eficiente. Esta revolución genómica se refleja en la síntesis de la primera célula artificial [3] y el diseño de la forma de vida cultivable de genoma más pequeño, Mycoplasma mycoides (JCV-Syn3.0) [4]. Más aún, ya existen cromosomas sintéticos de levaduras [5] y se ha planificado la construcción de una célula humana de genoma artificial [6]. A pesar de estos ejemplos de bigscience, aún es difícil predecir con exactitud el fenotipo de un organismo en base a la secuencia primaria de su genoma. Las bacterias poseen un genoma haploide de tamaño relativamente pequeño, 1×106-1×107 pares de bases (pb), por lo que han sido fundamentales para investigar esta temática [7]. Nuestro conocimiento de su genoma está evolucionando rápidamente. Hasta hace poco, se lo entendía como una molécula de DNA desnuda flotando estocásticamente en el citoplasma. Hoy sabemos que, por a su asociación a proteínas, se compacta unas 1000 veces para ocupar un 15-30% del volumen celular, con diferentes niveles de organización y plegamiento que permiten una disposición espacial específica que sigue una coreografía precisa a lo largo del ciclo celular [8-13]. Dicha estructura espacial se coordina en simultáneo con procesos fundamentales como son la replicación, la transcripción (a su vez acoplada a la traducción de proteínas) y la segregación del material genético. A pesar de estos avances, se desconoce aún cómo la organización del genoma influye en la fisiología celular y en la evolución de los microorganismos. Un parámetro que caracteriza muy bien ambas variables es la velocidad de crecimiento (interconvertible con el tiempo de generación que es el tiempo entre una división celular y la siguiente). Este es un parámetro clave que refleja la fisiología, la bioquímica y la competitividad celular [14]. La velocidad de crecimiento máxima varía drásticamente entre un microorganismo y otro con tiempos generacionales que pueden ir desde minutos a días o semanas. Por ejemplo, bacterias como Vibrio natrigens, Pectinobacterium carotovorum o Clostridium perfringens se dividen cada 10-15 minutos mientras que Mycobacterium leprae demora 10 días. A pesar de que la fisiología bacteriana es una disciplina con más de un siglo de estudio (por ejemplo las fases del crecimiento bacteriano se describieron ya en 1909[15]) aún se desconoce qué factores genéticos moldean la velocidad máxima de crecimiento de una especie.
Organización del genoma bacteriano
Tras la secuenciación de más de 30.000 genomas emerge una nueva imagen del cromosoma bacteriano y su evolución [1]. En contraste con las células eucariotas, el genoma bacteriano posee un contenido genético muy variable. Su evolución ocurre principalmente por transferencia horizontal de genes. Así, dos aislamientos de la misma especie bacteriana pueden tener un 20% de divergencia en contenido génico. A pesar de tal flexibilidad, las bacterias muestran una organización muy estricta a nivel de secuencia del genoma conservada filogenéticamente [16,17]. Universalmente, las bacterias presentan único origen de replicación (oriC) desde donde se inicia la duplicación del DNA bidireccionalmente hasta la región terminal (ter) en el extremo opuesto del cromosoma. Así, el genoma se organiza longitudinalmente a lo largo de un eje ori-ter que divide al DNA en dos replichores de un tamaño similar (Figura 1). Existen pocos estudios sobre cómo esta estructura influye en la fisiología celular y en la evolución del genoma [18,-26].
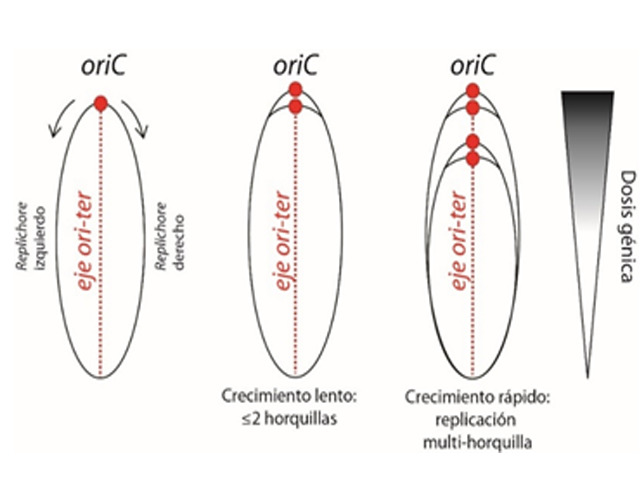
Figura 1: Estructura del genoma bacteriano. Izquierda y centro: la replicación comienza en el único oriC y procede (flechas) a lo largo de dos replichores simétricos. Cuando las bacterias crecen lentamente el genoma tiene tiempo para replicar el cromosoma antes de la división celular. Derecha: durante el crecimiento rápido, se superponen ciclos de replicación ya que el tiempo de división es más corto que el que requiere la duplicación del DNA. Esto genera un gradiente decreciente de dosis génica a lo largo de ambos replichores (triangulo) (Modificado [22]).
Varios trabajos muestran que esta organización influye fuertemente en la fisiología y en la evolución bacteriana. Por ejemplo, las inversiones de segmentos de DNA ocurren con una frecuencia relativamente alta. Algunas abarcan cientos de Kpbs. En el caso de las grandes inversiones, generalmente solo son viables aquellas que ocurren simétricamente respecto del eje ori-ter. De esta manera ambos replichores mantienen un tamaño similar [16,20,27]. También se ha observado que los genes esenciales están co-orientados con la replicación. Motivos de secuencia implicados en la reparación y la segregación se encuentran más frecuentemente en la hebra líder de la replicación [16,17]. Más específicamente, se han descrito también patrones en el genoma que podrían influir en la velocidad máxima de crecimiento bacteriana.
Localización genómica y expresión
La Genómica Comparada revela que la posición de ciertos genes en el cromosoma está ampliamente conservada. Los genes que codifican las proteínas asociadas al nucleoide, los factores sigma de la RNA polimerasa y las topoisomerasas tienen una localización conservada que se correlaciona además con su momento de expresión en las fases de crecimiento bacteriano: los genes que se expresan en la fase exponencial se localizan cerca del oriC mientras que aquellos que se expresan durante la fase estacionaria se ubican cerca de la región ter [28,29]. Paralelamente, se descubrió que los genes que codifican para la maquinaria de expresión de la información genética como la RNAP, los RNAt, los RNAr y las proteínas ribosomales (PR) tienden a ubicarse cerca del oriC. Dicha correlación se observa solo en bacterias de crecimiento rápido lo que sugiere fuertemente que la localización de estos genes influiría en la fisiología celular [30,31]. Se especuló entonces que esta localización traería el beneficio de aumentar mayor dosis génica durante la replicación (Figura 1). Esto es particularmente cierto en bacterias de crecimiento rápido como Escherichia coli o Vibrio cholerae que se dividen en un tiempo menor al requerido para la replicación del DNA. En consecuencia, inician la replicación más de una vez por división celular, fenómeno llamado replicación multi-horquilla (Figura 1). Así, las células hijas heredan un cromosoma parcialmente replicado evitándose la generación de células anucleadas. Esto conduce a que las regiones próximas al oriC posean 4, 8 o más copias que los genes en la región terminal lo que genera un gradiente de dosis y expresión a lo largo de ambos replichores (Figura 1). Así, la posición cromosómica puede condicionar el nivel de expresión que puede alcanzar un gen. Los genes para la maquinaria de traducción y trascripción están entonces en mayor dosis durante la fase exponencial de crecimiento, el momento en que más se los requiere.
Estudios recientes demuestran que la expresión de un fenotipo no depende solo de la presencia o ausencia de su(s) gen(es) codificantes sino también del contexto genómico en el que se encuentra(n). Por ejemplo, para que el programa de esporulación funcione correctamente en Bacillus subtilis, dos de los genes reguladores de la cadena de fosfotransferencia deben estar en extremos opuestos del cromosoma [23]. Análogamente, la localización genómica del operón comCDE en Streptocuccus pneumoniae es crucial para que la bacteria pueda inducir su programa de competencia natural frente a lesiones en el DNA [24]. De la misma manera, recientemente describimos que la posición de los genes que codifican para las PR impactan fuertemente en la velocidad de crecimiento, la aptitud adaptativa (fitness) y la infectividad de V. cholerae (ver más adelante) [21,22]. Otro caso paradigmático es el gen fis de E. coli, cuya relocalización reduce su fitness por la modificación simultánea de diversos fenotipos [19]. Los trabajos citados apuntan, en líneas generales, a dos mecanismos principales por los cuales la posición en el cromosoma afecta al fenotipo: por un cambio de dosis del gen en cuestión [18] o bien, debido a la alteración de su localización física [19,32].
Sintonía fina de la velocidad de crecimiento: relocalización del principal locus de PR en V. cholerae
Recientemente, investigamos experimentalmente cómo la estructura del genoma influye en la fisiología celular. Como modelo usamos V. cholerae, un patógeno de relevancia local y global. Como agente etiológico del cólera, afecta anualmente a entre 1.4 y 4.3 millones de personas causando entre 21.000 y 143.000 muertes [33]. Desde 2016 se desarrolla en Yemen la epidemia más grande de la historia con más de un millón de infectados y 2200 muertes. En nuestra región se desarrolló una gran epidemia a fines de los ’90 y más recientemente, fue célebre la ocurrida en Haití en 2010 [33,35]. V. cholerae es un habitante de aguas costeras con un ciclo de vida que alterna entre un período de amplificación, durante la infección, y una fase de persistencia en el ambiente [36]. El crecimiento rápido se asocia con el período de amplificación, mientras que el crecimiento lento se asemeja a las condiciones ambientales de la fase de persistencia.
Además de la importancia sanitaria, V. cholerae es particularmente interesante porque, con un tiempo generacional de 17 minutos, está entre las bacterias de crecimiento más rápido conocidas. Este rasgo correlaciona con una estructura genómica que sigue las tendencias observadas en organismos de crecimiento veloz: alto número de operones ribosomales y RNAts así como genes de PR y RNAP cercanos al oriC del cromosoma principal (oriC1). Otro rasgo interesante es que es un microorganismo que rompe con otro paradigma. Habitualmente se piensa que las bacterias poseen un solo cromosoma. Sin embargo, al menos un 10% de las bacterias poseen al menos 2 cromosomas. V. cholerae es uno de los modelos mejor estudiados de bacterias con múltiples cromosomas. Posee un cromosoma principal de ~3Mpb que aloja a la mayoría de los genes esenciales y un cromosoma secundario de ~1Mpb con mayoría de genes de función desconocida. La replicación de ambos cromosomas está perfectamente coordinada: el cromosoma principal comienza su replicación y cuando alcanza los 2/3 del replichore se gatilla el oriC del cromosoma secundario. De esta manera la replicación de ambos cromosomas concluye simultáneamente.
Para entender los vínculos entre la estructura del genoma y la fisiología celular, nos focalizamos en el locus s10-spec-α (S10), un sitio de 13,2 Kpb que alberga la mitad de los genes de PR. Para ello, desarrollamos herramientas de recombinación basadas en fagos lamboides que permiten una manipulación sin precedentes del genoma bacteriano [21]. Gracias a este desarrollo, generamos un set de cepas isogénicas donde S10 fue relocalizado a distintos sitios del genoma de manera de colocar el locus a diferentes distancias del oriC1 e incluso en el cromosoma secundario (Figura 2a).
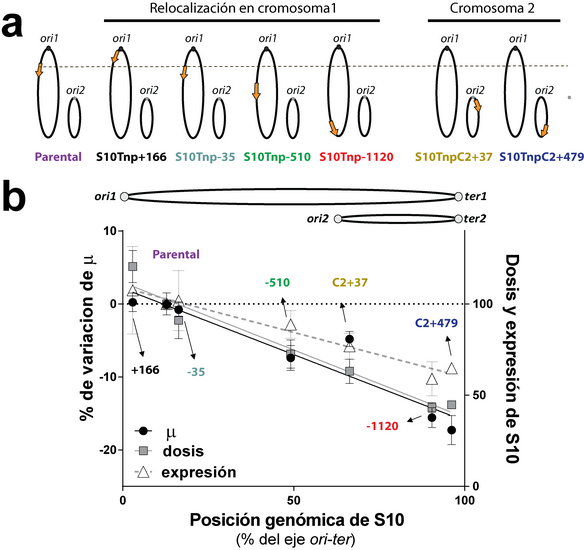
Figura 2: La posición genómica de S10 determina la dosis del locus su expresividad y determina la velocidad de crecimiento (µ) de V. cholerae. a) Mediante edición del genoma se reubicó S10 (flecha naranja) en diferentes sitios del genoma generándose una colección de cepas que permite estudiar la relación localización-fenotipo. b) Determinación de la velocidad de crecimiento (negro, eje izquierdo), la dosis (gris, ordenada derecha) y la expresión (blanco, eje derecho) de S10 en las diferentes cepas. Se observa una fuerte correlación de las 3 variables con la posición genómica de S10 (abscisa) (Modificado[22]).
Para nuestra sorpresa, ninguna de estas derivadas resultó afectada en su viabilidad ni en su morfología macroscópica o microscópica. Es decir que la localización genómica del locus es flexible. Luego, analizamos esta colección de cepas en condiciones óptimas de crecimiento. Descubrimos que la velocidad máxima de crecimiento de cada cepa, la dosis génica y la expresión de S10 variaban en proporción inversa a la distancia entre S10 y oriC1 (Figura 2b) [21].
En medio mínimo, cuando no ocurre la replicación multi-horquilla (Figura 1), la velocidad de crecimiento resultó independiente de la posición genómica del locus. Todos estos experimentos sugerían fuertemente que la localización de S10 determina la velocidad máxima de crecimiento al proveer una dosis de S10 que maximiza la expresión de estas PR. Sin embargo, al relocalizar S10 se altera simultáneamente la dosis y la sub-localización celular del locus. De esta manera, si la reubicación de estos genes es deletérea por motivos ajenos a la dosis génica (por ejemplo, si la inserción per se es perjudicial o si S10 es requerido en cis), los efectos observados serían similares. Para clarificar esta cuestión, se construyeron mutantes con dos copias de S10 lejos del oriC1. De esta manera se generaron cepas donde, si bien se alteró la localización espacial de S10, se mantiene su dosis génica. Estas cepas complementaron el defecto en la velocidad de crecimiento demostrando que la posición genómica del locus es importante para la fisiología celular dado que provee la dosis de S10 esencial para la fisiología bacteriana [21]. Por otro lado, experimentos de infección en Drosophila melanogaster, la mosca de la fruta, mostraron que la relocalización de S10 reduce de manera drástica la infectividad de V. cholerae lo que sugiere que la posición de estos genes es importante en las condiciones ambientales que afronta el patógeno [21].
Con el objeto de revelar efectos independientes de la dosis estudiamos en detalle estas cepas en medio mínimo empleando ensayos de competencia [22]. Estos son más sensibles que las curvas de crecimiento para determinar reducciones en el fitness [37]. Así detectamos que cuando desplazamos S10 a la región ter del cromosoma 1 o del cromosoma 2, el fitness de la bacteria se reduce de manera muy significativa. Esto sugería que se trataba de efectos independientes de la dosis. Sin embargo, para nuestra sorpresa, la adición de una segunda copia de S10 restituyó el fitness de la bacteria. Mediante la secuenciación masiva de estas cepas hallamos, que la relocalización de S10 produce una reducción del 30% de la dosis génica de S10 en ausencia de replicación multi-horquilla. Los cambios en la dosis correlacionan muy ajustadamente con los efectos observados en el fitness de las cepas. En contraste con lo que proponían los estudios bioinformáticos, la posición genómica de las PR es fundamental para proveer la dosis génica óptima independientemente de la presencia de la replicación multi-horquilla [22]. En el caso de V. cholerae implica que la posición de estos genes impacta en ambos estadios de su ciclo de vida, tanto en la fase infectiva de la bacteria (crecimiento rápido) como durante su persistencia en el ambiente (crecimiento lento). Pero además esto sugiere que el posicionamiento genómico de los genes que codifican para el flujo de la información genética podría influenciar la evolución de las bacterias de crecimiento lento. Así esta clase de aproximación puede emplearse para reprogramar la velocidad del crecimiento bacteriano. Por otro lado, la dosis de S10 podría ser limitante del crecimiento bacteriano. O dicho de otra manera, quizás aumentando su dosis podríamos acelerar su crecimiento. Para probar esta posibilidad creamos cepas con 2, 3 y hasta 4 copias del locus (Figuras 3a y 3b)
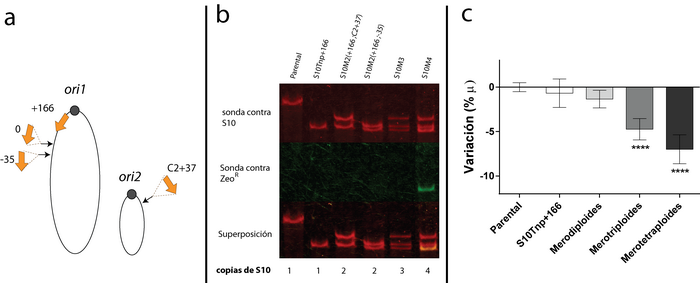
Figura 3: Impacto del aumento de la ploidía de S10 en V. cholerae. a) Se insertaron varias copias S10 (flechas naranjas) dentro del genoma de la cepa + 166. Los sitios de inserción, dibujados como flechas negras y las coordenadas (en pares de kilobases desde la ubicación original), se muestran sobre una representación de los cromosomas de V. cholerae. b) SouthernBlotcon DNA genómico digerido con una enzima de restricción de las cepas indicadas en el panel superior. Las sondas se dirigieron a S10(rojo) o a un marcador asociado(ZeoR) cuando los tamaños obtenidos eran similares (verde). Los cambios en el genotipo se evidencian por el cambio de tamaño de S10 al moverse (parental vs S10Tnp + 166). Luego se agrega la segunda y tercera copia de S10Tnp-35 y S10TnpC2+37 (flechas negras). La cuarta copia de S10 viene de una cepa que origina un fragmento de restricción con un tamaño similar al del alelo proveniente de +166. Sin embargo, puede distinguirse por una señal de sonda S10 incrementada y la sonda verde. La ploidía de S10 para cada cepa se muestra en el panel inferior. c) El efecto de ploidía S10 en la velocidad de crecimiento se cuantificó promediando la tasa de crecimiento(μ) obtenida en varios experimentos independientes para cada cepa mutante y normalizándolo a μ de la cepa parental. Los resultados se expresan como porcentaje de la variación (% μ) con respecto a la cepa parental(Modificado[22].
Es decir, creamos cepas merodiploides, merotriploides y merotetrapliodes para S10. Las cepas merodiploides mostraron un crecimiento similar al de la cepa parental indicando que un pequeño exceso de S10 no es perjudicial. Mientras tanto, la adición de una tercera y cuarta copia del locus impactó en el crecimiento celular, ya que las cepas merotriploides y la merotetraploide mostraron reducciones significativas de su velocidad de crecimiento con respecto a la parental (Figura 3c).
Por lo tanto, la velocidad de crecimiento no puede mejorarse mediante el aumento de la dosis de S10, lo que demuestra que el número de copias del locus no es limitante para el crecimiento bacteriano. La célula puede tolerar los efectos perjudiciales putativos de una copia extra, pero aumentar la ploidíade S10 aún más, como en las cepas merotriploide y merotetraploide, perjudica la fisiología celular (Figura 3c). Estos resultadossumados a la flexibilidad en la localización genómica de S10, sugiere que el locus, a lo largo de la evolución, derivó a la posición genómica óptima para el crecimiento de V. cholerae.
Remarcas finales
A lo largo de nuestro trabajo mostramos un efecto claro del reposicionamiento de S10 sobre la fisiología, la infectividad y la competitividad de V. cholerae. Creemos que este locus es la punta del iceberg de muchos más grupos de genes por descubrir. También las herramientas experimentales que desarrollamos y el marco teórico que empleamos servirán para poner a prueba muchas hipótesis que provienen de la genómica comparada pero que no se han puesto a prueba experimentalmente. Ya hemos comenzado la búsqueda de nuevos casos, por ejemplo, ya estamos estudiando el rol de la localización de los genes de la RNA polimerasa bacteriana. Además, estamos iniciando el estudio de otros modelos bacterianos, en particular, nos interesa acelerar el tiempo generacional de bacterias de crecimiento lento. La aplicación de esta metodología a otras bacterias y a otros genes proporcionará información sobre las reglas de la organización del genoma. Además, comprender los factores genómicos que afectan la velocidad de crecimiento permitirá reprogramar el crecimiento bacteriano, ayudar a predecir el comportamiento de sistemas biológicos más complejos y desarrollar mejores modelos teóricos [38,39]. Nuestros estudios contribuirán al desarrollo del diseño de genomas, la bioingeniería y la biotecnología en el contexto del surgimiento de la Biología Sintética y de la invención de nuevas herramientas para la edición de genomas.
Agradecimientos
Agradezco a las estudiantes de mi grupo Belén Bordignon, Leila Bartrolí y Julieta Viglino por leer cuidadosamente este artículo.
Referencias:
1. Land M. et al. (2015) Insights from 20 years of bacterial genome sequencing. Functional & integrative genomics 15:141-161, doi:10.1007/s10142-015-0433-4
2. Kosuri S, Church GM (2014) Large-scale de novo DNA synthesis: technologies and applications. Nature methods 11: 499-507, doi:10.1038/nmeth.2918
3. Gibson DG et al. (2010) Creation of a bacterial cell controlled by a chemically synthesized genome. Science 329: 52-56, doi:10.1126/science.1190719.
4. Hutchison CA 3rd et al. (2016) Design and synthesis of a minimal bacterial genome. Science 351, aad6253, doi:10.1126/science.aad6253).
5. Annaluru N et al. (2014) Total synthesis of a functional designer eukaryotic chromosome. Science 344: 55-58, doi:10.1126/science.1249252.
6. Boeke JD et al. (2016) GENOME ENGINEERING. The Genome Project-Write. Science 353: 126-127, doi:10.1126/science.aaf6850
7. Robinson NP, Bell SD (2005) Origins of DNA replication in the three domains of life. The FEBS journal 272: 3757-3766, doi:10.1111/j.1742-4658.2005.04768.x.
8. Le TB, Laub MT (2014) New approaches to understanding the spatial organization of bacterial genomes. Current opinion in microbiology 22: 15-21, doi:10.1016/j.mib.2014.09.014-
9. Reyes-Lamothe R, Nicolas E, Sherratt DJ (2012) Chromosome replication and segregation in bacteria. Annual review of genetics 46: 121-143, doi:10.1146/annurev-genet-110711-155421.
10. Dorman CJ (2013) Genome architecture and global gene regulation in bacteria: making progress towards a unified model? Nature reviews. Microbiology 11: 349-355, doi:10.1038/nrmicro3007.
11. Toro E, Shapiro L (2010) Bacterial chromosome organization and segregation. Cold Spring Harbor perspectives in biology 2, a000349, doi:10.1101/cshperspect.a000349.
12. Campos M, Jacobs-Wagner C (2013) Cellular organization of the transfer of genetic information. Current opinion in microbiology 16: 171-176, doi:10.1016/j.mib.2013.01.007.
13. Lioy V S et al. (2018) Multiscale structuring of the E. coli chromosome by nucleoid-associated and condensin proteins. Cell 172: 771-783 e718, doi:10.1016/j.cell.2017.12.027.
14. Monod J (1949) The Growth of Bacterial Cultures. Annual Reviews in Microbiology 3: 371-3949.
15. Lane-Claypon JE (1909) Multiplication of Bacteria and the Influence of Temperature and some other Conditions thereon. The Journal of hygiene 9: 239-248 (1909).
16. Rocha EP (2008) The organization of the bacterial genome. Annual review of genetics 42: 211-233, doi:10.1146/annurev.genet.42.110807.091653.
17. Touchon M, Rocha EP (2016) Coevolution of the Organization and Structure of Prokaryotic Genomes. Cold Spring Harbor perspectives in biology 8, a018168, doi:10.1101/cshperspect.a018168.
18. Slager J, Veening JW (2016) Hard-Wired Control of Bacterial Processes by Chromosomal Gene Location. Trends in microbiology 24: 788-800, doi:10.1016/j.tim.2016.06.003.
19. Gerganova V et al. (2015) Chromosomal position shift of a regulatory gene alters the bacterial phenotype. Nucleic acids research 43: 8215-8226, doi:10.1093/nar/gkv709.
20. Esnault E, Valens M, Espeli O, Boccard F (2007) Chromosome structuring limits genome plasticity in Escherichia coli. PLoS genetics 3, e226, doi:10.1371/journal.pgen.0030226.
21. Soler-Bistue A et al. (2015) Genomic location of the major ribosomal protein gene locus determines Vibrio cholerae global growth and infectivity. PLoS genetics 11, e1005156, doi:10.1371/journal.pgen.1005156.
22. Soler-Bistue A, Timmermans M. Mazel D (2017) The proximity of ribosomal protein genes to oriC enhances Vibrio cholerae fitness in the absence of multifork replication. mBio 8, doi:10.1128/mBio.00097-17.
23. Narula J et al. (2015) Chromosomal arrangement of phosphorelay genes couples sporulation and DNA replication. Cell 162: 328-337, doi:10.1016/j.cell.2015.06.012 (2015).
24. Slager J, Kjos M, Attaiech L, Veening JW (2014) Antibiotic-induced replication stress triggers bacterial competence by increasing gene dosage near the origin. Cell 157: 395-406, doi:10.1016/j.cell.2014.01.068.
25. Fitzgerald S et al. (2015) Re-engineering cellular physiology by rewiring high-level global regulatory genes. Scientific reports 5, 17653, doi:10.1038/srep17653.
26. Sobetzko P, Glinkowska M, Travers A. Muskhelishvili G (2013) DNA thermodynamic stability and supercoil dynamics determine the gene expression program during the bacterial growth cycle. Molecular bioSystems 9: 1643-1651, doi:10.1039/c3mb25515h.
27. Repar J, Warnecke T (2017) Non-Random inversion landscapes in prokaryotic genomes are shaped by heterogeneous selection pressures. Molecular biology and evolution 34: 1902-1911, doi:10.1093/molbev/msx127.
28. Muskhelishvili G, Travers A (2014) Order from the order: how a spatiotemporal genetic program is encoded in a 2-D genetic map of the bacterial chromosome. Journal of molecular microbiology and biotechnology 24: 332-343, doi:10.1159/000368852.
29. Sobetzko P, Travers A, Muskhelishvili G (2012) Gene order and chromosome dynamics coordinate spatiotemporal gene expression during the bacterial growth cycle. Proceedings of the National Academy of Sciences of the United States of America 109: E42-50, doi:10.1073/pnas.1108229109.
30. Couturier E. Rocha EP (2006) Replication-associated gene dosage effects shape the genomes of fast-growing bacteria but only for transcription and translation genes. Molecular microbiology 59: 1506-1518, doi:10.1111/j.1365-2958.2006.05046.x.
31. Vieira-Silva S, Rocha EP (2010) The systemic imprint of growth and its uses in ecological (meta)genomics. PLoS genetics 6, e1000808, doi:10.1371/journal.pgen.1000808.
32. Bryant JA, Sellars LE, Busby SJ, Lee DJ (2014) Chromosome position effects on gene expression in Escherichia coli K-12. Nucleic acids research 42: 11383-11392, doi:10.1093/nar/gku828.
33. Harris JB, LaRocque RC, Qadri F, Ryan, ET, Calderwood SB (2012) Cholera. Lancet 379: 2466-2476, doi:10.1016/S0140-6736(12)60436-X.
34. Domman, D et al. (2017) Integrated view of Vibrio cholerae in the Americas. Science 358: 789-793, doi:10.1126/science.aao2136.
35. Hu D et al. (2016) Origins of the current seventh cholera pandemic. Proceedings of the National Academy of Sciences of the United States of America 113: E7730-E7739, doi:10.1073/pnas.1608732113.
36. Nelson EJ, Harris J B, Morris J G Jr., Calderwood, SB, Camilli A (2009) Cholera transmission: the host, pathogen and bacteriophage dynamic. Nature reviews. Microbiology 7: 693-702, doi:10.1038/nrmicro2204.
37. Concepcion-Acevedo J, Weiss H N, Chaudhry WN, Levin BR (2015) Malthusian Parameters as Estimators of the Fitness of Microbes: A Cautionary Tale about the Low Side of High Throughput. PloS one 10, e0126915, doi:10.1371/journal.pone.0126915.
38. Karr JR et al. (2012) A whole-cell computational model predicts phenotype from genotype. Cell 150: 389-401, doi:10.1016/j.cell.2012.05.044.
39. Scott M, Hwa T (2011) Bacterial growth laws and their applications. Current opinion in biotechnology 22: 559-565, doi:10.1016/j.copbio.2011.04.014.
|
Revista QuímicaViva Número 2, año 17, Agosto 2018 quimicaviva@qb.fcen.uba.ar |