Descubrimiento de las propiedades oncogénicas de los antígenos MAGE
Martin Monte
Laboratorio de Oncología Molecular, Departamento de Química Biológica e IQUIBICEN (UBA-CONICET), Facultad de Ciencias Exactas y Naturales, Universidad de Buenos Aires.
Resumen
Desde su descubrimiento como antígenos en células de melanoma humano, la interpretación de las proteínas MAGE (Melanoma Antigen Genes) ha variado mucho. Se ha pasado de verlas como moléculas promisorias para desarrollar vacunas anti-cáncer a concluir que su expresión está asociada a la promoción tumoral y probablemente ambas características sean verdaderas. En este artículo se revisan los principales hitos en la caracterización funcional de proteínas MAGE. Comenzando por el atractivo modelo de proteínas expresadas en tumores que inhiben al oncosupresor p53, se han observado y demostrado distintos mecanismos por los cuales diferentes proteínas MAGE contribuyen con el fenotipo tumoral. Comienzo con una revisión histórica de las proteínas MAGE para luego detenerme en los principales descubrimientos que llevaron a caracterizar estas proteínas como potenciales oncogenes.
Palabras clave: MAGE, vacunas, oncosupresores, oncogenes, ubiquitinación
Discovering the oncogenic properties of MAGE antigens
Summary
Since its discovery as antigens in human melanoma cells, the view of MAGE proteins (Melanoma Antigen GEnes) has greatly varied. They switched from promising antigens to develop anti-cancer vaccines to conclude that its expression is associated with tumor promotion, and probably both characteristics are true. Starting with the attractive model of proteins expressed in tumors that inhibit the p53 oncosuppressor, different mechanisms have been demonstrated by which different MAGE proteins contribute to the tumor phenotype. This article begins with a historical review of the MAGE proteins and then stops at the main discoveries that led to characterize these proteins as potential oncogenes.
Keywords: MAGE, vaccines, oncosuppressors, oncogenes, ubiquitination
Descubrimiento de los genes MAGE: una promesa para la inmunoterapia
La historia de los antígenos tumorales de la familia MAGE (Melanoma Antigen Genes) tiene origen reciente. Si bien la búsqueda de antígenos tumorales que fueran reconocidos por el sistema inmune propio (autologous typing), estaba en desarrollo desde la segunda mitad de los años ‘70 [1], fue recién en 1991, cuando gracias a progresos tecnológicos en este campo se descubrieron los antígenos MAGE. Estos surgieron como fruto de una colaboración entre inmunólogos del Memorial Sloan-Kettering Cancer Center (NYC, USA), de la Universidad de Mainz (Mainz, Alemania) y del Ludwig Institute for Cancer Research (Bruselas, Bélgica) quienes identificaron tres proteínas del melanoma de un paciente (MZ-2) capaces de activar sus propios linfocitos T [2]. Estos antígenos de melanoma humano se denominaron Mage1, Mage2 y Mage3 y sus principales características eran: su expresión en melanoma y en células sanas de la línea germinal masculina, su ausencia en células somáticas sanas, y su alta similitud de secuencia. Este singular perfil de expresión en células tumorales y testículo resultó ser posteriormente compartido con otras proteínas que se agruparon en la familia de Cancer/Testis Antigens (CTA) [3].
Los siguientes 10 años de estudio de proteínas MAGE fueron obviamente focalizados hacia el campo de la inmunología tumoral y el desarrollo de vacunas. El perfil de expresión de proteínas MAGE era muy promisorio para el desarrollo de vacunas, ya que las únicas células sanas que presentan estos antígenos están al resguardas por a la barrera hemato testicular. No obstante la realización de varios ensayos clínicos utilizando péptidos derivados de Mage3, los resultados fueron variados y no muy satisfactorios, aunque actualmente se buscan variantes que puedan arrojar mejores resultados con nuevas propuestas de ensayos clínicos [4].
Paralelamente a su desarrollo en el campo de la inmunología tumoral, surgieron otros datos fundamentales para el estudio de proteínas MAGE. En principio, la expresión de genes MAGE no está restringida a melanoma, sino que también fueron detectados en tumores de distinto origen histológico. Más aún, dentro de un mismo tipo histológico, el tumor puede expresar o no genes MAGE y si los expresa pueden ser distintos miembros de la familia [5]. Otro avance importante fue la observación de que, detrás de los 3 genes MAGE encontrados en el trabajo del grupo de Thierry Boon [2], existía una gran familia de genes similares. Esta gran familia MAGE consta hoy de más de 60 genes con características propias [6]. Todos los miembros de esta familia poseen un dominio característico llamado “dominio de homología MAGE” (MHD, MAGE Homology Domain). Este dominio es de aproximadamente 200 amino ácidos y en muchos casos cubre casi dos tercios de la proteína. La estructura de la familia se ha dividido en dos grandes grupos: MAGE-I y MAGE-II. Esta característica es muy importante, ya que solo los genes miembros del grupo MAGE-I mantienen su expresión específica en tumores y testículo (familia CTA), mientras que los MAGE-II poseen expresión celular ubicua y un MHD menos conservado. A su vez, los genes MAGE-I mapean únicamente en sectores discretos (clusters A, B, C) del cromosoma X denominados MAGE-A (11 genes), MAGE-B (10 genes), MAGE-C (3 genes) [5] (Figura 1), mientras que los genes del grupo MAGE-II localizan en el cromosoma X y en otros cromosomas. Luego de esta nueva organización, las proteínas Mage1, Mage2 y Mage3 descubiertas en 1991, pasaron a denominarse MageA1, MageA2 y MageA3, ya que eran parte del grupo MAGE-A. También se evidenció que el mecanismo epigenético que causaba la ausencia de expresión en células somáticas normales era la metilación de sus promotores [7].
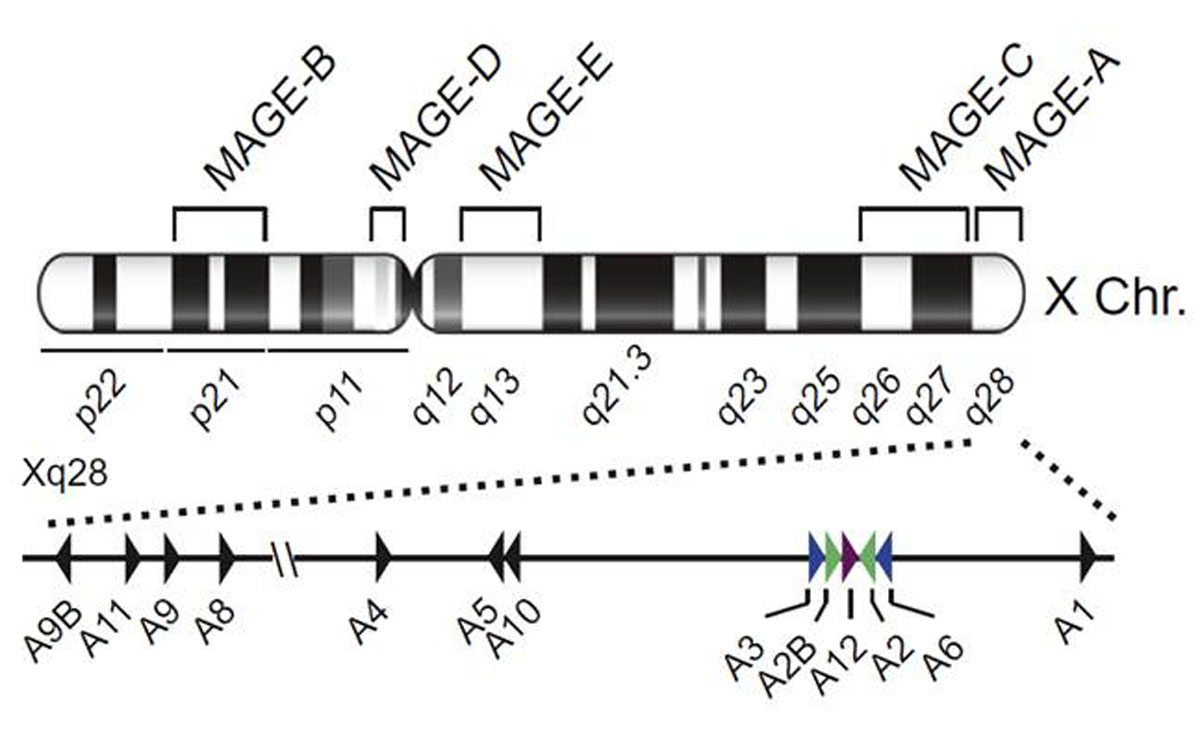
Figura 1: Esquema de la posición de los clusters de genes MAGE en el cromosoma X humano. En la figura de observan los clusters A, B y C (MAGE-I) así como los clusters D y E (MAGE-II). También se muestra en detalle el posicionamiento de los genes MAGE-A dentro de su cluster génico. Adaptado de [4].
A principios de los años 2000, la visión generalizada sobre los genes MAGE sugería que su expresión podría indicar buen pronóstico ya que convertían a las células tumorales que los expresaban en potenciales blancos del sistema inmune. Además, dada la homología de secuencia, se preveía que estas proteínas desconocidas tendrían, muy probablemente, funciones redundantes. Existían pocos sistemas de detección confiable; los pocos anticuerpos desarrollados contra alguna proteína MAGE específica, hoy se sabe que tenía reacción cruzada con otras proteínas MAGE similares pero hasta el momento no descubiertas.
Caracterización funcional de las proteínas MAGE-A: la relación con p53
A mediados de los 2000, me encontré estudiando el interesante mundo de los genes MAGE porque que había identificado uno de ellos, MageA6, como proteína asociada a GTSE-1, un regulador del oncosupresor p53 [8, 9]. Los primeros ensayos indicaban que MageA6 contribuía con la inhibición transcripcional de p53 y sugería que una proteína de expresión tumoral podía funcionar como inhibidor de este supresor tumoral central. Las preguntas en ese momento de total desconocimiento sobre la función de las proteínas MAGE eran tantas que me llevaron a estudiar varias de ellas. Una de las mayores sorpresas fue que algunas proteínas MAGE no compartían la misma localización celular a pesar de la homología de secuencia. Esto sin dudas sugería que no tenían funciones redundantes y que pequeñas zonas no conservadas (probablemente en su región N-terminal) podían dar características distintivas a estas proteínas. Desde ese momento, parte de mi proyecto de estudio se focalizaría en comprender su función celular y si las proteínas MAGE eran funcionalmente redundantes. En el primer trabajo demostramos que la proteína MAGE-A que más potentemente inhibía p53 era MageA2 y que el mecanismo involucraba la unión de MageA2 a p53 y el reclutamiento de desacetilasas de histonas (HDACs) como efectores de la inhibición de la transcripción inducida por p53. El trabajo también mostró la relevancia de la expresión de MageA2 en la respuesta apoptótica mediada por p53 cuando se trataban cultivos primarios de melanoma humano con quimioterápicos [10] (Figura 2).
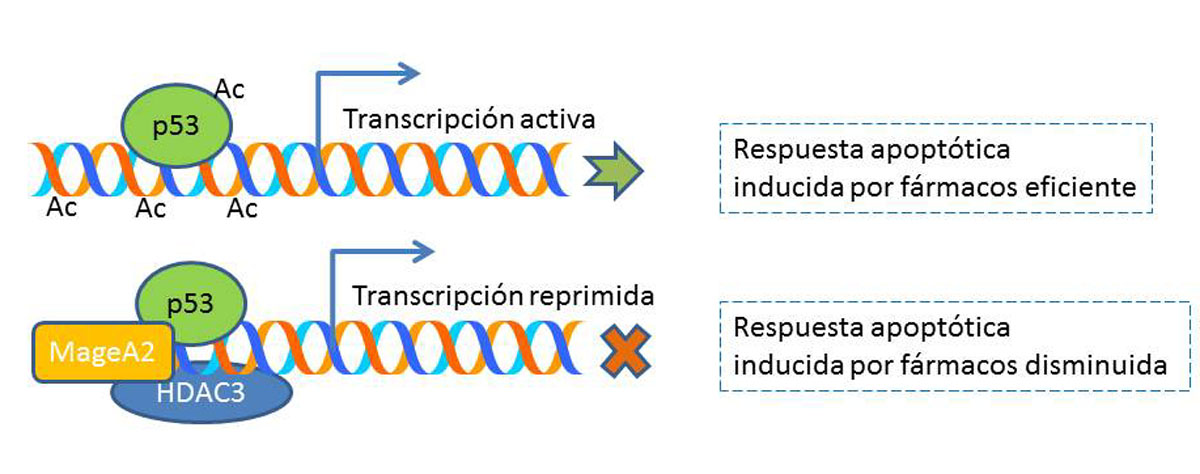
Figura 2: Esquema del modelo propuesto para la represión de p53 por MageA2 mediada por el reclutamiento de HDAC3 [10]
El desafío en ese momento era doble: por una parte se demostraba que la expresión de proteínas MAGE era beneficiosa para las células tumorales ya que, al menos MageA2 (y MageA6/A3 en menor grado), actuaba como un potente inhibidor del oncosupresor p53. Esto causaba una disminuida respuesta apoptótica en células de melanoma frente a fármacos utilizados en quimioterapia. Además, se ponía en duda la teoría de la redundancia funcional: no todas las proteínas MAGE ejercen la misma función. Luego de nuestro trabajo, varios otros grupos confirmaron la relación de la expresión de algunas proteínas MAGE y la inhibición de p53 utilizando diferentes modelos tumorales [10–13]. Posteriormente a mi incorporación al Departamento de Química Biológica (FCEN, UBA, Buenos Aires), completamos otro trabajo en colaboración con el laboratorio LNCIB (Trieste, Italia) donde demostramos que MageA2 también es capaz de interactuar con el oncosupresor PML-IV durante el proceso senescencia inducida por el oncogén Ha-ras (RasV12), evitando el arresto y colaborando con la proliferación celular [14]. Estos trabajos y otros producidos por otros laboratorios continuaron en la misma dirección, sugiriendo que la expresión de proteínas MAGE-A actuaba en distintas vías de señalización favoreciendo procesos asociados al cáncer [15].
Efecto de las proteínas MAGE como reguladores transcripcionales en vías asociados con el cáncer
Mientras nosotros y otros investigadores nos dedicábamos a estudiar las consecuencias de la expresión de las proteínas MAGE en la amplia vía de p53, también se evidenciaban efectos sobre otras vías relacionadas con el cáncer. Se mostraba que otro factor transcripcional afectado por la expresión de genes MAGE era el Receptor de Andrógenos (AR), un receptor nuclear de hormonas esteroideas y mediador del crecimiento del cáncer de próstata. El equipo de Elisabeth Wilson (North Carolina, USA) demostró que MageA11 actúa como un co-activador de AR y que su expresión en cáncer de próstata potencia la actividad de este receptor nuclear y factor de transcripción [16, 17].
También fue reportada una relación MAGE con el factor inducible por hipoxia (HIF-1alfa). HIF-1alfa es factor importante de promoción tumoral ya que está involucrado en la neovascularización y formación de metástasis. Los bajos niveles celulares de HIF son controlados por las prolil-hidrolxilasas PHD. PHD cataliza la unión de un grupo oxidrilo a una prolina deHIF-1alfa que a su vez favorece la unión de HIF-1alfa a la ubiquitina E3 ligasa VHL. Finalmente, VHL causa la ubiquitinación de HIF-1alfa y su degradación vía proteasoma [18]. Ha sido demostrado que MageA11 se asocia a PHD e inhibe su actividad prolil-hidroxilasa y este efecto termina ocasionando la estabilización indebida de HIF-1alfa [19]. A su vez, MageA11 es degradado por p14ARF, un oncosupresor que está involucrado en la activación de p53 y que también ejerce funciones directas en la inhibición del ciclo celular [20].
También surgieron otras vinculaciones entre proteínas MAGE-A y vías asociadas a la proliferación celular: MageA1 recluta HDAC1 para inhibir la transcripción mediada por SKIP1 [21]. También se mostró que los genes Mage-A3 y Mage-A6 son blancos transcripcionales de la vía de FGFR2. La activación de FGFR2-IIIb impide la señalización del oncogén BRAF vía MAPKs y por lo tanto se comporta como un supresor tumoral [22]. Se comprobó que FGF7/FGFR2-IIIb no solo inhibe la señalización del oncogén BRAF sino promueve el silenciamiento de los genes MageA3 y MageA6 por metilación y desacetilación de sus promotores [23]. De esta manera, la señalización vía FGFR2-IIIb bloquea dos potenciales vías pro-oncogénicas. Ver Tabla 1.
Tabla 1: Principales interacciones de proteínas MAGE-I y sus efectos biológicos.
| Proteína MAGE | Interactor | Efecto biológico |
|---|---|---|
| MageA1 | SKIP1 | Represión transcripcional mediante reclutamiento de HDAC1 [21] |
| MageA2 | p53 | Represión transcripcional mediante reclutamiento de HDAC3 en células estresadas y disminución de apoptosis [10] |
| MageA2 | PML IV | Inhibición de la senescencia inducida por oncogenes [14] |
| MageA2 | Mdm2 | Estabilización de Mdm4 e inhibición de p53 [24] |
| MageA2 | p53 | Disminución de unión p53 a cromatina en células no estresadas [24] |
| MAGE (varios) | KAP1 | Represión transcripcional de p53 [12] |
| MageA3/A6 | KAP1 | Degradación de AMPK1, inhibición de autofagia y aumento de la vía de mTOR [25] |
| MageA11 | AR | Activación transcripcional de AR [26] |
| MageA11 | MageA6 | Estabilización de MageA11 e hiperactivacion de AR [27] |
| MageA11 | p14ARF | Degradación de MageA11 independiente de ubiquitinación [28] |
| MageA11 | PHD | Acumulación de HIF1 en normoxia [19] |
| MageB2 | HDAC1 | Aumento de la actividad E2F y proliferación celular [29] |
| MageC2 | Rbx1 | Estabilización de Ciclina E y proliferación celular [30] |
El panorama del resultado de la expresión de genes MAGE-A se iba aclarando: su expresión afectaba, mediante mecanismos diferentes, la actividad de factores de transcripción y vías de señalización asociadas al cáncer, guiando siempre el comportamiento de las células tumorales hacia contextos menos regulados y más agresivos. Además, se esclarecía la idea que no todos las proteínas MAGE-A ejercían la misma función y que la determinación de la expresión génica debía considerarse específicamente para algunos miembros de la familia MAGE-A y no en su conjunto. De esta manera, hoy en día, la expresión de genes MAGE-A en muestras tumorales humanas correlaciona con mal pronóstico, resistencia a fármacos y estadios avanzados de la enfermedad. Citamos aquí solo alguno de los últimos trabajos realizados en pacientes con: carcinoma urinario [31], carcinoma oral de células escamosas [32], adenocarcinoma de pulmón [33], carcinoma de mama [34] y cáncer hepatocelular [35].
Más recientemente, iniciamos estudios funcionales sobre proteínas de la subfamilia MAGE-B. Varios años atrás, la localización nucleolar de MageB2 (a diferencia de la localización nuclear de las proteínas MAGE-A) nos había hecho plantear la inconsistencia en la teoría de la superposición de función en las proteínas MAGE. Finalmente pudimos encarar la caracterización funcional de esta proteína y observamos que no afectaba la vía de p53, pero sí a la de los factores de transcripción E2Fs involucrados en la proliferación celular. De hecho, la expresión de MageB2 evidenció su efecto proliferativo no solo en células en cultivo, sino también en tumores murinos (melanoma B16) modificados por nosotros para su expresión y creciendo en ratones de experimentación [29]. El mecanismo de activación de E2F-1, E2F-2 y E2F-3, involucraba la unión de MageB2 con HDAC1 (inhibidor de E2Fs reclutado por pRb) y desensamblando el complejo HDAC1/pRb/E2F. De esta manera se genera E2F libre y transcripcionalmente activo para la inducción de la proliferación celular [29] (Figura 3). Hemos detectado en MageB2 una secuencia en su región amino terminal que le permite pasar rápidamente del nucleoplasma al nucléolo respondiendo a distintas condiciones de estrés celular. Las funciones de MageB2 en el nucléolo están actualmente en estudio en nuestro laboratorio.
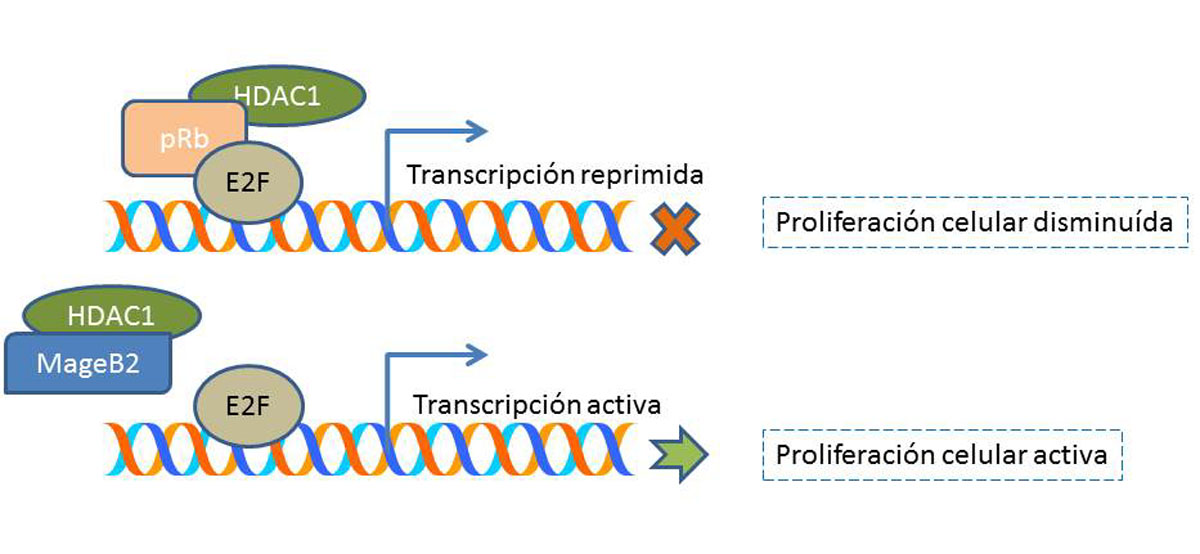
Figura 3: Esquema del modelo propuesto para la activación de factores E2F por MageB2. La expresión de MageB2 desensambla el complejo E2F/pRb/HDAC1 dejando a E2F como factor de transcripción activo [29].
Proteínas MAGE y ubiquitinación
En el año 2010, el campo de acción de las proteínas MAGE invadió también la regulación de la estabilidad de proteínas. El grupo de Ryan Potts (Dallas, USA), realizó una serie de purificaciones por afinidad en tándem seguidas de espectrometría de masas (TAP/MS), llevándolos a detectar que algunas proteínas MAGE se asociaban a E3 ubiquitina ligasas con dominios RING [36]. Específicamente, caracterizaron la unión MageD1/NSE1; MageC2/TRIM28; MageA6/TRIM28 y MageD1/Praja1. Si bien todas las proteínas poseen un domino RING característico de las ubiquitina E3 ligasas de la familia RING, solo Praja1 había sido caracterizada anteriormente como E3 ligasa. Tanto NSE1 como TRIM28 (también llamado KAP1) eran conocidas por su acción en la reparación del ADN y la inhibición de la transcripción. Sin embargo, el complejo de estas con proteínas MAGE específicas, evidenciaron una clara actividad ubiquitina ligasa [36]. Este trabajo abrió un nuevo horizonte para las proteínas MAGE asociado a la regulación de la degradación proteica. Uno de los trabajos más importantes derivado de estas observaciones, fue que el complejo MageA6/TRIM28, una ubiquitina ligasa específica de tumores (debido a la expresión diferencial de MageA6), degrada a AMPK (AMP activated protein kinase) [37]. Esta es una enzima capaz de inhibir vías activadas en cáncer como la de mTOR y por ende considerada un supresor tumoral [38].
Con respecto al rol de proteínas MAGE en la regulación de la estabilidad proteica, también existen reportes que indican su acción en procesos opuestos, es decir, la estabilización de proteínas [24, 30] mediante la inhibición de ubiquitina E3 ligasas y no su potenciación. En este contexto, nosotros recientemente evidenciamos un nuevo escalón en la complejidad de estas proteínas: MageA6 protege a MageA11 de su ubiquitinación y degradación por el proteasoma [27]. Esto indica que tumores que expresan MageA11 se ven afectados a nivel proteína por la co-expresión de otro gen MAGE-A, en este caso, MageA6. Demostramos que MageA6 y MageA11 interactúan mediante sus dominios MHD, resultando en la disminución en la ubiquitinación de las lisinas 140 y 145 de MageA11. Comprobamos que MageA6 y MageA11 están frecuentemente co-expresados en tumores de próstata y de testículo y que MageA6 potencia la actividad de MageA11 como co-activador de AR [27] (Figura 4).
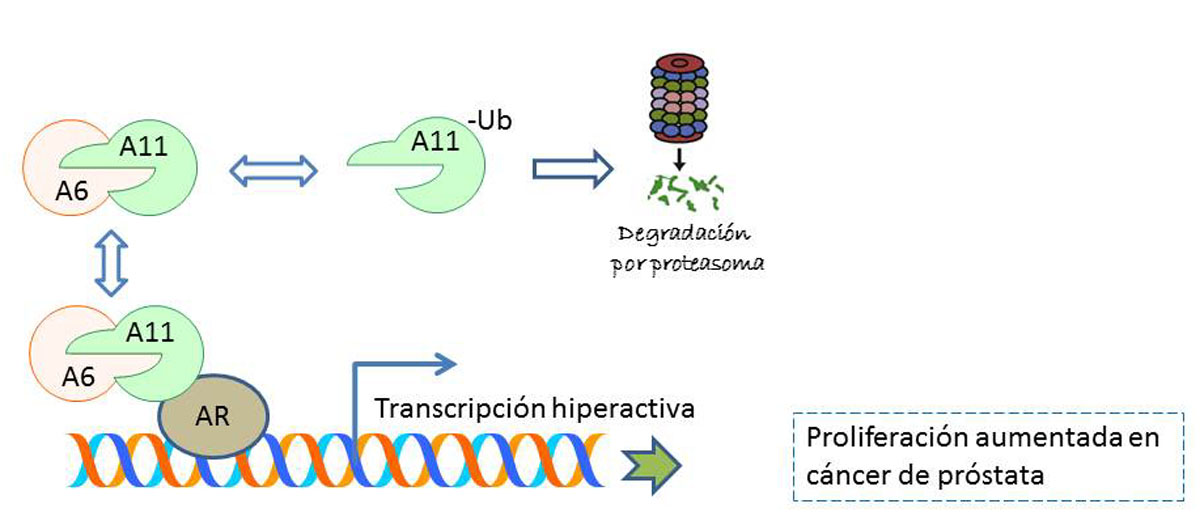
Figura 4: Esquema del modelo propuesto para la colaboración de MageA6 en la activación de AR por MageA11. La interacción de MageA6 y MageA11 por sus dominios MHD, protege la degradación de MageA11 por el sistema ubiquitina-proteasoma [27].
Conclusiones:
El descubrimiento de proteínas tumorales de la familia MAGE provocó grandes expectativas ya que evidenciaba un claro potencial como blancos de inmunoterapia. Hasta el momento se realizó un número elevado de intentos muy bien diagramados aunque con resultados no tan alentadores. En base a esto, se proyectan actualmente nuevas formulaciones y modificaciones con esperanzas de mejores resultados.
También se postulaba que la expresión de proteínas MAGE podría correlacionar con buen pronóstico ya que esos marcadores señalaban a las células tumorales que las expresaban como blancos de sistema inmune. Sin embargo, es poco probable que las células tumorales expresen proteínas que perjudicaran su propio crecimiento: durante el proceso de oncogénesis se seleccionan células capaces de evadir los distintos mecanismos de oncosupresión y no su propia eliminación.
Varios de los estudios posteriores utilizando técnicas de bioquímica, biología molecular y biología celular, evidenciaron un claro perfil pro-oncogénico en un buen número de proteínas MAGE-I (expresión tumoral). Estos trabajos establecieron que distintas proteínas MAGE-I pueden poseer efectos que guían hacia la promoción tumoral interviniendo en vías de señalización específicas.
En estos casi 25 años desde el descubrimiento de los genes MAGE, pero poco más de 10 años desde su estudio profundo a nivel celular se han elucidado varios aspectos, pero también se deja de manifiesto grandes problemas por la complejidad del mundo MAGE. El camino trazado en cuanto a la función de las proteínas MAGE-I como proteínas pro-tumorales se seguirá explorando. Sin embargo, dada la complejidad inesperada se necesitará un estudio más sistemático. La problemática que se vislumbra está asociada a los siguientes puntos: i) se comenzó afrontando el estudio de marcadores de melanoma que pronto se convirtieron en marcadores de casi todos los tumores humanos; ii) de tres proteínas (MageA1, MageA2 y MageA3) se pasó a tener que considerar más de 60; iii) la expresión tipo CTA (Cancer-Testis Antigens) del 100% de los genes MAGE-I está en revisión ya que en algunos organismos se detectó expresión de algún miembro MAGE-I en células no tumorales adicionales a testículo, como las células estaminales [39] y cerebro [40]. Un poco menos sorprendente es su expresión durante la embriogénesis [41]; iv) hemos puesto de manifiesto la posibilidad de interacción funcional entre diferentes proteínas MAGE-I por su dominio altamente conservado, el MHD [27], que sumado a su frecuente co-expresión en células tumorales, postula la existencia de una red de regulación interna.
Considerando que el fin último en la investigación biomédica es aportar conocimiento que se pueda aplicar a la salud humana, en el futuro próximo se espera profundizar sobre la base que se ha forjado en este campo, en forma ordenada y considerando los nuevos puntos de complejidad biológica. De esta forma, no solo serán seleccionados miembros de esta familia génica que puedan ser utilizados con valor pronóstico o diagnóstico de determinados cánceres humanos, sino la potencialidad de inhibir las vías afectadas por su expresión.
Referencias:
1. Jäger E, and Knuth A. (2012) The discovery of cancer/testis antigens by autologous typing with T cell clones and the evolution of cancer vaccines. Cancer Immunology 12: 6.
2. van der Bruggen P, Traversari C, Chomez P, Lurquin C, De Plaen E, Van den Eynde B, et al. (1991) A gene encoding an antigen recognized by cytolytic T lymphocytes on a human melanoma. Science 254: 1643–7 DOI: 10.1126/science.1840703.
3. Simpson AJG, Caballero OL, Jungbluth A, Chen Y-T, and Old LJ. (2005) Cancer/testis antigens, gametogenesis and cancer. Nature Review Cancer 5: 615–625 DOI: 10.1038/nrc1669.
4. Lee AK, and Potts PR. (2017) A Comprehensive Guide to the MAGE Family of Ubiquitin Ligases. Journal of Molecular Biology 429: 1114–1142 DOI: 10.1016/j.jmb.2017.03.005.
5. Chomez P, Backer O De, Bertrand M, Plaen E De, Boon T, and Lucas S. (2001) An Overview of the MAGE Gene Family with the Identification of All Human Members of the Family An Overview of the MAGE Gene Family with the Identification of All Human Members of the Family 1. Cancer Research 61: 5544–5551.
6. Weon JL, and Potts PR. (2015) The MAGE protein family and cancer. Current Opinion in Cell Biology 37: 1–8 DOI: 10.1016/j.ceb.2015.08.002.
7. De Smet C, Lurquin C, Lethé B, Martelange V, and Boon T. (1999) DNA methylation is the primary silencing mechanism for a set of germ line- and tumor-specific genes with a CpG-rich promoter. Molecular Cell Biology 19: 7327–35.
8. Monte M, Benetti R, Collavin L, Marchionni L, Del Sal G, and Schneider C. (2004) hGTSE-1 Expression Stimulates Cytoplasmic Localization of p53. Journal of Biological Chemistry 279: 11744–11752 DOI: 10.1074/jbc.M311123200.
9. Monte M, Benetti R, Buscemi G, Sandy P, Del Sal G, and Schneider C. (2003) The cell cycle-regulated protein human GTSE-1 controls DNA damage-induced apoptosis by affecting p53 function. Journal of Biological Chemistry 278: 30356–30364.
10. Monte M, Simonatto M, Peche LY, Bublik DR, Gobessi S, Pierotti MA, et al. (2006) MAGE-A tumor antigens target p53 transactivation function through histone deacetylase recruitment and confer resistance to chemotherapeutic agents. Proceedings of the National Academy of Science of the United States of America 103: 11160–5.
11. Ladelfa MF, Peche LY, Toledo MF, Laiseca JE, Schneider C, and Monte M. (2012) Tumor-specific MAGE proteins as regulators of p53 function. Cancer Letters 325: 11–17 DOI: 10.1016/j.canlet.2012.05.031.
12. Yang B, O’Herrin SM, Wu J, Reagan-Shaw S, Ma Y, Bhat KMR, et al. (2007) MAGE-A, mMage-b, and MAGE-C proteins form complexes with KAP1 and suppress p53-dependent apoptosis in MAGE-positive cell lines. Cancer Research 67: 9954–9962 DOI: 10.1158/0008-5472.CAN-07-1478.
13. Marcar L, MacLaine NJ, Hupp TR, and Meek DW. (2010) Mage-A cancer/testis antigens inhibit p53 function by blocking its interaction with chromatin. Cancer Research 70: 10362–10370 DOI: 10.1158/0008-5472.CAN-10-1341.
14. Peche LY, Scolz M, Ladelfa MF, Monte M, and Schneider C. (2012) MageA2 restrains cellular senescence by targeting the function of PMLIV/p53 axis at the PML-NBs. Cell Death Differentiation 19: 926–936 DOI: 10.1038/cdd.2011.173.
15. Barker PA, and Salehi A. (2002) The MAGE proteins: Emerging roles in cell cycle progression, apoptosis, and neurogenetic disease. Journal of Neurosciences Research 67: 705–712 DOI: 10.1002/jnr.10160.
16. Bai S, and Wilson EM. (2008) Epidermal-growth-factor-dependent phosphorylation and ubiquitinylation of MAGE-11 regulates its interaction with the androgen receptor. Molecular Cell Biology 28: 1947–63 DOI: 10.1128/MCB.01672-07.
17. Karpf AR, Bai S, James SR, Mohler JL, and Wilson EM. (2009) Increased expression of androgen receptor coregulator MAGE-11 in prostate cancer by DNA hypomethylation and cyclic AMP. Molecular Cancer Research 7: 523–35 DOI: 10.1158/1541-7786.MCR-08-0400.
18. Jaakkola P, Mole DR, Tian YM, Wilson MI, Gielbert J, Gaskell SJ, et al. (2001) Targeting of HIF-alpha to the von Hippel-Lindau ubiquitylation complex by O2-regulated prolyl hydroxylation. Science 292: 468–72 DOI: 10.1126/science.1059796.
19. Aprelikova O, Pandolfi S, Tackett S, Ferreira M, Salnikow K, Ward Y, et al. (2009) Melanoma antigen-11 inhibits the hypoxia-inducible factor prolyl hydroxylase 2 and activates hypoxic response. Cancer Research 69: 616–624 DOI: 10.1158/0008-5472.CAN-08-0811.
20. Minges JT, Grossman G, Zhang P, Kafri T, and Wilson EM. (2015) Post-translational down-regulation of melanoma antigen-A11 (MAGE-A11) by human p14-ARF tumor suppressor. Journal of Biological Chemistry 290: 25174–25187 DOI: 10.1074/jbc.M115.663641.
21. Laduron S, Deplus R, Zhou S, Kholmanskikh O, Godelaine D, De Smet C, et al. (2004) MAGE-A1 interacts with adaptor SKIP and the deacetylase HDAC1 to repress transcription. Nucleic Acids Research. 32: 4340–4350 DOI: 10.1093/nar/gkh735.
22. Kondo T, Zheng L, Liu W, Kurebayashi J, Asa SL, and Ezzat S. (2007) Epigenetically controlled fibroblast growth factor receptor 2 signaling imposes on the RAS/BRAF/mitogen-activated protein kinase pathway to modulate thyroid cancer progression. Cancer Research 67: 5461–70 DOI: 10.1158/0008-5472.CAN-06-4477.
23. Kondo T, Zhu X, Asa SL, and Ezzat S. (2007) The cancer/testis antigen melanoma-associated antigen-A3/A6 is a novel target of fibroblast growth factor receptor 2-IIIb through histone H3 modifications in thyroid cancer. Clin. Cancer Research 13: 4713–20 DOI: 10.1158/1078-0432.CCR-07-0618.
24. Marcar L, Ihrig B, Hourihan J, Bray SE, Quinlan PR, Jordan LB, et al. (2015) MAGE-A Cancer/Testis Antigens Inhibit MDM2 Ubiquitylation Function and Promote Increased Levels of MDM4. PLoS One 10: e0127713 DOI: 10.1371/journal.pone.0127713.
25. Pineda CT, Ramanathan S, Fon Tacer K, Weon JL, Potts MB, Ou Y-H, et al. (2015) Degradation of AMPK by a Cancer-Specific Ubiquitin Ligase. Cell 160: 715–728 DOI: 10.1016/j.cell.2015.01.034.
26. Bai S, He B, and Wilson EM. (2005) Melanoma antigen gene protein MAGE-11 regulates androgen receptor function by modulating the interdomain interaction. Molecular Cell Biology 25: 1238–57 DOI: 10.1128/MCB.25.4.1238-1257.2005.
27. Laiseca JE, Ladelfa MF, Cotignola J, Peche LY, Pascucci FA, Castaño BA, et al. (2017) Functional interaction between co-expressed MAGE-A proteins. PLoS One 12: e0178370 DOI: 10.1371/journal.pone.0178370.
28. Minges JT, Grossman G, Zhang P, Kafri T, and Wilson EM. (2015) Post-translational Down-regulation of Melanoma Antigen-A11 (MAGE-A11) by Human p14-ARF Tumor Suppressor. Journal of Biological Chemistry 290: 25174–87 DOI: 10.1074/jbc.M115.663641.
29. Peche LY, Ladelfa MF, Toledo MF, Mano M, Laiseca JE, Schneider C, et al. (2015) Human MageB2 protein expression enhances E2F transcriptional activity, cell proliferation, and resistance to ribotoxic stress. Journal of Biological Chemistry 290: 29652–29662 DOI: 10.1074/jbc.M115.671982.
30. Hao J, Song X, Wang J, Guo C, Li Y, Li B, et al. (2015) Cancer-testis antigen MAGE-C2 binds Rbx1 and inhibits ubiquitin ligase-mediated turnover of cyclin E. Oncotarget 6: 42028–42039 DOI: 10.18632/oncotarget.5973.
31. Makise N, Morikawa T, Nakagawa T, Ichimura T, Kawai T, Matsushita H, et al. (2016) MAGE-A expression, immune microenvironment, and prognosis in upper urinary tract carcinoma. Human Pathology 50: 62–9 DOI: 10.1016/j.humpath.2015.11.007.
32. Brisam M, Rauthe S, Hartmann S, Linz C, Brands RC, Kübler AC, et al. (2016) Expression of MAGE-A1-A12 subgroups in the invasive tumor front and tumor center in oral squamous cell carcinoma. Oncology Reports 35: 1979–86 DOI: 10.3892/or.2016.4600.
33. Zhai X, Xu L, Zhang S, Zhu H, Mao G, and Huang J. (2016) High expression levels of MAGE-A9 are correlated with unfavorable survival in lung adenocarcinoma. Oncotarget 7: 4871–81 DOI: 10.18632/oncotarget.6741.
34. Wang H, Sang M, Geng C, Liu F, Gu L, and Shan B. (2016) MAGE-A is frequently expressed in triple negative breast cancer and associated with epithelial-mesenchymal transition. Neoplasma 63: 44–56 DOI: 10.4149/neo_2016_006.
35. Gu X, Fu M, Ge Z, Zhan F, Ding Y, Ni H, et al. (2014) High expression of MAGE-A9 correlates with unfavorable survival in hepatocellular carcinoma. Scientific Reports. 4: 6625 DOI: 10.1038/srep06625.
36. Doyle JM, Gao J, Wang J, Yang M, and Potts PR. (2010) MAGE-RING protein complexes comprise a family of E3 ubiquitin ligases. Molecular Cell 39: 963–974 DOI: 10.1016/j.molcel.2010.08.029.
37. Pineda CT, Ramanathan S, Fon Tacer K, Weon JL, Potts MB, Ou Y-H, et al. (2015) Degradation of AMPK by a cancer-specific ubiquitin ligase. Cell 160: 715–28 DOI: 10.1016/j.cell.2015.01.034.
38. Hardie DG, and Alessi DR. (2013) LKB1 and AMPK and the cancer-metabolism link - ten years after. BMC Biology 11: 36 DOI: 10.1186/1741-7007-11-36.
39. Lifantseva N, Koltsova A, Krylova T, Yakovleva T, Poljanskaya G, and Gordeeva O. (2011) Expression patterns of cancer-testis antigens in human embryonic stem cells and their cell derivatives indicate lineage tracks. Stem Cells International 2011: 795239 DOI: 10.4061/2011/795239.
40. Morgan RA, Chinnasamy N, Abate-Daga D, Gros A, Robbins PF, Zheng Z, et al. (2013) Cancer regression and neurological toxicity following anti-MAGE-A3 TCR gene therapy. Journal of Immunotherapy. 36: 133–51 DOI: 10.1097/CJI.0b013e3182829903.
41. Gjerstorff MF, Harkness L, Kassem M, Frandsen U, Nielsen O, Lutterodt M, et al. (2008) Distinct GAGE and MAGE-A expression during early human development indicate specific roles in lineage differentiation. Human Reproduction 23: 2194–2201 DOI: 10.1093/humrep/den262.
|
Revista QuímicaViva Número 3, año 16, Diciembre 2017 quimicaviva@qb.fcen.uba.ar |