La complejidad de lo simple: la célula bacteriana
Mercedes Berlanga 1 y Ricardo Guerrero 2,3
1 Departamento de Biología, Sanidad y Ambiente, Sección de Microbiología, Facultad de Farmacia y Ciencias de la Alimentación. Universidad de Barcelona 2 Departamento de Patología Terapéutica Experimental, Laboratorio de Microbiología Molecular y Antimicrobianos, Facultad de Medicina, Campus de Bellvitge. Universidad de Barcelona
3 Barcelona Knowledge Hub de la Academia Europaea.
Barcelona, España.
mberlanga@ub.edu | RGuerrero@iec.cat
El famoso entomólogo y padre de la sociobiología Edward O. Wilson, de la Universidad de Harvard, escribió el siguiente párrafo para finalizar su libro autobiográfico Naturalist (1994, Island Press, Washington, p. 364): “Si pudiera empezar de nuevo y revivir mi visión en el siglo XXI, sería ecólogo microbiano. Diez mil millones de bacterias viven en un gramo de tierra, lo que puedo coger entre mis dedos pulgar e índice. Representan miles de especies, casi ninguna de las cuales son conocidas por la ciencia. Penetraría en ese mundo con la ayuda de la microscopía moderna y con las herramientas del análisis molecular. Iría abriendo camino a través de bosques clonales extendidos entre granos de arena, viajaría en un submarino imaginario dentro de gotas de agua que tendrían el tamaño de lagos y rastrearía a depredadores y presas para descubrir nuevas formas de vida y extrañas redes tróficas”.
Un vasto universo de “vida invisible”, todavía inexplorado, será observado en los próximos años. La combinación e integración de la ecología, de la genómica, de la proteómica y de otros estudios moleculares, será la llave que abrirá las puertas del misterio. Pero, como ocurre con cada descubrimiento, esa nueva visión no hará más que cohibirnos al percatarnos, aún más, de la enorme hermosura y diversidad de la naturaleza.
Origen y desarrollo de la Microbiología
Para entender qué son y qué hacen los microorganismos hay que ir atrás en el tiempo y repasar históricamente la visión que los científicos han ido teniendo del inmenso e “invisible” mundo microbiano [1-3]. Así, podemos destacar tres grandes Edades de Oro de la Microbiología. En la primera (finales del siglo XIX y principios del XX), los avances tecnológicos (autoclaves, filtros, incubadoras, etc.) y el desarrollo de las técnicas básicas para el aislamiento y cultivo axénico (o “puro”), permitieron a los fundadores de la microbiología, Louis Pasteur (1822–1895) y Robert Koch (1843–1910), confirmar que los microorganismos eran la causa de las enfermedades infecciosas y los agentes contaminantes de los alimentos y de las aguas.
Figura 1. Tres cepas de Bacillus licheniformis aisladas de los tapetes microbianos del Delta del Ebro, España, en el medio de cultivo MRS agar (De Bioplásticos, Madrid: CYTED con permiso).
La evolución intelectual y las aplicaciones prácticas de esta Primera Edad de Oro impulsaron la microbiología a constituir una de las principales ramas de la biología y la medicina. Durante esta época, la microbiología estaba centrada en el estudio de los microorganismos patógenos (infecciosos) y en la respuesta del hospedador infectado.
En la Segunda Edad de Oro (aproximadamente las décadas de 1940 a 1980), los microbiólogos adquirieron una comprensión detallada del metabolismo, la estructura y la genética de los microorganismos gracias, entre otros, a los trabajos pioneros de Martinus W. Beijerinck (1851–1931) y Albert J. Kluyver (1988–1956). Baas Becking (1895–1963) estuvo muy influido por los estudios de Beijerink, y estableció la base para una visión general del papel de las bacterias en el ciclo de los nutrientes en la biosfera, y por tanto de las interacciones entre la vida y la Tierra. Sugirió el concepto de Gaia más de 30 años antes de la propuesta explícita de Lovelock. Baas Becking dijo “todo está en todas partes, pero el ambiente selecciona”, principio prevalente hoy en día en los estudios modernos de biogeografía microbiana y metagenómica de hábitats. Kluyver es el fundador de la bioquímica comparada, propuso la idea de la unidad bioquímica de la vida y utilizó los microorganismos para elucidar las vías metabólicas y las transformaciones energéticas de la materia. La continuidad y unidad de la vida que conocemos se pone de manifiesto en la uniformidad de los sistemas genéticos y de la composición molecular que la integran (“Lo que es cierto para Escherichia coli lo es también para el elefante”, dijo Jacques Monod [1910-1976]). Durante este período, la microbiología estaba totalmente fragmentada, con una total separación e incomunicación entre los microbiólogos clínicos, por una parte, y los microbiólogos bioquímicos, genéticos y ambientales, por la otra.
La Tercera Edad de Oro (a partir de la década de 1980) comienza con la utilización de técnicas moleculares en general, y en el s. XXI de las técnicas “-ómicas” (metagenómica, metatranscriptómica, metaproteómica, o metabolómica. Estas aproximaciones han abierto perspectivas insospechadas en la detección, la identificación y el estudio funcional de los microorganismos en la naturaleza, donde hasta hace poco se pensaba que eran escasos e indetectables. En la actualidad, la microbiología ha vuelto a unificarse. Los campos de la fisiología, genética, ecología, y patogénesis microbianas pueden interaccionar, no sólo porque comparten bases de datos de genómica y herramientas moleculares, sino por la constatación de que los microorganismos utilizan mecanismos similares para llevar a cabo diversas funciones en los diferentes hábitats considerados. Como resultado, las subdisciplinas de la microbiología no son ahora campos aislados de estudio. En un poema (Moretum, versos 103-104) de Virgilio escribe: E pluribus unum, es decir, hay una unidad en la variedad. A todos los microbiólogos nos une el mismo interés y entusiasmo: nuestra ciencia y sus protagonistas, los microorganismos.
Como resultado de este extraordinario desarrollo tecnológico, podríamos considerar el inicio de la Cuarta Edad de Oro, cuyos logros y descubrimientos futuros aún no los podemos prever. Esta Edad de Oro se centraría básicamente en el holobionte, el estudio de la relación simbiótica entre el animal o planta y su microbiota asociada [4, 5], con espacial relevancia, la microbiota (conjunto de microorganismos) o microbioma (conjunto de genes de la microbiota) humanas. Ejemplo de ello esla creación de consorcios internacionales de investigación como el Human Microbiome Project (HMP) (http://hmpdacc.org), el Metagenomics of the Human Intestinal Tract (MetaHIT) (http://www.metahit.eu), el International Human Microbiome Consortium (IMHC) (http://www.human-microbiome.org). En la actualidad, se sabe poco sobre los mecanismos que permiten la supervivencia y la tolerancia a largo plazo de las comunidades indígenas microbianas o por qué estos microorganismos no producen una respuesta inflamatoria crónica perjudicial. Quizás uno de los grandes desafíos es poder entender la dinámica temporal de la comunicación bioquímica entre el huésped y su microbiota, no sólo en la escala de tiempo de una vida humana, sino también en la escala de tiempo evolutivo, en relación con los cambios globales en la dieta y los factores ambientales estresantes [6]. Es interesante entender como cambios en la microbiota puede estar relacionada con enfermedades específicas y cuál sería la mejor manera de manipular la microbiota desde un punto de vista terapéutico para obtener beneficios para la salud humana.
Las bacterias son también organismos complejos
En 1962, Roger Y. Stanier (1916–1982) y Cornelis B. van Niel (1897–1985) propusieron el concepto de “bacteria” (célula sin núcleo), que permitió a los (micro)biólogos dividir los organismos vivos en dos grandes grupos: procariotas y eucariotas. La simple observación de una bacteria en el microscopio no es muy reveladora, ya que la mayoría aparecen como pequeños bastones o esferas, sin rasgos característicos. Inicialmente se creía que los procariotas carecían de cualquier organización interna u otras características típicas de los eucariotas. Sin embargo, en las últimas décadas se ha demostrado que la estructura y la función de la célula procariota son mucho más intrincadas de lo que inicialmente se había pensado. Resumamos algunas:
Complejidad metabólica de la célula bacteriana
A pesar de esta aparente simplicidad morfológica, los procariotas presentan una enorme diversidad metabólica, que les permite ocupar los nichos ecológicos más diversos imaginables. Los procariotas son capaces de utilizar una amplia gama de sustancias reducidas (orgánicas e inorgánicas) y de agentes oxidadorespara generar gradientes electroquímicos y producir energía y poder reductor; además del metabolismo fermentador. Los agentes oxidadores son variados, por ejemplo, oxígeno, nitrato, sulfato, Fe (III), As (IV), Se (VI), etc.), dependiendo de las condiciones del hábitat [7].
En las comunidades naturales, las poblaciones microbianas interactúan constantemente entre sí, estableciendo complejas redes metabólicas, en el que se combinan las vías metabólicas y sus sistemas enzimáticos de los diferentes organismos residentes. Durante mucho tiempo, se pensaba que algunos metabolismos bacterianos, como el crecimiento anaeróbico a partir de compuestos de metano o amonio, no existían, e incluso que no eran posibles. Esto llevó a la idea de que el amonio y el metano eran inertes en condiciones de anoxia. Sin embargo, se observó que un consorcio microbiano en sedimentos anóxicos (constituido por una arquea metanotrofa y una bacteria sulfato reductora) podían oxidar el metano a dióxido de carbono acoplado a la desnitrificación, proceso que tenía lugar en ausencia total de oxígeno [8]. Por otra parte, la oxidación anaerobia del amonio (“anamox”, como se ha denominado a este proceso) contribuye de manera significativa al reciclaje biológico de nitrógeno, y se estima que representa hasta el 50% de la producción de N2 en el mar. El anamox es un proceso realizado por un grupo monofilético del dominio Bacteria, el filo Planctomycetes [9].
Complejidad estructural
Dado que las células procariotas no tienen orgánulos distintivos como los que se encuentran en las eucariotas, se pensó que su citoplasma no era más que un saco que contenía el material genético, enzimas solubles y los diferentes componentes de la maquinaria molecular, sin ningún tipo de estructuración interna. Pero se ha visto que los procariotas también tienen citoesqueleto, como las células eucariotas, lo que demuestra que el citoesqueleto no fue una invención de los eucariotas, sino que se originó ya en los procariotas. Procesos esenciales de la célula procariota, como el mantenimiento de la forma celular, la segregación del DNA, la división celular, formación de cuerpos de inclusión tales como polihidroxialcanoatos o magnetosomas, y algunos tipos de movimiento sobre superficies dependen del citoesqueleto [10, 11)].
El citoesqueleto aunque media en la citoquinesis y en la segregación cromosómica tanto en bacterias como en eucariotas, las funciones de sus elementos específicos se invierten [12]. En eucariotas la citoquinesis está mediada por moléculas de actina y en bacterias por tubulina (FtsZ). En cambio, la segregación del DNA, en bacterias està mediada por moléculas de actina (MreB y ParM) y en eucariotes por los microtúbulos. Esta aparente inversión de las funciones de actina y tubulina podría representar una evolución convergente: tal vez el último antepasado común universal de bacterias y eucariotas tenia actina y tubulina, pero todavía no dedicaban esas proteínas a funciones específicas. Alternativamente, una inversión de la función actina y tubulina pudo haber tenido lugar en uno de los dos linajes (Fig.2).
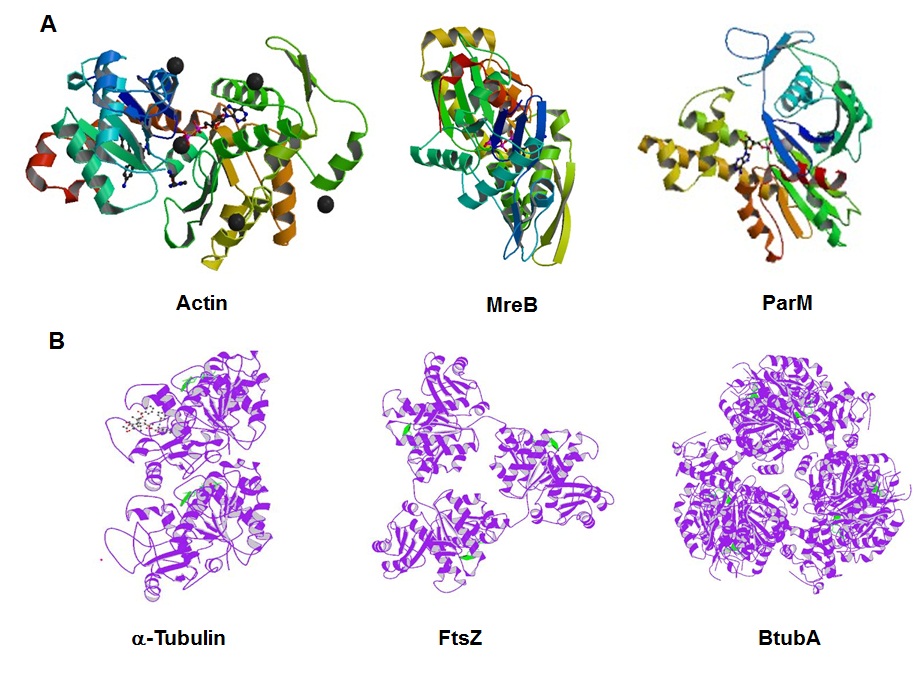
Figura 2. Comparación estructural de las proteínas del citoesqueleto de procariotas y eucariotas. Las estructuras proceden de Protein Data Bank (PDB). (A) actina; MreB de Thermotoga maritima; ParM del plásmido R1 Escherichia coli. (B) Bos tauro alfa-tubulina; FtsZ de Methanococcus jannaschii; BtubA de Prosthecobacter dejongeii. (De International Microbiology 11(3), p.164, con permiso).
También sabemos que la pared de las bacterias es una estructura dinámica. Las bacterias Gram-negativas, patógenas y no patógenas, secretan vesículas de la membrana externa (OMVs, del inglés Outer-Membrane Vesicles) durante su crecimiento en diferentes tipos de ambientes, tales como cultivo planctónico, en biofilms, en tejidos o mucosas en mamíferos. Las bacterias podrían utilizar las OMVs para mejorar su supervivencia e inducir cambios en su entorno. Las vesículas son una forma de comunicación por el cual las bacterias interactúan con las células procariotas y eucariotas en el medio. También se les atribuye numerosas funciones biológicas como la liberación de toxinas, la modulación del sistema inmunitario, pueden facilitar la infección y patogenicidad, el transporte de moléculas de señalización entre células bacterianas, transferencia de material genético, y la formación de biofilms [13, 14].
Las OMVs son producidas por evaginaciones de la pared durante el crecimiento de la bacteria y, no son productos de la lisis celular. Estas vesículas tienen unas dimensiones heterogéneas de 20 a 250 nm de diámetro, contienen membrana externa, proteínas de membrana externa, componentes del espacio periplasmático, en ocasiones también DNA y RNA. Algunas de estas vesículas también arrastran membrana citoplasmática. Es interesante destacar que los perfiles de proteínas de las OMV comparados con los de la membrana externa son similares pero no idénticos. Algunas proteínas están en mayor representación y otras no están presentes, lo que sugiere que la formación y liberación de las OMV no ocurre al azar sino que es un proceso activo y regulado. La formación de vesículas puede verse afectada por varios factores, tales como la temperatura, disponibilidad de nutrientes, oxidación, percepción de quórum, etc. Entender cómo, cuándo, dónde y por qué de estos cambios en la pared de las bacterias sin que se produzca inestabilidad de las envolturas de la célula y preservando la viabilidad de la célula es esencial para comprender la biogénesis y función de las OMV. La formación de vesículas no parece ser exclusiva de las bacterias Gram-negativas, también se han observado en las Gram-positivas (p.ej., Staphylococcus aureus) y células eucariotas [15].
Complejidad de algunos procesos de diferenciación
La muerte celular programada, o apoptosis, es un proceso fundamental en eucariotas, muy conservado evolutivamente, y que es crucial para la adecuada embriogénesis, el mantenimiento del sistema inmunitario y la eliminación de las células dañadas. La apoptosis es diferente del proceso aleatorio de la muerte celular necrótica, ya que elimina las células individuales sin inducir una respuesta inflamatoria. La apoptosis también existe en varios protistas (eucariotas unicelulares). Pero, la “apoptosis” también parece importante en algunos procesos bacterianos, como la lisis de la célula madre en la esporulación en las baciláceas, la lisis de las células vegetativas en la formación de los cuerpos fructíferos en las mixobacterias, y la transformación del DNA liberado de las células de los estreptococos sometidos a autolisis espontánea [16-18]. En las bacterias, la apoptosis parece estar determinada por una unidad genética compuesta de dos genes. El segundo gen codifica una toxina estable, y el primer gen codifica una antitoxina lábil que interfiere con la acción letal de la toxina. Este sistema genético toxina-antitoxina (por ejemplo, el sistema mazEF) se ha encontrado en E. coli y en otras bacterias, incluyendo varios patógenos [19].
Complejidad en la descripción de las especies bacterianas
Desde la primera secuenciación completa del genoma (Haemophilus influenzae), en julio de 1995, hasta julio de 2017, en la base de datos JGI_Gold (http://gold.jgi.doe.gov/) contienen 277.621 organismos de Bacteria, 2.408 de Archaea, 17.551 de Eukarya y 8.256 de virus. La genómica (en el caso de especies microbianas individuales) reveló las diferencias entre microorganismos estrechamente relacionados y la variabilidad en el contenido genético, que contienen elementos móviles como profagos, secuencias de inserción, transposones, y plásmidos. La secuencia total del genoma nos ayuda a responder cuál es el repertorio completo de genes de una especie bacteriana determinada. Este conjunto de genes de la especie se ha denominado pangenoma, que incluye los genes “centrales” (core), presentes en todas las cepas, y los genes “variables”, que pueden cambiar de una cepa a otra [20]. Para algunas especies, tal variación puede ser significativa, por ejemplo, si examinamos los genomas de diferentes cepas secuenciadas de Escherichia coli comensales y patógenas, todas comparten 2.200 genes (genoma central) pero el resto de sus genes pueden encontrarse sólo en una o dos cepas (genoma variable). La diversidad genómica de E. coli representa un modelo de pangenoma abierto que contiene un reservorio de más de 13.000 genes, muchos de los cuales podrían ser factores de virulencia no caracterizados [21]. El estudio de los genomas en un marco evolutivo ha demostrado ser útil para comprender el funcionamiento de los organismos sin olvidar que el ambiente es donde evolucionan y funcionan el patrimonio genético de los organismos, y de él dependen, en último extremo, la supervivencia, la organización y modulación del genoma de las poblaciones, y la respuesta a las constantes variaciones del ambiente. El estudio del pangenoma ayudará a la comprensión de los microbiólogos de la taxonomía bacteriana, por ejemplo, los análisis genómicos de Shigella —una de las principales causas de disentería— confirman que este microorganismo es una E. coli, en lugar de formar un género independiente [22]. Resultados que vienen a corroborar estudios realizados a finales de la década de los 80. Sin embargo, desde el punto de vista clínico, se continúa utilizando Shigella como género diferente a E. coli. La taxonomía avanza más rápidamente que la tradición o utilización práctica de algunos microorganismos, bien en el ámbito clínico (p.ej., E. coli, Shigella), bien en el biotecnológico (p.ej., la bacteria productora de polihidroxialcanoato: Hydrogenomonas eutrophus ? Alcaligenes eutropha ? Ralstonia eutropha ? Wautersia eutropha ? Cupriavidus necator; en la mayoría de artículos, se sigue utilizando los nombres de Alcaligenes o Ralstonia).
La complejidad del sistema inmunitario de las bacterias
La revista Science eligió en 2015 el sistema CRISPR (repeticiones palindrómicas espaciadas agrupadas regularmente) como el avance científico más importante del año. El desarrollo de esta técnica de procesado y modificación de genomas supone una herramienta fundamental para la ingeniería genética, comparable a lo que representó la PCR en la década de 1980. Es difícil recordar una revolución que haya impactado la biología más rápidamente que el sistema CRISPR. El mecanismo del sistema CRISPR, utilizado originalmente por las bacterias para defenderse contra la infección de los virusy plásmidos, podría ser utilizado como una técnica sencilla y fiable para la edición (modificación) del genoma de cualquier célula, como las de mamíferos y otros organismos [23, 24].
El descubrimiento del sistema CRISPR lo hizo en 1993 Francisco Mojica, de la Universidad de Alicante, cuando aún no había terminado su tesis doctoral. Mojica estaba trabajando con la arquea Haloferax mediterranii, que es muy abundante en las salinas. Mojica estudiaba las modificaciones genéticas inducidas por las altas concentraciones de sales en regiones específicas del genoma de la arquea; estas modificaciones alteraban el funcionamiento de algunas proteínas que cortaban el DNA. Una de esas regiones contenía diferentes secuencias repetitivas espaciadas regularmente. Dado que estas regiones repetitivas eran muy abundantes, Mojica supuso que seguramente tenían un papel muy importante en la adaptación del microorganismo en el ambiente de extrema salinidad donde vivía. En 2002, junto con el investigador holandés Ruud Jansen, que también estaba trabajando en el sistema, acuñó el nombre con que se conoce actualmente: CRISPR, que es el acrónimo de clustered regularly interspaced palindromic repeats (“repeticiones palindrómicas espaciadas y agrupadas regularmente”). Después de encontrar un fragmento de bacteriófagos en una de las secuencias, Mojica propuso la hipótesis que el sistema CRISPR es un sistema de adaptación “inmunitaria” de la célula frente a la infección por bacteriófagos. CRISPR es un sistema genético casi universal de protección e inmunización de las células procariotas [25].
Para concluir, esta revisión debemos ser conscientes de que hemos avanzado mucho en el conocimiento de la naturaleza de los microbios y de las técnicas que podemos emplear tanto para ultizarlos en nuestro beneficio como para evitar o curar sus efectos nocivos. Sabemos mucho más que antes pero debemos interiorizar que todavía nos queda mucho más por aprender. No debemos olvidar la acertada frase de Francis Bacon (1561–1628) en su Advancement of Learning: “Si un hombre comienza con certezas, terminará en dudas; pero si se contenta con comenzar con dudas, terminará con certezas”.
Referencias:
1. Schaechter M, Kolter R, Buckley M (2004) Microbiology in the 21st century. Where are we and where are we going? Report American Academy of Microbiology. American Society for Microbiology.
2. Maloy D, Schaechter M (2006) era of microbiology: a Golden Phoenix. International Microbiology 9: 1-9.
3. Maloy S (2007) Basic research on bacteria. The essential frontier. Report on the American Society for Microbiology and National Institutes of Health Workshop on Basic Bacterial Research.
4. Zilber-Rosenberg I, Rosenberg E (2008) Role of microorganisms in the evolution of animals and plants: the hologenome theory of evolution. FEMS Microbiology Reviews 32: 723-735.
5. Guerrero R, Margulis L, Berlanga M (2013) Symbiogenesis: the holobiont as a unit of evolution. International Microbiology 16: 133–143.
6. Aagaard K, Stewart CJ, Chu D (2016) Una destination, viae diversae. EMBO reports 17:1679–1684.
7. Guerrero R, Berlanga M (2006) Life's unity and flexibility: the ecological link. International Microbiology 9: 225-235.
8. Cui M, Ma A, Qi H, Zhuang X, Zhuang G (2015) Anaerobic oxidation of methane: an “active” microbial process. Microbiology open 4: 1-11.
9. Fuerst JA, Sagulenko E (2011) Beyond the bacterium: planctomycetes challenge our concepts of Microbial structure and function. Nature Reviews Microbiology 9: 403–413.
10. Guerrero R, Berlanga M (2007) The hidden side of the prokaryotic cell: rediscovering the microbial world. International Microbiology 10: 157-168.
11. Cho H (2015) The role of cytoskeletal elements in shaping bacterial cells. Journal of Microbiology and Biotechnology 25: 307-316.
12. Graumann PL (2004) Cytoskeletal elements in bacteria. Current Opinion in Microbiology 7: 565–571.
13. Lee EY, Choi DS, Kim KP, Gho YS (2008) Proteomics in Gram-negative bacterial outer membrane vesicles. Mass Spectrometry Reviews 27: 535–555.
14. Schwechheimer C, Kuehn M (2015) Outer-membrane vesicles from Gram-negative bacteria: biogenesis and functions. Nature Reviews Microbiology 13: 605–619.
15. Pathirana RD, Kaparakis-Liaskos M (2016) Bacterial membrane vesicles: Biogenesis, immune regulation and pathogenesis. Cellular Microbiology 18: 1518–1524.
16. Lewis K (2000) Programmed death in bacteria. Microbiology and Molecular Biology Reviews 64: 503-514.
17. Guerrero R, Berlanga M (2009) The evolution of microbial life: paradigma changes in Microbiology. Contributions to Science 5: 55–61.
18. Koonin EV, Zhang F (2016) Coupling immunity and programmed cell suicide in prokayotes: life-or-death choices. Bioessays 38. doi 10.1002/bies.201600186.
19. Ramisetty BC, Natarajan B, Santhosh RS (2015) mazEF-mediated programmed cell death in bacteria: “What is this?” Critical Reviews in Microbiology 41: 89-100.
20. Bentley S (2009) Sequencing the species pan-genome. Naure Reviews Microbiology 7: 258–259.
21. Rasko DA, Rosovitz MJ, Myers GS, Mongodin EF, Fricke WF, Gajer P et al (2008) The pangenome structure of Escherichia coli: comparative genomic analysis of E. coli commensal and pathogenic isolates. Journal of Bacteriology 190: 6881-6893.
22. Pettengill EA, Pettengill JB, Binet R (2016) Phylogenetic analyses of Shigella and enteroinvasive Escherichia coli for the identification of molecular epidemiological markers: whole-genome comparative analysis does not Support distinct genera designation. Frontiers in Microbiology 6: 1572.
23. Makarova KS, Haft DH, Barrangou R, Brouns SJJ, Charpentier E (2011) Evolution and classification of the CRISPR-Cas Systems. Nature Reviews Microbiology 9: 467–477.
24. Saisman J, Dellaire G (2017) genome editing in the CRISPR era. Biochemistry and Cell Biology 95: 187-201.
25. Mojica FJ, Rodriguez-Valera F (2016) The discovery of CRISPR in archaea and bacteria. FEBS Journal 283: 3162-3169.
Mercedes Berlanga es profesora de la Universidad de Barcelona.
Ricardo Guerrero es profesor de la Universidad de Barcelona y Director Académico de Barcelona Knowledge Hub de la Academia Europaea
|
Revista QuímicaViva Número 2, año 16, Agosto 2017 quimicaviva@qb.fcen.uba.ar |