Curso de
Introducción al conocimiento científico experimental*
por Dra. Celia E. Coto
Capítulo 15
Herramientas
moleculares: Biotecnología
Parte 3.

Introducción.
En este tercer capítulo de la serie Herramientas
moleculares abordaremos, entre otros, el aprendizaje de dos técnicas
nuevas la inmunotransferencia y la reacción en cadena de la polimerasa (PCR).
Veremos además qué son las sondas de ADN
y el uso de los ratones transgénicos y detallaremos ejemplos de aplicación
práctica, con ello habremos completado las expectativas planteadas en la figura
1 de los capítulos 13 y
Sondas moleculares de ADN o ARN.
Una sonda (probe en inglés)
es un artefacto que permite medir la profundidad del fondo del mar. También una
sonda, aplicada en Medicina, es un tubo que se introduce en un órgano o
conducto determinado ya sea para toma de muestra, para observar un tejido o
para alimentación. El sentido es el mismo, la búsqueda de algo con una especie
de detector. Cuando hablamos de sondas de ADN o ARN nos referimos a segmentos de ácido nucleico
de cadena simple utilizados como anzuelos para detectar una pieza de ADN fija
en un soporte o suelta dentro de una sopa de moléculas. Como podemos ver en la
figura 1 la utilización de una sonda para localizar genes (por ejemplo) se basa
en el principio de la complementariedad (ver capítulo 13). La atracción, como
si fueran imanes, entre dos hebras complementarias de ADN simple cadena o entre
una hebra de ADN y una de ARN siguiendo esta regla: A-T/C-G/T-A/G-C/. Es decir
si en una de las hebras hay A en la complementaria habrá T y viceversa, también
si en una hebra hay C en la otra habrá G y viceversa. La excepción es el ARN
que en lugar de T tiene uracilo (U). En este caso
Recordando este patrón bastará que la sonda
esté marcada con un fluorocromo (grupo químico que fluorece expuesto a la luz)
de una determinada longitud de onda o con un isótopo radiactivo como para poder
revelar a qué segmento de ácido nucleico se pegó la sonda. De esta manera
podremos averiguar si lo que estamos buscando se encuentra en la muestra.

Figura 1. Interpretación artística
de cómo funciona una sonda. La pintura fue encontrada en las páginas del
buscador Yahoo utilizando la palabra anzuelo. Nos pareció que la atracción
ejercida sobre el pez por el anzuelo refleja lo que ocurre entre secuencias que
se complementan. Desconocemos el nombre del pintor. El asterisco * señala la
sonda marcada.
Si ustedes recuerdan lo aprendido en el
capítulo 13 se trata de una reacción de hibridización de ácidos nucleicos.
Las
sondas pueden obtenerse mediante diferentes metodologías, ya sea mediante
reacciones biológicas moleculares o por síntesis química agregando nucleótido
por nucleótido hasta completar como máximo un polinucleótido de 150 unidades,
la forma de obtención se esquematiza en la figura 2.
![]()
![]()
![]()
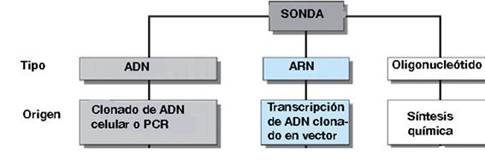
Figura 2. Tipos y modos de
obtención de sondas moleculares.
Aplicaciones
del uso de sondas. Nuevas tecnologías
La aplicación de dos de
las técnicas que hemos visto: las sondas de ADN o ARN y la reacción de
hibridización ha conducido a nuevas tecnologías de reciente aplicación
conocidas como micromatrices (microarrays)
de ADN o chips de ADN . Se trata de una manera nueva de estudiar un gran número
de genes en forma simultánea y se espera una aplicación universal en la ciencia
y en la vida diaria en un futuro próximo. Para llevar a cabo esta técnica y
crear las micromatrices se utiliza una computadora para aplicar con alta
precisión sobre un portaobjetos, gotas minúsculas que contienen ADN de genes.
Luego las placas se hibridizan con ADN complementario (cADN) marcado en forma
fluorescente o radiactiva y luego con una computadora se mide la intensidad de
cada punto fluorescente o radiactivo.
Hasta hace poco era posible estudiar en el laboratorio la actividad de
unos pocos genes y para ello se empleaban técnicas algo complicadas. Hoy en
día, la tecnología de las micromatrices está revolucionando la manera de cómo
estudiar y comparar la actividad de muchos genes simultáneamente. Existen
varios tipos de micromatrices, pero la más sencilla consiste en colocar
ordenadamente en un portaobjetos minúsculas cantidades de oligonucleótidos
correspondientes a cada gen que se quiere estudiar (P.R.Barrero. “Aplicaciones
de la técnica de microarrays en ciencias biomédicas: presente y futuro”.
Quimicaviva Nº 3. Año 4. dic 2005).
Hay un ejemplo que he sacado de la
bibliografía que se refiere a la detección de mutaciones en los genes humanos
BRCA1 y BRCA2 involucrados en el cáncer de mama. El microchip de ADN consiste
en un pequeño disco de vidrio encajado en un plástico parecido a un chip de
computadora. En su superficie se pegan cientos o miles de secuencias sintéticas
cortas de ADN de cadena simple, la suma de todas estas secuencias corresponden
al gen normal entero. En este caso en
particular los chips contienen la versión sintética de ADN de BRCA1 y BRCA2 en
forma de oligonucleótidos sintéticos que, como dijimos más arriba, en total
conforman el gen entero. Como se muestra en la figura 3 si se quiere averiguar
si una paciente tiene mutaciones en sus genes se procede según se explica en la
leyenda de la figura.
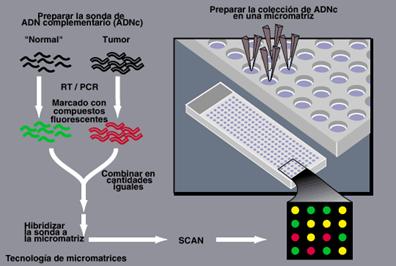
Figura 3. Se obtiene ADN de una muestra de sangre de
la paciente y otra muestra que se supone no contiene mutaciones( normal). Luego
se desnaturaliza el ADN de las muestras de modo de separar las cadenas dobles y
obtener cadenas de una sola hebra que son cortadas luego hasta obtener
moléculas más pequeñas. (La técnica de RT/PCR la veremos más adelante). Luego
se marca cada conjunto de ADN con colorante fluorescente verde la normal y con
colorante rojo la muestra incógnita, se combinan ambas en proporciones
idénticas y se insertan en los chips de modo de permitir su hibridización o
pegado. Si la persona no tiene mutaciones, ambas cadenas rojas y verdes se
pegarán al chip, en ese caso cuando se lean los colores con un rayo láser se
los verá de color amarillo. Si esto no ocurre porque las dos partes no se fijan
bien, no hibridizan, conviene investigar que está pasando, para eso el
investigador tomará las muestras correspondientes y las continuará analizando.
Western
blot
Es
una técnica que permite detectar la presencia de una proteína en un
homogeneizado o extracto de un tejido. El verbo blot en inglés significa manchar con tinta y el nombre Western
(oeste, occidental ) le fue dado por el investigador W. Neal Burnette
(Analytical Biochemistry, 112:195-203, 1981) como un juego de palabras por los
nombres Southern y Northern de las
técnicas que se usan con el mismo fin pero se aplican a los ácidos nucleicos.
Todo empezó cuando Edwin Southern. diseñó la técnica,
que lleva su nombre, para detectar ADN, luego por oposición al punto cardinal
Sur, se llamó Northen aplicada al ARN . Finalmente como una especie de broma la
técnica de inmunoblotting
(inmunotransferencia) para proteínas terminó siendo denominada Western.
Para llevar a cabo un Western blot, una
muestra de proteínas desnaturalizadas con SDS se someten a una electroforesis
en gel (PAGE) [ver Capítulo 14]. Al cabo de la corrida obtendremos un patrón de bandas que dependerá
del número de proteínas presentes en la muestra y de su tamaño. Para proceder a
su transferencia a una membrana de nitrocelulosa, vamos a poner en íntimo
contacto el gel con una membrana de nitrocelulosa o PVC y los colocaremos en un
aparato para que se produzca una electrotransferencia de las bandas de proteína
del gel a la membrana. En ese proceso se logra una transferencia cuantitativa,
manteniendo además el mismo patrón del gel. Una vez completada esta operación
las bandas se revelan por el agregado de un anticuerpo específico (ver
capítulos 8 y 9).
De esta forma se visualizarán las bandas de
proteínas y se podrá calcular la
concentración de cada una en la muestra original. Recuerden que el uso de
anticuerpos le otorga a la reacción una especificidad que de otro modo no tiene. Por otra parte, los anticuerpos pueden
usarse para revelar antígenos presentes directamente en muestras de células o
tejidos mediante las técnicas conocidas como
inmunofluorescencia o inmunoperoxidasa.
Un ejemplo de
la técnica se observa en la figura 4. La comparación de los
CV
T CV T

A B
Figura
4. Western blot. En el panel A se muestra una corrida de
electroforesis de mezclas de proteínas obtenidas a partir de un extracto de
células infectadas con virus VSV y
reveladas con coomassie blue. En el panel B se muestran los mismos
extractos celulares pero luego de la corrida electroforética las proteínas se
transfirieron a una membrana de PVDF y se realizó un western blot incubando con
un inmunosuero preparado contra la glicoproteína del virus. En las calles
indicadas como CV, las células están infectadas con el virus de la estomatitis
vesicular (VSV). En las calles indicadas con T, las células están infectadas y
tratadas con un compuesto con actividad antiviral. La banda que se observa en
el panel B corresponde a la glicoproteína viral de VSV (Gvsv). Se puede
apreciar el efecto del antiviral por la disminución del tamaño de la banda en
la calle T si se compara con CV.
(La foto es gentileza de
patrones de los geles coloreados
no muestran las diferencias que se ven fácilmente por el efecto del
inmunosuero.
Aplicaciones del western blot a la
medicina diagnóstica
La técnica del western
blot tiene muchas aplicaciones en los laboratorios de investigación pero
también ya ha sido transferida al diagnóstico de las enfermedades virales. Para
el caso de las infecciones con el virus HIV es una de las técnicas usuales para
detectar la presencia de anticuerpos circulantes contra las proteínas del virus
que es una indicación muy fuerte de que la persona se encuentra infectada por
este virus. En la figura 5 se representa la organización estructural del virus
HIV (VIH) agente causal del Síndrome de inmunodeficiencia adquirida (SIDA).
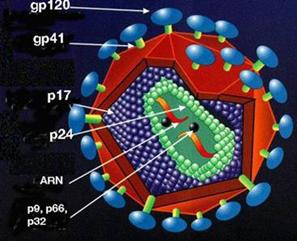
Figura 5. Estamos viendo un corte
de una partícula de virus. Contiene dos copias idénticas de ARN (coloreadas de
naranja) en la parte más interna del virus. Cada hebra tiene 9500 nucleótidos de longitud y es probable
que estén unidas. Las dos esferitas negras representan a las enzimas
transcriptasa reversa-ARNasa H* (p66) e integrasa (p32). A su vez el ARN está
asociado a una proteína de nucleocápside de carácter básico (p9). El conjunto
de ribonucleoproteína (ARN + p9) más las enzimas se conoce con el nombre de core
(núcleo). El core está protegido por una especie de caja que en los virus toma
el nombre de cápside
(nombre derivado del griego que quiere decir eso mismo: caja) . Esta estructura protectora (color verde en el
dibujo) tiene forma de cono truncado y está constituida por la proteína de la
cápside (CA) p24. La cápside está rodeada o contenida a su vez por otra
proteína (azul) conocida como la proteína de matriz (MA) p17. Ésta, a su vez,
interacciona con una envoltura o membrana de dos capas. Esta envoltura
(roja) la adquiere el virus al brotar de
la célula, se trata de la membrana que rodea al citoplasma celular. Pero el
virus inserta en la envoltura espículas o espinas (especies de pinchos)
formadas de glicoproteínas (llamadas así porque son moléculas de proteínas
azucaradas). Las espículas tienen forma de un hongo de sombrero. Estos
honguitos tienen un tallo (verde claro) enterrado en la membrana (roja) formado
por la gp41 y el sombrero (azul) está formado por la gp120. Tienen como función
ser los receptores virales, es decir, los que le permiten reconocer receptores
en las células que van a infectar. Una vez que se pegan a la célula se
desencadena un proceso de entrada del virus que luego termina en la producción
de muchos virus por cada virus que entró.
Nota: la abreviatura p es por proteína y el número que le
sigue se refiere al peso molecular, cuando se trata de glicoproteínas la
abreviatura es gp.
* ya vimos
en qué consistía la enzima retrotranscriptasa, cuando se transcribe una hebra
de ADN a partir del ARN del virus hay una enzima que debe separar amabas
cadenas para que se forme como resultado final un ADN doble, esa enzima se
conoce con el nombre de ARNasa H
¿Para qué complicarme la vida con la estructura del
HIV? se preguntarán ustedes. No se trata de una manía de Viróloga que a toda
costa muestra a un virus, la idea es que se entienda lo complejo que es este
virus desde el punto de vista de su composición proteica. De acuerdo con esta realidad
es de esperar que una persona infectada tenga circulando en su sangre
anticuerpos contra las proteínas glicosildas o no señaladas en la partícula viral o virión
El test del Western blot para el virus HIV se basa
en la presencia de anticuerpos contra las proteínas virales mencionadas.
Vayamos por partes:
1) a partir de virus purificado y tratado
adecuadamente se puede correr un gel de proteínas (PAGE) en el que aparecerán
las bandas correspondientes a las proteínas del virus como se ve en el control
positivo de la figura 7. Y según lo que se usó para obtener las proteínas se
obtendrán otras que no forman parte del virión pero que tienen que ver con el
ciclo de replicación. Recordemos que las proteínas primeramente se
desnaturalizan con SDS y luego migran, por efecto de la aplicación de una
corriente eléctrica, según su peso molecular.
Cuando termina la corrida se transfieren a una
membrana de nitrocelulosa y conservan la posición original que tenían en el
gel. ¡De acuerdo a su peso molecular!
2) Luego la membrana se incuba (se pone en
contacto), por ejemplo sumergiéndola, con una solución diluída del suero de una
persona que se sospecha que pueda estar infectada. Luego de un tiempo, los
anticuerpos contra las proteínas del virus van a reaccionar produciéndose la
unión antígeno-anticuerpo (repasar el capítulo 9). Por ejemplo: si hay
anticuerpos contra p24 en la banda donde se ubicó el antígeno o polipéptido p24
ahora también están los anticuerpos. FALTA VERLOS. Se hace luego un lavado de
la tira, si los anticuerpos no están pegados se van con el lavado, los pegados
quedan. Ahora es cuestión de revelar su presencia.
3) Para revelar la presencia de la unión específica
antígeno-anticuerpo un método eficaz es el método indirecto como se muestra en
la figura 6 y que fuera explicado en el capítulo 9 para la reacción de Elisa
indirecta.
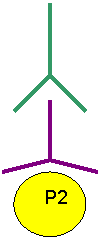
Figura 6. Esquema
de revelado de las proteínas en un Western blot. Supongamos que P1, P2 y P3 son
proteínas del virus HIV a las que se les pegó el anticuerpo respectivo presente
en el suero del paciente. Para revelar la presencia de ese complejo lo que se
hace es incubar con un segundo anticuerpo que está dirigido contra
Detección de fragmentos de ADN y ARN.
El
principio de las técnicas para detectar fragmentos de ADN (Southern blot) o ARN
(Northen blot) es esencialmente el mismo la diferencia reside en que para
localizar las bandas de interés no vamos a poder usar anticuerpos porque los
antígenos por excelencia son las proteínas y no los ácidos nucleicos. La
metodología de trabajo se complica y por lo tanto creemos que no es pertinente
recargar el contenido de este capítulo con detalles propios de los laboratorios
de investigación.
Reacción de la polimerasa en cadena.
El
propósito de esta reacción es obtener un elevado número de copias de un gen, para ello es necesario
disponer de gran cantidad de cebador o primer. Tal como se observa en la figura
8 hay tres pasos importantes en la reacción de PCR que se repiten en un total
de
Paso
1. Desnaturalización de las cadenas a 94º C
(recuerden que las cadenas se separan a
alta temperatura). Durante el proceso de desnaturalización la cadena doble se
abre y se forman dos hebras de cadena simple, además todas las reacciones
enzimáticas que tienen lugar en ese momento paran, como por ejemplo la
elongación de un ciclo previo.
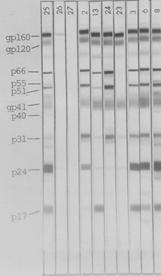
Figura 7. Western blot de varios sueros de pacientes. La primera tira muestra un
control positivo, se observa además de las proteínas del virus una gp 160 que
no forma parte del virus pero sí de las proteínas que aparecen durante el ciclo
infectivo. La tira con el número 27 es un control negativo. El resto de las
muestras tienen bandas virales. La discusión sobre el grado de positividad de
cada muestra no es de incumbencia de la
información que brindamos en este curso.
Paso
2. Anillado a 54º C
(recuerden el capítulo 13 en donde se explicó que al enfriarse la solución las
cadenas tienden a reunirse nuevamente formando el dúplex). Los primers se
encuentran flotando en la solución, de un lado a otro, o hacia arriba o hacia
abajo debido al movimiento Browniano (las partículas pequeñas en una solución tienen
movimientos erráticos o azarosos debido al efecto de la temperatura). Las uniones iónicas entre el cebador y la cadena simple molde se forman
y rompen en forma constante hasta que al encontrarse las zonas complementarias
esta unión se hace más estable y se mantiene lo suficiente para que una
polimerasa se enganche al cebador y comience a copiar la cadena molde.
Paso 3. Extensión a 72º C. Esta
polimerasa especial aislada de bacterias que viven en aguas termales calientes,
tiene como temperatura de trabajo la de
72º C. Los
cebadores que están unidos débilmente a esta temperatura se separan, en cambio,
los complementarios al gen tienen uniones suficientemente fuertes de modo que
la polimerasa sigue copiando la cadena. Recuerden que los nucleótidos se
agregan desde
Debido a
que ambas cadenas se copian durante la reacción de PCR se produce un aumento
exponencial del número de copias del gen. Así, si suponemos que hay una sola
copia del gen antes de que se inicien los ciclos, después de un ciclo habrá dos
copias, después de dos habrá cuatro, luego ocho y así sucesivamente. Un detalle
más, a lo largo de los ciclos los primers
que se usan son siempre los mismos.
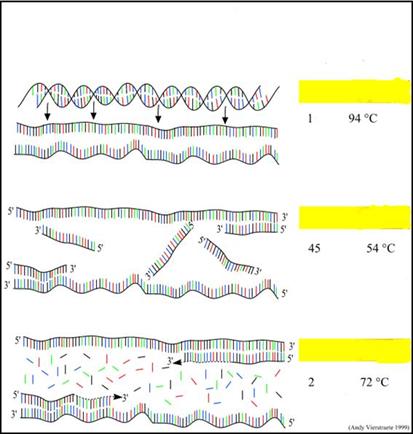
Figura 8. Pasos
de la reacción en cadena de la
polimerasa (PCR). El primer paso se realiza por 1 minuto a
http: //users.ugent.be/~avierstr/index.html.
Puede
ocurrir que la reacción haya fallado por diferentes razones por eso es
necesario comprobar que el ADN que se obtuvo corresponde al tamaño del gen que
se quería copiar. Para comprobar si la copia del gen sintetizado es la correcta
lo que se hace es correr un gel para poder determinar si el pedazo de ADN
producido en la reacción corresponde a lo esperado. Para ello los productos de
la reacción se corren en un gel comparativamente con una mezcla de fragmentos
de ADN de diferente tamaño. Si estamos sperando copiar un ADN de 1800 pares de
bases la reacción no servirá si aparece una band equivalente a 500 pares de bases.
Si observamos la figura 9 se entenderá perfectamente la situación, hay
diferentes razones por las que puede fallar la reacción pero no hacen al
objetivo de la información que aquí brindamos.
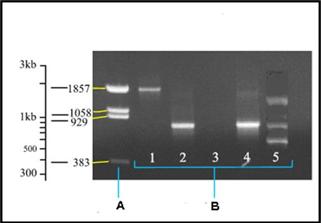
Figura 9. Gel
del ADN producto de la reacción de PCR, se trata de un gel de agarosa
transferido y revelado como se explicó antes. En la calle A se corrió una
mezcla de ADN de diferentes tamaños, notar que la escala del eje Y es
logarítmica. En la calle B 1 hay un fragmento de tamaño aproximado de 1850
bases. En las calles, 2 y 4 hay fragmentos de 800 bases de largo y en la calle
5 hay varias bandas indicando que los primers se unieron a varios genes. Mientras que en la calle 3 no hay producto,
la reacción no tuvo lugar.
http: //users.ugent.be/~avierstr/index.html.
De acuerdo con estos resultados
sólo la muestra corrida en la calle 1 es la que deseábamos obtener. De ahora en
más el ADN sintetizado podrá ser utilizado para clonar, para diagnóstico y con
propósitos de investigación pura.
Ratones transgénicos.
Los
ratones transgénicos son ratones que portan un gen que no les es propio,
generalmente de origen humano, así pueden constituirse en modelos de estudio de
enfermedades humanas y ensayo de drogas. La técnica se conoce desde 1980, los
animales transgénicos son herramientas poderosas para el estudio de
enfermedades genéticas ya que si bien el mismo tipo de manipulaciones puede
hacerse en cultivos celulares la interacción de los transgenes con proteínas,
hormonas, neurotransmisores y otros componentes del organismo sólo se pueden
dar en un ambiente fisiológico sólo en el animal entero. Aplicando otras
metodologías la inserción de un gen en una planta, por ejemplo, puede permitir
el estudio de la resistencia a una plaga.
Uno de los modos de introducir artificialmente un gen se muestra en la
figura 10. En este caso el ADN inyectado se integra al cromosoma del ratón en
cualquier lado (al azar), a veces, se integran copias múltiples. Algunos
ratones de la primera descendencia tendrán células derivadas del implante y
células que no lo son. Luego mediante cruzamiento de ratones machos y hembras
se logrará obtener una camada de ratones homogénea que exprese el gen de
interés.
Existe
otra metodología para producir ratones transgénicos pero dejaremos la
información para el lector curioso. Un ejemplo práctico de esta técnica se
encuentra en haber logrado una línea de vacunos que expresan en la leche la
proteínas transferrina humana.
Conclusión: Espero que estos tres últimos
capítulos les haya permitido asomarse al campo de la biotecnología derivada
fundamentalmente de las aplicaciones del conocimiento de la estructura del ADN.
Dado el uso generalizado de estas tecnologías en la medicina e industria
creemos que todos debemos conocer, al menos, de qué nos están hablando en este
siglo.
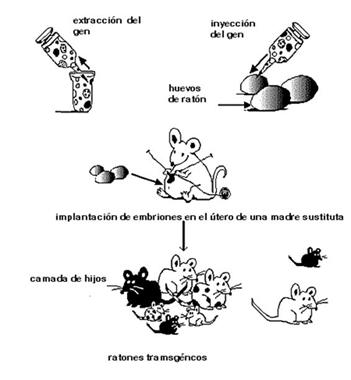
Figura 10. Pasos experimentales para obtener ratones
transgénicos. Se aisla un gen de interés y luego se inyecta en huevos
fertilizados de ratón. Los embriones se implantan en una ratona madre
sustituta, en la primera camada de hijos (dos semanas de gestación) algunos
serán transgénicos. En el dibujo estos ratones aparecen en negro y son los que
expresan el gen transfectado. Este dibujo se obtuvo del sitio “The National
Health Museum” fue traducido para el curso.
http://www.accessexcellence.org/RC/VL/GG/transgenic.htm.
*
Este curso es una contribución de Química Viva educativa (e-Lab) a la propagación del conocimiento
científico entre los estudiantes de la escuela secundaria. Departamento de
Química Biológica. Facultad de Ciencias Exactas y Naturales. Universidad de
Buenos Aires.
Volver al menú de selección de capítulo.
|
|
Revista QuímicaViva Revista Electrónica del Depto. de Química Biológica, Facultad de Ciencias Exactas y Naturales, Universidad de Buenos Aires, Argentina. quimicaviva@qb.fcen.uba.ar |