Curso de
Introducción al conocimiento científico experimental*
por
Dra. Celia E. Coto
Capítulo 14
Herramientas moleculares: Biotecnología
Parte 2.

Introducción.
Vimos en el capítulo anterior
los conocimientos indispensables para entender algunas de las reacciones
biológicas que se utilizan en la actualidad y cuyos nombres trascienden ya al
público en general. Sin embargo, los lectores sabrán comprender que pretender
resumir en un único capítulo lo ocurrido
con
En
esta segunda parte, continuaremos con una forma sencilla de presentación de las
aplicaciones concretas de las técnicas derivadas del conocimiento de
Alcances
de las técnicas moleculares.
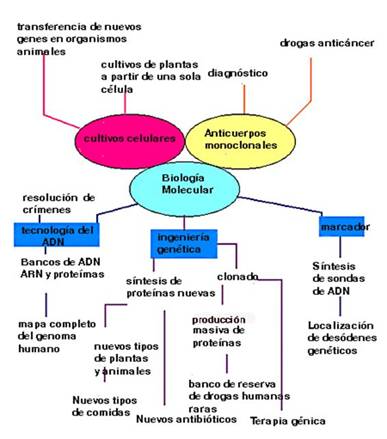
Figura 1. Esquema de los alcances del uso de la
tecnología del ADN recombinante.
Ingeniería
genética
La
ingeniería genética se define como la alteración deliberada del genoma de un
organismo por medio de la manipulación de su ADN de modo de producir un cambio
en sus características hereditarias. Como toda definición, la de ingeniería
genética es breve y concisa. Sin embargo, quisiera realizar algunos
comentarios. Es cierto que la ingeniería genética es una alteración deliberada
del genoma de un organismo, dicho de otro modo, es el cambio de un gen o genes
por el/los de otro organismo con técnicas de laboratorio. Pero no siempre la
ingeniería genética se aplica con ese objetivo de producir cambios en las
características hereditarias. Esta técnica va más allá, de la producción de
organismos rediseñados como nuevas especies, permite también otras aplicaciones
como la terapia génica, la producción de reactivos y el diseño de equipos para
diagnóstico e investigación.
Más
herramientas moleculares.
En el
capítulo 13 estudiamos las técnicas del ADN recombinante
que tienen que ver con el uso de una molécula de ADN como molde e hicimos
hincapié en los casos de la transferencia de información del ADN al ARN, nos
falta discutir sobre una técnica conocida como transcripción reversa
de uso tan común como la descripta antes. La transcripción
reversa, se denomina así porque utilizando un molde de ARN es posible
sintetizar ADN, usando para ello la enzima transcriptasa
reversa.
Por un período que abarcó desde
la publicación de la estructura del ADN hasta la demostración de la existencia
de la transcriptasa reversa ocurrida en 1970 el dogma
central de la biología molecular era el presentado en la figura 2. El estudio
de la replicación y comportamiento de un grupo de virus causantes de leucemia y
tumores en animales cuyo genoma o ácido nucleico era ARN llevó a sospechar que
replicaban a través de un ADN intermediario.
En el año 1970 H.Temin y D.Baltimore
recibieron el premio Nobel por su descubrimiento de
la enzima transcriptasa reversa, responsable de
transcribir ADN usando una hebra de ARN como molde, aislada a partir de
partículas virales. Posteriormente a este espectacular hallazgo este grupo de
virus pasó a denominarse retrovirus y a la familia a la que pertenecen Retroviridae.
Hay que lamentar que un virus miembro de esta familia, de reciente aparición,
ha matado ya a más de dos millones de personas, se trata del virus HIV (virus
de la inmunodeficiencia humana) agente causal del SIDA (síndrome de
inmunodeficiencia adquirida). Nosotros preferimos llamar al virus HIV y no VIH
(su forma española), porque los virus tienen una nomenclatura universal en la
que
![]() ADN
ADN
![]()
ARN
![]()
Proteína
Figura 2. Muestra el flujo de información del dogma central
de la biología molecular, flechas verdes).
La flecha roja muestra que el flujo contrario también existe, es la
llamada transcripción reversa.
Cebadores
o primers
Se conoce
con el nombre de cebador o primer a
una secuencia polinucleotídica de longitud variable,
no inferior a cinco nucleótidos. Cebador y primer
son nombres equivalentes, es decir, uno es la traducción del otro. Cebador se
refiere a todo dispositivo capaz de iniciar una reacción física o química. El
papel de los cebadores para la
duplicación de las cadenas de ADN dentro de la célula es fundamental. Si
uno quiere hacer una copia para que se inicie la síntesis de una nueva cadena
utilizando como molde una cadena simple es necesario que el extremo de la
cadena sea doble. Esto se logra pegando un primer
en el extremo
Los cebadores se
preparan sintetizándolos en forma apropiada de modo que se complementen con la
zona del ADN que deseamos copiar. Si en lugar de una hebra de ADN se usa como
molde una de ARN necesitamos la presencia de la transcriptasa
reversa.
1.
2.
3.
![]()
cebador 1
cebador 2
![]()
4. ÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄ
ÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄ
ÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄ
ÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄÄ
Ä = A Ä = T Å = C Ä = G
Figura 3. Síntesis de nuevas cadenas de ADN utilizando
cebadores. En el paso 1 tenemos las dos cadenas unidas formando un dúplex. Por
calentamiento podemos separarlas, es lo que muestra el paso 2. Recordemos la
orientación de las cadenas
Resumen
breve de lo aprendido hasta ahora en este capítulo y en el anterior.
·
Los principios de complementariedad entre las bases que
forman el ADN para asegurar la síntesis de cadenas idénticas.
·
La importancia de las uniones hidrógeno que permiten dicha
complementariedad.
·
La existencia de las enzimas de restricción que permiten cortar
al ADN en numerosos fragmentos dejando la posibilidad de insertar en la cadena
cortada, fragmentos de ADN de otros organismos.
·
La importancia de los plásmidos
en el clonado molecular.
·
La hibridización molecular entre
cadenas ADN/ADN ó ADN/ARN.
·
La transcripción reversa que
permite la síntesis de ADN a partir de ARN.
·
La necesidad de un cebador o primer para iniciar la
síntesis de cadenas nuevas.
·
Saber que la replicación del ADN es semiconservativa.
Hasta este punto todo lo que aprendimos tuvo que ver con los aspectos
teóricos del campo molecular del ADN sin entrar en detalles de cómo esos
procesos ocurren dentro de una célula y cómo se llevan a cabo en el
laboratorio. Presentaremos a continuación los fundamentos de algunas técnicas
en uso, necesarias para realizar experimentos con ADN en el laboratorio.
Electroforesis
en gel: técnica para separar fragmentos de ADN.
La electroforesis en gel es una técnica ya conocida desde hace mucho tiempo que
se usa para separar moléculas en base a sus características físicas tales como
tamaño, forma y punto isoeléctrico1. Es útil para separar fragmentos
de ADN de distinto tamaño y aplicar en forma analítica (para el análisis
directo) o preparativa (conseguir una cierta cantidad), es el caso cuando se
purifican moléculas antes de aplicar otros métodos. Específicamente, la
electroforesis aplicada al ADN se usa para
purificar moléculas que serán usadas, entre otros objetivos en la
reacción de PCR (reacción de polimerasa en cadena),
clonado molecular, secuenciación y técnicas de inmunotransferencia.2 La electroforesis en gel
ha tenido su mayor aplicación para la separación de proteínas.
Para realizar una electroforesis se debe
elegir un soporte (papel, celulosa, poliacrilamida, agar) sobre el que se siembra la muestra y sumergir el
soporte en una solución dentro de una
bandeja. Cuando se separan proteínas o ácidos nucleicos cortos (oligonucleótidos) se usa acrilamida,
según la concentración de acrilamida se forman geles de distinta porosidad. Para asegurar el entramado del
gel se agrega una sustancia que favorece el
entrecruzado formando así el gel conocido como poliacrilamida que se diseña de acuerdo al tipo de
moléculas que se desee separar. Recordemos que la poliacrilamida
es un neurotóxico y debe ser manipulada con cuidado.
Para el caso del ADN el soporte ideal es un gel de agarosa (no tóxica), en especial para moléculas de ADN de
gran tamaño. Esta sustancia proviene de un alga marina. La agarosa
se vende en polvo y se disuelve en agua destilada por calentamiento, al dejar
enfriar la solución en un molde apropiado de forma rectangular o circular (caja
de Petri) forma una capa de un material parecido a la
gelatina pero más consistente que tiene una matriz conteniendo poros o canales.
La electroforesis permite separar las moléculas por tamaño sometiéndolas a la
acción de un campo eléctrico. Las moléculas se moverán dentro de la matriz del
soporte, a través de los canales del agar. hacia uno de los polos y la velocidad de migración dependerá
de su tamaño, cuanto más pequeñas sean se moverán más rápidamente. La figura 4
muestra un aparato de electroforesis horizontal con sus conexiones, mientras
que la figura 5 muestra un aparato vertical.

Figura 4. Aparato de electroforesis horizontal, los cables
se conectan a una fuente de poder. En las canaletas se coloca la solución
buffer y en el centro del aparato se
apoya la muestra sembrada sobre una capa de agar
sobre un soporte de vidrio. Mediante un artefacto llamado peine se crean una
serie de calles en el agar por las que correrán las
muestras de ADN o de proteínas.

Figura 5. Aparato de electroforesis vertical.
Cuando se
aplica la corriente eléctrica al gel, la fuerza
electromotriz mueve a las moléculas a través de la matriz del gel hacia el ánodo (electrodo positivo) si las moléculas
están cargadas negativamente o hacia el cátodo (electrodo negativo) si están
cargadas positivamente. Para el caso de los ácidos nucleicos, éstos tienen cargas negativas debido a su esqueleto azúcar-fosfato (ver
capítulo 13).
Las proteínas pueden tener formas
complejas y cargas diferentes en consecuencia
puede ser que proteínas del mismo tamaño no migren en el gel a velocidades similares. Por esa razón las proteínas se
desnaturalizan en presencia del detergente dodecil-
sulfato de sodio más conocido como SDS. Este detergente recubre a las proteínas
con una carga neta negativa. Además, la cantidad que se une es proporcional al
tamaño de la proteína de modo que este tratamiento les confiere una carga
negativa y además todas las proteínas tienen una carga similar relacionada a su
masa. Las proteínas desnaturalizadas toman la forma de bastones largos y en
consecuencia la velocidad de migración en el gel no
depende de la carga y forma sino de su tamaño. En la figura 5 se muestra una
corrida electroforética de proteínas.

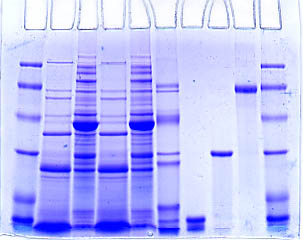
A 1 2
3 4 5
6 7 8 B
Figura
6. Gel de proteínas. Corrida de
electroforesis de mezclas de proteínas reveladas con coomassie
blue. Las flechas (A y B) indican las calles donde se sembró la mezcla
de proteínas de referencia. Los números indican las calles de siembra. Si
observamos con detenimiento, sin tener referencias exactas de este experimento,
vemos que en las calles 1 y 3 se ha sembrado la misma muestra. Lo mismo ocurre si comparamos las calles 2 y 4. En las calles
7 y 8 hay una sola proteína pero no es la misma ya que su posición relativa en
el gel es muy distinta. La flecha roja muestra la
serie de muescas que se hacen utilizando la ayuda de un peine especial, estas
muescas delimitan las calles en donde se siembran las muestras. Las cabezas de flechas verde y negras muestran las bandas correspondientes
a las proteínas cuyo peso molecular se conoce. En los bordes de los geles las corridas suelen deformarse, observar el extremo
izquierdo y compararlo con el derecho.
Tal como
describimos antes las calles extremas del gel de la
figura 5 contienen una mezcla de proteínas de distinto peso molecular (escalera)
y las calles del centro permitieron analizar distintas mezclas de proteínas
conteniendo varios constituyentes. El peso molecular de cada banda se puede
calcular por referencia a los pesos moleculares patrones.
La figura
7 muestra un gel para ADN.
![]()

Figura 7. La flecha parte desde el lugar de siembra,
la presencia de un colorante permite visualizar el lugar de la siembra. A su
vez, a medida que transcurre la electroforesis el colorante permite observar el
frente de la corrida y de ese modo permite elegir el momento de corte, antes de
que salgan del gel. Cada segmento coloreado
corresponde a una calle donde se puede correr una muestra en particular
Cuando se desea teñir ADN se puede
recurrir al bromuro de etidio, a un colorante
fluorescente o la muestra puede contener un nucleótido radiactivo. Así es
posible fotografiar la corrida iluminada con rayos ultravioleta. Si la muestra
contiene un nucleótido radiactivo, se revela por autorradiografía.
Métodos y tinciones hay muchos lo importante aquí es que los lectores adquieran
el concepto de cómo se pueden separar las moléculas según su tamaño. Las
moléculas de ADN doble son casi bastones por lo que al igual que las proteínas
su velocidad depende de su tamaño. En la figura 8 se ve un ejemplo de corrida
de ADN, es la misma muestra de ADN cortada con distintas enzimas de restricción
(ver capítulo 13). Cada enzima da lugar a un patrón de corte diferente de
acuerdo con la secuencia del ADN cortada.
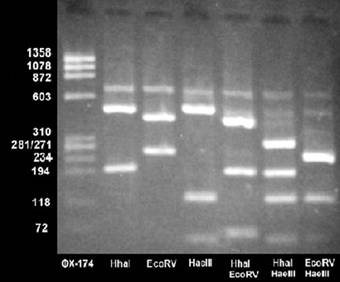
Figura 8. Patrón de restricción de una muestra de
ADN, en la calle de la izquierda se corrió la muestra patrón que permite
determinar los pesos moleculares. En las calles siguientes la misma muestra de
ADN fue sometida a la acción de diferentes enzimas de restricción cuyos nombre figuran en la base de la corrida.
Momento de recapitular
Ya aprendimos cómo puede separarse una
muestra de ADN sólo falta que la saquemos del gel,
para eso cortaremos la banda del agar y la
obtendremos en forma pura, pero antes de detenernos en cómo hacerlo vamos a ver
las aplicaciones directas. Al tener separado un trozo de ADN correspondiente a
un gen viral podríamos, por ejemplo, producir un biofármaco.
Ejemplo de aplicación de las técnicas moleculares:
Obtención de la vacuna contra el virus de la hepatitis B.
Los virus
además de ser extremadamente pequeños (sólo pueden ser vistos con el
microscopio electrónico) están compuestos por una
genoma rodeado de proteínas que protegen a este ácido nucleico y además le
permiten saltar de célula en célula para poder multiplicar ya que son parásitos
obligados. Es decir que sólo pueden reproducirse dentro de una célula ya que
ésta contiene todos los elementos
necesarios para sintetizar ácidos nucleicos.
La figura 9 muestra un corte esquemático de la
partícula del virus de la hepatitis B.
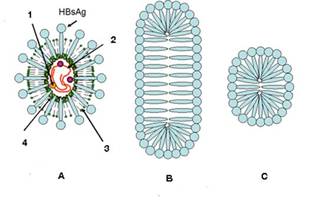
Figura 9. El diagrama A muestra a la partícula de
virus completa conocida con el nombre de partícula de Dane,
cuyo tamaño es de 40 nm. 1. Enzima polimerasa, 2. ADN, 3.core
(núcleo interno) 4. Membrana. Se destaca en la membrana el antígeno HBsAg denominado así por ser el antígeno o proteína viral
de superficie. En la sangre de las personas infectadas circulan también dos
tipos de partículas más que no son infecciosas porque carecen de ADN. La
partícula B es filamentosa con una longitud de 200nm y
Cuando un virus ingresa a un organismo,
el organismo mediante el auxilio del sistema inmune sensa
que hay proteínas extrañas circulando por ahí. Entonces como estudiamos en el
capítulo 8 de este curso, primero aparecerá una respuesta natural innata y
luego de un tiempo una respuesta inmune específica.
¿Qué es lo
que ven las células del sistema inmune que las alertan? Ven, reconocen o sensan antígenos (proteínas externas del virus) que para el
caso del virus de la hepatitis B vemos que se trata del antígeno de superficie HBsAg como se muestra en la figura
Veamos
entonces cómo se procedió a obtener el gen del HBsAg,
para ello nos vamos a detener en la figura 10 que muestra una representación
del genoma del virus de hepatitis B.
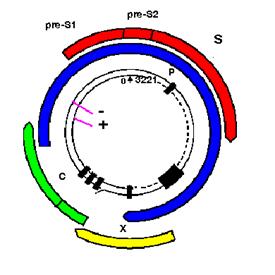
Figura 10. Genoma del virus de la hepatitis B http://wwwmicro.msb.le.ac.uk/3035/HBV.html.
Los dos círculos negros indican que se
trata de un ADN circular doble cadena aunque una porción dibujada con rayitas
es de cadena simple. Por convención, a una de las cadenas se la denomina menos
(-) y la otra es más (+). Se muestra el origen de la replicación en el 0 y al
lado el número 3221 que se refiere al número total de nucleótidos que tiene el
genoma viral. Las flechas de colores muestran el tamaño de los genes y el
sentido de su lectura. En azul se señala el gen de la polimerasa
viral (P), en verde la proteína de la cápside C
(cubierta viral), en amarillo el gen X que no tiene una función definida y en rojo el gen S
correspondiente al antígeno de superficie. Nótese que hay dos regiones pre-S1 y pre-S2 que no son
necesarias para la expresión del gen S en un vector.
Es interesante destacar que los genes
poseen nucleótidos compartidos ( se superponen) es lo que se conoce con el
nombre de genes solapados, si miramos la figura vemos, por ejemplo, que el gen
S tiene una región común con el gen P, pero al empezar en otro nucleótido y
terminar previamente resulta un polipéptido diferente. Esta situación refleja
la exquisita regulación de la información creada por los virus para subsistir,
así un genoma tan pequeño como el del virus de la hepatitis B codifica por
cinco proteínas.
Volviendo a los pasos de la obtención de la vacuna contando
ya con el conocimiento de las técnicas.
1.
Localización del gen S en el genoma viral
2.
Corte con enzima de restricción.
3.
Separación del ADN en un gel de agarosa.
4.
Obtención del ADN.
5.
Recombinación con un plásmido
bacteriano (ver figura 12 del capítulo
13)
6.
Producción de bacterias conteniendo los plásmidos
7.
Recombinación del plásmido
bacteriano con uno de levadura para su expresión.
8.
Crecimiento de la levadura recombinante
en tanques de fermentación.
9.
Purificación de la proteína HBsAg
a partir del líquido del cultivo.
10.
Formulación para la vacuna. Envasamiento
y distribución.
11.
Aplicación inyectable. Inmunización-vacunación específica.
Conclusiones
He
tratado de explicar en la forma más sencilla posible las técnicas moleculares
desarrolladas a partir del conocimiento de la estructura del ADN y de las
enzimas de restricción así como el principio de otras técnicas de uso común en
los laboratorios. Los que deseen aprender en profundidad pueden consultar
libros de texto o la información que se brinda en Internet ,
ésta última de gran valor suele tener, sin embargo, algunos errores. Creo
conveniente reservar para un próximo capítulo la explicación de
otra técnicas de aplicación general como la inmunotransferencia
y
1. Punto isoeléctrico:
la carga de una molécula de proteína variará según el pH
de la solución en que se encuentre. El punto isoeléctrico
de una proteína es el pH al cual la carga neta de la
molécula es cero.
2. Inmunotransferencia esta técnica se explicará en el capítulo 15
*
Este curso es una contribución de Química Viva educativa (e-Lab) a la
propagación del conocimiento científico entre los estudiantes de la escuela
secundaria. Departamento de Química Biológica. Facultad de Ciencias Exactas y
Naturales. Universidad de Buenos Aires.
Volver al menú de selección de capítulo.
|
|
Revista QuímicaViva Revista Electrónica del Depto. de Química Biológica, Facultad de Ciencias Exactas y Naturales, Universidad de Buenos Aires, Argentina. quimicaviva@qb.fcen.uba.ar |